简介
转录因子 (Transcription Factors,TFs) 是一类通过结合特定的DNA序列,从而调控基因表达的蛋白质。它们在细胞分化、发育、代谢、应激响应以及疾病(如癌症)中发挥关键作用。了解转录因子的结合位点对于揭示基因调控机制、疾病研究和药物开发具有重要意义。
先前我已经做了一个转录因子靶基因预测APP/R包(TF Target finding/TFTF),广受好评。但是有同学提出,预测出结果之后如何进行后续实验验证,如何获得结合位点,今天以JASPAR数据库Scan功能做简要介绍。
JASPAR数据库(http://jaspar.genereg.net/)是一个广泛使用的转录因子结合位点(Transcription Factor Binding Sites,TFBS)的免费开源数据库,包含了多种物种的转录因子结合位点信息,广泛应用于基因调控研究和功能预测。
步骤
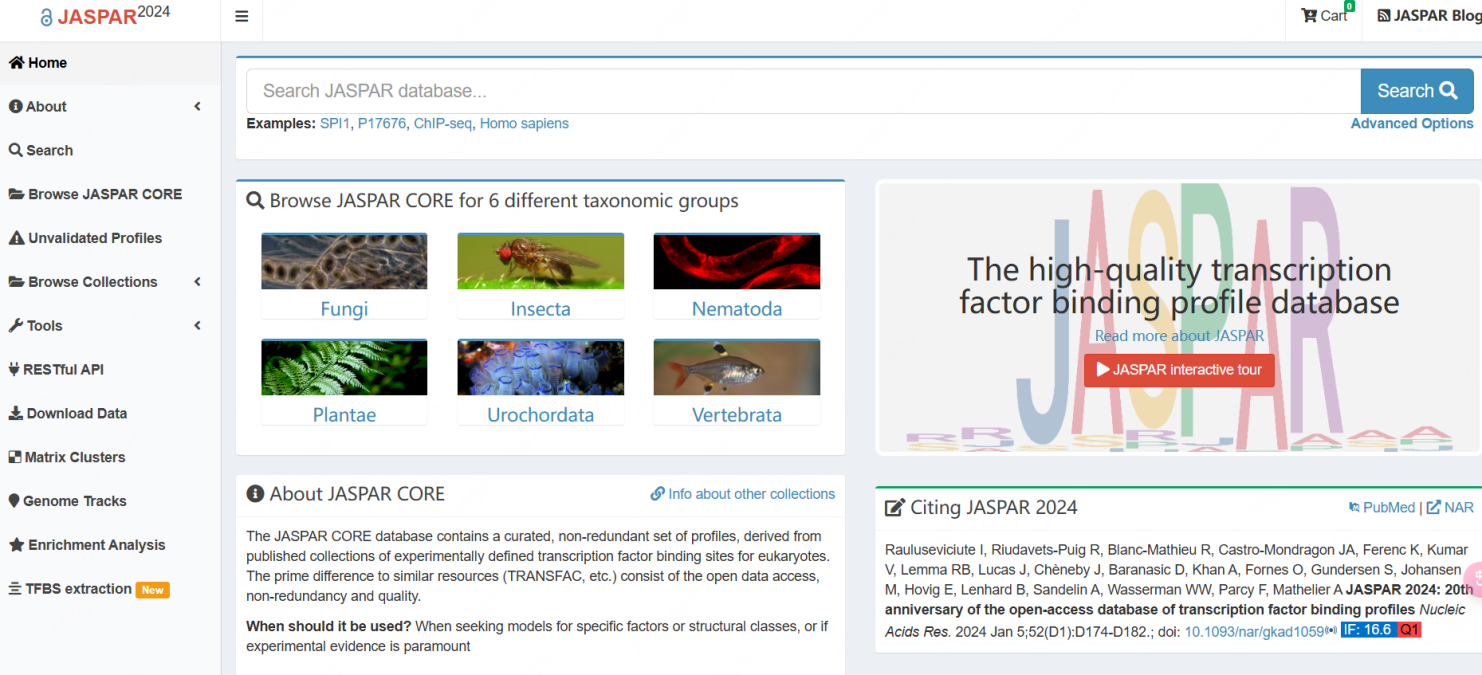
利用JASPAR进行转录因子结合位点预测的步骤如下(以人源FOXO3为例):
A.在JASPAR网站上检索FOXO3,勾选对应物种的转录因子条目。
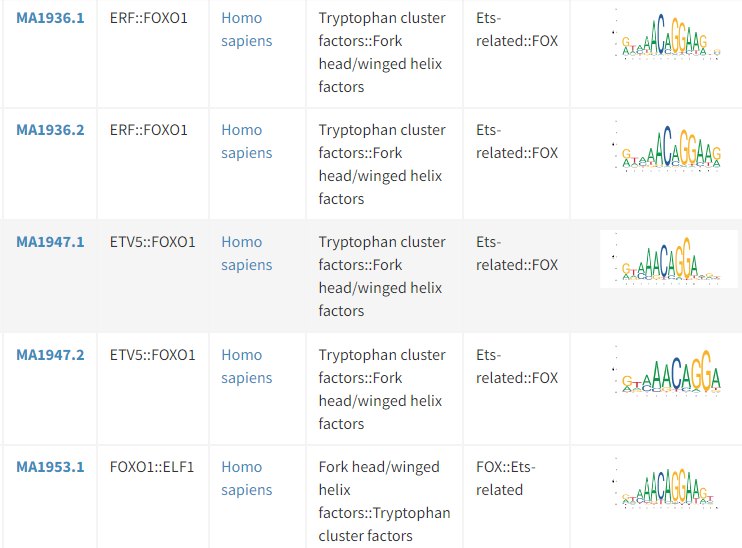
PS:一个转录因子可能有多个条目,比如下图中除了人中的特异motif,还有人鼠公共motif,可以都选,具体根据自己的目的选择。另外,有些转录因子(如FOXO1)可能结合不同的伴侣蛋白发挥转录调控作用,可能具有不同的motif,这需要查阅文献资料进行选择,或者都纳入,看哪种结合位点最多最可靠。
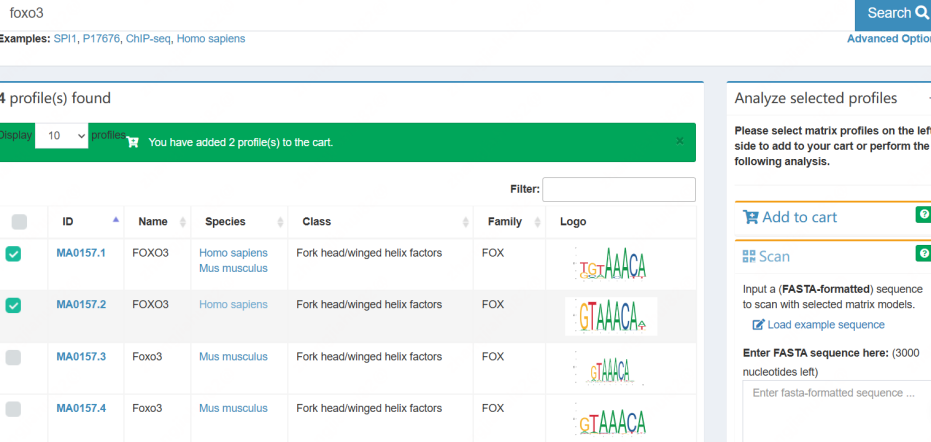
B.勾选对应条目后进行,展开右边scan,可以看到一个输入框,其中需要输入启动子序列。
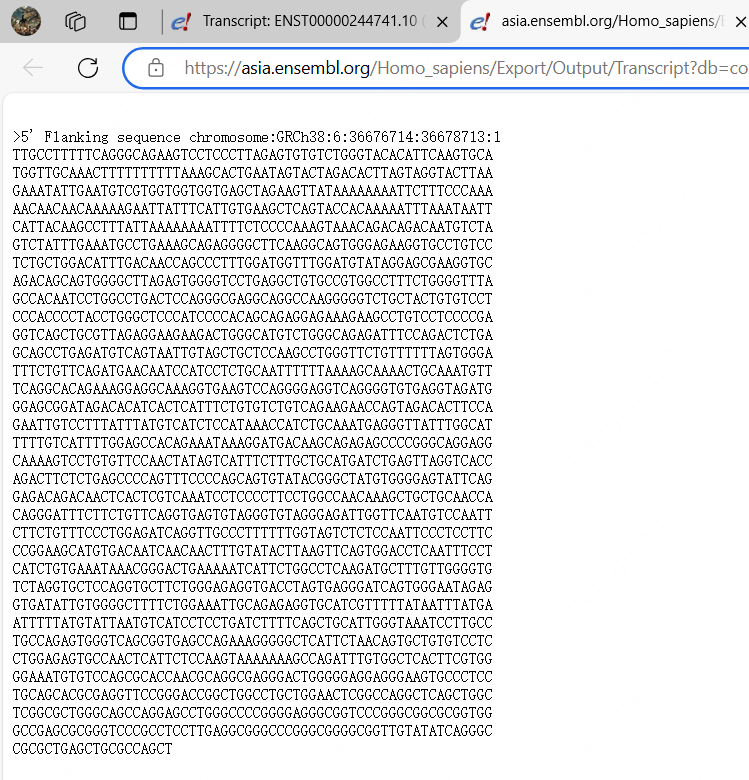
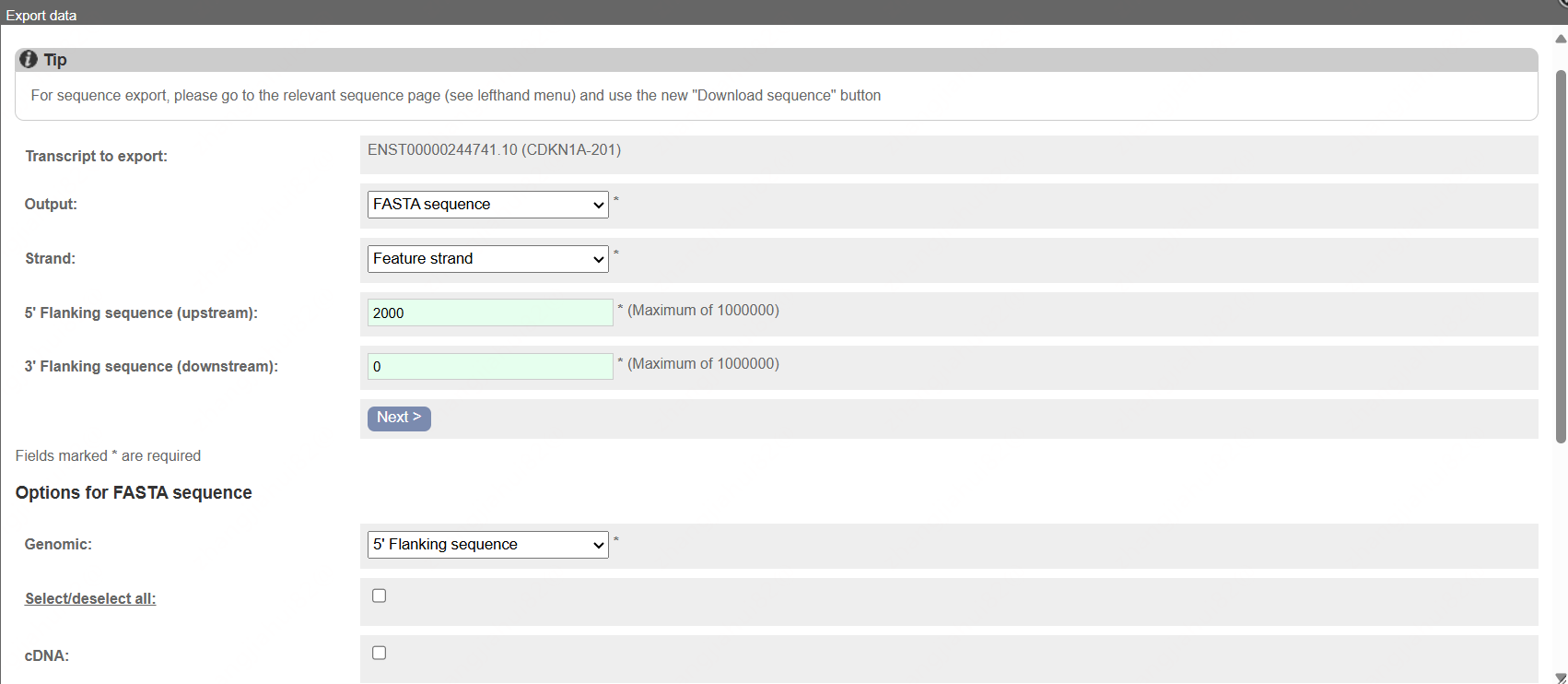
C.检索基因:打开Ensemble数据库(https://www.ensembl.org/index.html),搜索所需基因(以CDKN1A为例),点击show transcripts,一般选择第一个蛋白编码转录本,具体根据自己的研究目的。

D.获取启动子序列:点击左侧Export,弹出窗口中5’Flanking sequence输入2000,及获取转录起始位点上游2000bp序列(TSS2000),具体多长可以根据需要选择1000-3000bp序列。Option for FASTA sequence:Genome选择5’flanking sequence,其它无需勾选。之后next选择text打开即可获得TSS2000序列。
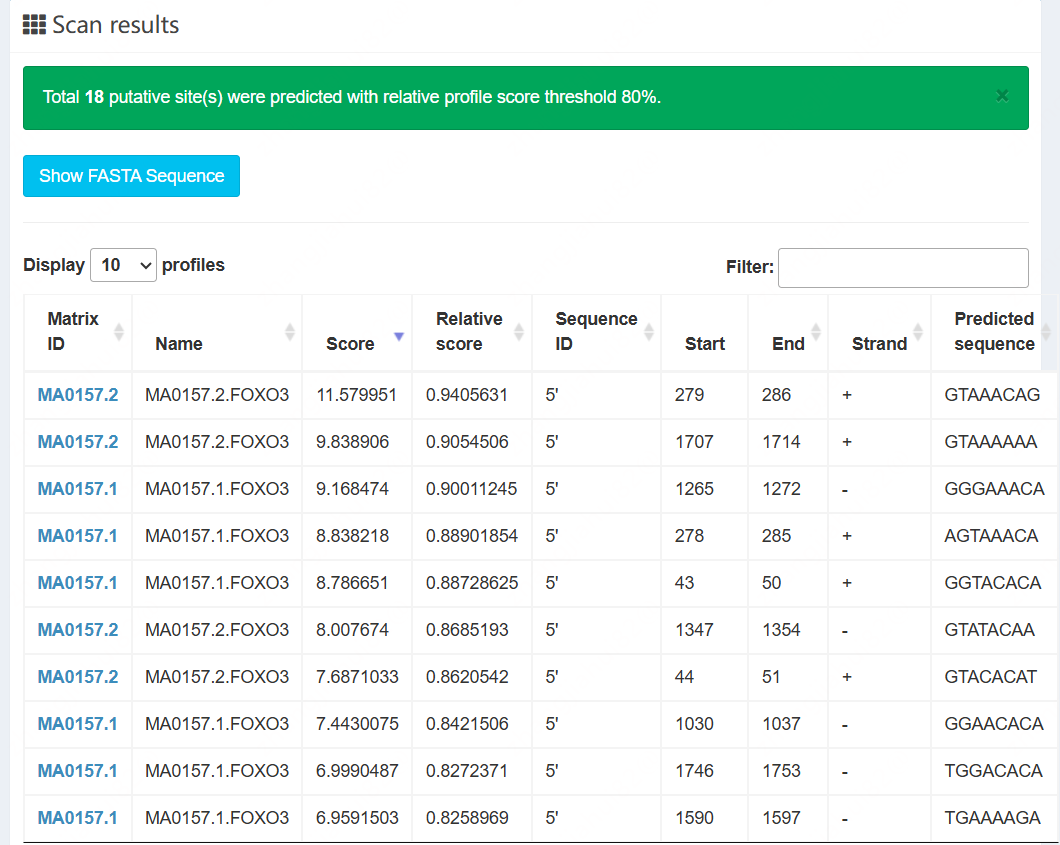
E.将序列(包括第一行>5’flanking sequence……)全部复制到前面JASPAR网站上Scan框中进行预测,Threshold根据预测结果可以适当调整,多的话调高一点。

F.扫描结果得分Score由高至低排序,可以选择最高的几个。
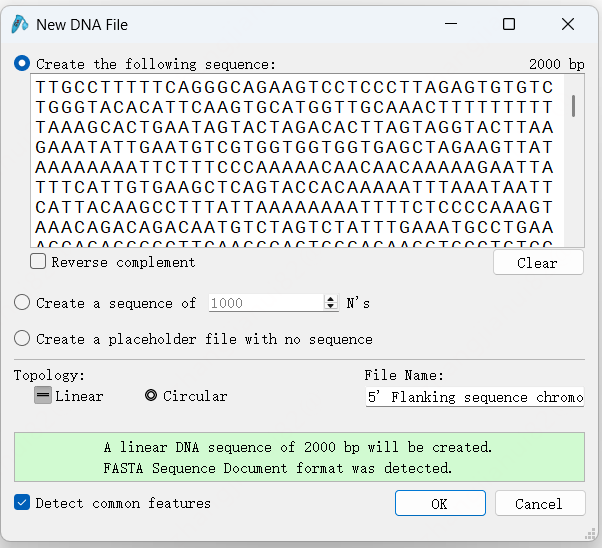
G.可视化位点定位:在snapgene软件中输入TSS2000序列,点击ok。复制JASPAR网站上得分Score最高者对应的预测序列到snapgene软件下方的find DNA sequence中,点击next即可出现预测序列位置。
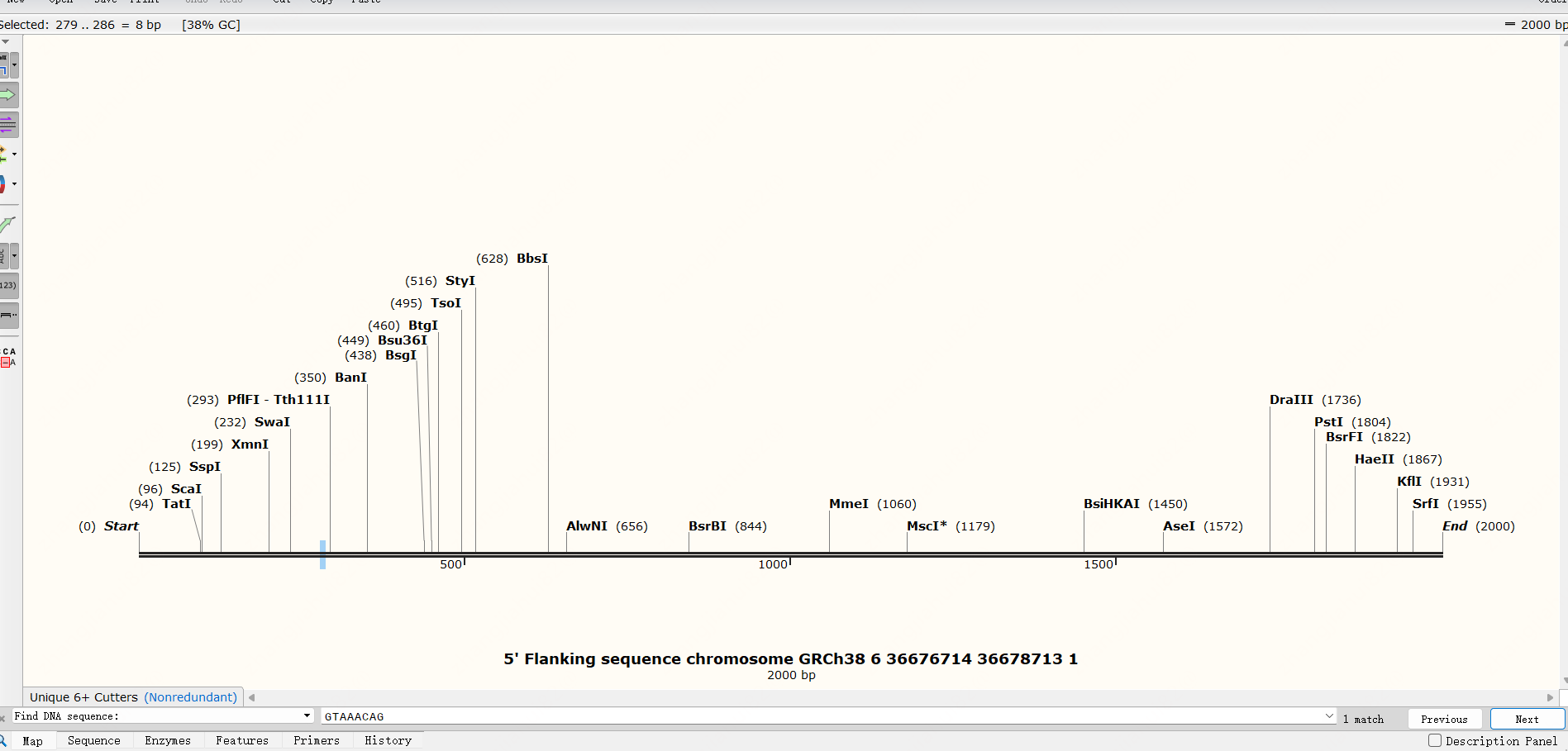
H.点击snapgene软件下方的sequence,选取复制包含预测序列的前后300-500左右侧翼序列,粘贴至primer premier进行引物设计。
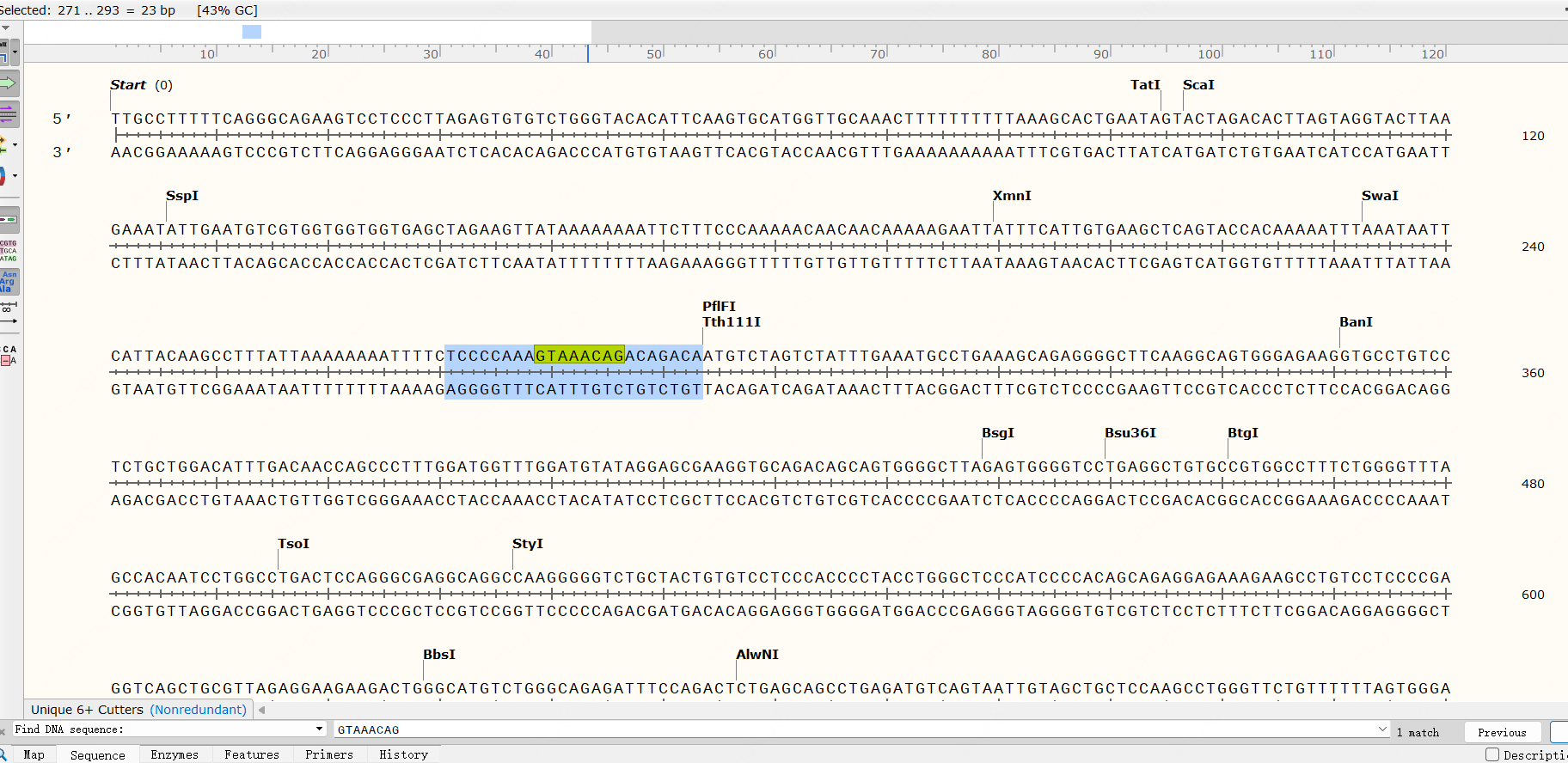
I.引物设计参考一般流程,此处不做介绍,需要注意的是设置上下游引物范围,保证上游引物和下游引物P出来的产物包含结合位点,并且最好位点在产物中居中一点。设计的引物需要根据荧光素酶报告基因载体MCS中酶切位点插入相应的限制酶识别序列及保护碱基。
我网站上有软件及相关介绍:Primer Premier 5.0 安装使用 – 王进的个人网站https://www.jingege.wang/2020/05/29/primer-premier-5-0-%e5%ae%89%e8%a3%85%e4%bd%bf%e7%94%a8-2/