
NCBI
NCBI (National Center for Biotechnology Information)是指美国国立生物技术信息中心,NCBI研究小组包括计算机科学家,分子生物学家,数学家,生物化学家,实验物理学家,和结构生物学家,集中于计算分子生物学的基本的和应用的研究。这些问题包括基因的组织,序列的分析,和结构的预测。目前研究计划的一些代表是:检测和分析基因组织,重复序列形式,蛋白domain和结构单元,建立人类基因组的基因图谱,HIV感染的动力学数学模型,数据库搜索中的序列错误影响的分析,开发新的数据库搜索和多重序列对齐算法,建立非冗余序列数据库,序列相似性的统计显著性评估的数学模型和文本检索的矢量模型。另外,NCBI研究者还坚持推动与NIH内部其他研究所及许多科学院和政府的研究实验室的合作。
了解更多:https://www.ncbi.nlm.nih.gov/
miRBase 21.0
miRBase序列数据库是一个提供包括 miRNA 序列数据、注释、预测基因靶标等信息的全方位数据库,是 miRNA 研究权威数据依据之一。microRNA 21.0版是最新版本,新版数据库的可靠性进一步提升,总计,新增了4196条发夹前体序列和5441条成熟miRNA,并清除了一些不明确的和错误注释的序列。
了解更多:http://www.mirbase.org/search.shtml

EXORBASE
EXORBASE是由人血液外体的RNA SEQ数据分析衍生的环状RNA(CyCRNA)、长非编码RNA(LNCRNA)和信使RNA(mRNA)的储存库,还包括来自已发表文献的实验验证。EXORBASE基于正常人和不同疾病患者的标准化RNA SEQ数据,整合和可视化RNA表达谱。EXOBASE的目的是收集和表征人类血液外体中的所有长RNA物种。提供了注释、表达水平和可能的原始组织。EXORBASE将帮助研究人员识别血液外体中的分子标记,并将触发新的循环生物标志物发现和人类疾病的功能含义。

piRNABank
piRNA数据库是由印度科学家SaiLakshmiS及其合作者建立,以保存新发现的piRNA序列。piRNABank收录了最全面piRNA序列信息,其收录了包括人、小鼠和大鼠的近2000万个piRNA相关序列,经过同源序列去除、基因组定位等筛选,得到10万多个在基因组中具有唯一靶位点的piRNA序列。数据库支持物种和染色体方式的多种的搜索方式。piRNABank也可以在基因组大图谱上显示每个piRNA或piRNA簇。
了解更多:http://pirnabank.ibab.ac.in/
DIANA-LncBase
提供lncRNAs上的miRNA靶的综合注释, DIANA-LncBase保存了全转录组实验验证的和计算预测的人类和小鼠lncRNAs上的miRNA识别元件。 DIANA-LncBase中有效的实验支持条目超过了5000对相互作用,计算预测的相互作用超过1000万对。DIANA-LncBase保存了每个miRNA-lncRNA对的详细信息,例如外部链接,转录本基因组位置的图形绘制,结合位点的表征,lncRNA组织表达以及MREs的保守性得分和预测得分。

Starbase
Starbase是做lncRNA/circRNA/microRNA等研究常用的强大数据库,Starbase以多种测序数据为支持,整合多个预测软件,能够帮我们解决以下这些问题:①根据microRNA找非编码RNA(比如lncRNA,circRNA等);②根据microRNA找mRNA靶点;③查找ceRNA调控分子;④查找RNA的结合蛋白。
了解更多:http://starbase.sysu.edu.cn/

circRNADisease
circRNADisease是一个由circRNA和疾病实验人工支持的数据库,是了解人类疾病中circRNA作用的重要知识来源。circRNADisease中的每一个条目包括关于circRNA和疾病名称、circRNA表达模式、实验技术、circRNA合作伙伴、关于circRNA的简短功能描述、文献参考和其他注释。
了解更多:http://cgga.org.cn:9091/circRNADisease/
CSCD癌症相关的circRNA
CSCD(癌症特异性CyrRNA数据库)是为癌症特异性环蛋白开发的数据库。CSCD收集了来自87个癌细胞样品的可用RNA测序(总RNA与rRNA耗尽或波利亚富集)数据集。我们使用了4种流行的算法:CIRI、FIDEXCIRC、CURCRNAX FIDER和CURCExpRever进行了综合分析。然后我们收集仅在癌症样本中鉴定的CurcRNA,而不是任何正常样品。目前在CSCD有272152种癌症特异性环蛋白。
了解更多:http://gb.whu.edu.cn/CSCD/#
CycPediaV2
CycPediaV2是一个更新的综合数据库,包含来自六个不同物种的超过150个RNA SEQ数据集的circRNA注释,允许用户搜索、浏览和下载具有各种细胞类型/组织(包括疾病样本)的表达特征/特征的circRNA。此外,更新的数据库结合了人与小鼠之间的CrCRNs的保守性分析。最后,Web界面还包含用于比较样本之间的CyrRNA表达的计算工具。
了解更多:http://www.picb.ac.cn/rnomics/circpedia/
GO富集性分析
在获得特定的基因集之后,GO功能显著性富集分析能够确定差异基因行使的主要生物学功能。GO主要有三个ontology(ontology:Biological Process、Cellular Component、Molecular Function),分别描述基因的分子功能(Molecular Function)、细胞组分(Cellular Component)和生物过程(Biological Process)。GO的基本单位是term,每一个term都对应一个属性。
了解更多:http://www.geneontology.org/
KEGG富集性分析
KEGG 显著性富集分析能确定差异表达基因参与的最主要的生化代谢途径和信号转导途径。Kyoto Encyclopedia of Genes and Genomes(KEGG)是有关Pathway的主要公共数据库。在生物体内,不同基因相互协调行使其生物学功能,Pathway的分析有助于更进一步了解基因的生物学功能。
了解更多:https://www.kegg.jp/
TANRIC数据库
TANRIC数据库: 是lncRNA研究的利器,是癌症非编码RNA的地图集,包括20种癌症,超过8000个样品。TANRIC是一个交互式的数据分析和可视化平台,含有三大类的数据,包括lncRNA注释信息,RNA-Seq数据以及profiling数据。
了解更多:http://bioinformatics.mdanderson.org/main/TANRIC:Overview
FRNADB
FRNADB是一种新颖的数据库服务,它承载大量的非编码转录本,包括来自H-IV数据库的注释/非注释序列,非编码和RNADB。对包含的序列进行了一组计算分析。这些分析包括RNA二级结构基序发现、EST支持评价、顺式调控元件搜索、蛋白质同源性搜索等。
了解更多:http://togodb.biosciencedbc.jp/togodb/view/frnadb_summary#en
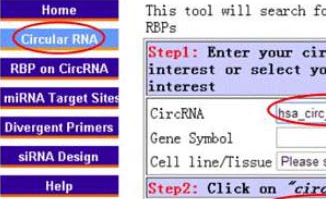
预测circRNA结合靶点—CircInteractome
数据库CircInteractome预测了已知的109个RNA结合蛋白数据集与circbase中的circRNA的结合位点,并利用Targetscan软件预测了miRNAs与circRNA的潜在结合位点。可进行circRNA分子检索、circRNA结合蛋白预测、PCR引物设计、siRNA干扰序列设计等操作。
了解更多:https://circinteractome.nia.nih.gov/
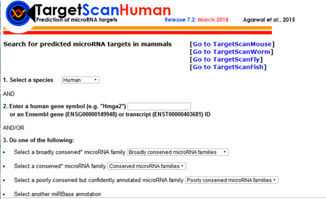
miRNA靶基因预测
Findtar3: http://bio.sz.tsinghua.edu.cn/
TargetScan: http://www.targetscan.org/
miRDB: http://mirdb.org/miRDB/index.html
RNA22v2: https://cm.jefferson.edu/rna22v2/
microRNA.org: http://www.microrna.org/microrna/home.do
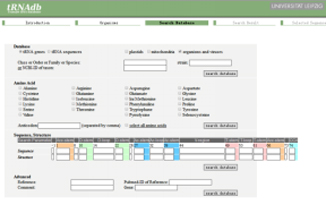
tRNA数据库
这里汇总了几个常用的tRNA数据库:
PlantRN:http://plantrna.ibmp.cnrs.fr/
tRFdabase:http://genome.bioch.virginia.edu/trfdb/
tRNAdb:http://trna.bioinf.uni-leipzig.de/DataOutput/
tRNADB-CE:http://trna.ie.niigata-u.ac.jp/cgi-bin/trnadb/index.cgi
.tRNA命名: http://www.genenames.org/cgi-bin/genefamilies/set/478
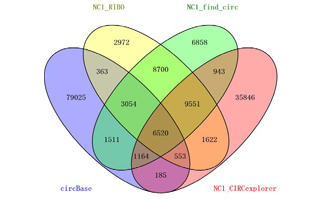
韦恩图
用于显示元素集合重叠区域的图示,能够展示不同数据之间的交集、差集,最多可以输入30个列表。
BioVenn: http://www.cmbi.ru.nl/cdd/biovenn/index.php
Venn: http://bioinformatics.psb.ugent.be/webtools/Venn/
VENNY: http://bioinfogp.cnb.csic.es/tools/venny/index.html
Venn Diagram Plotter: http://omics.pnl.gov/software/venn-diagram-plotter
VennDiagram: R package http://cran.r-project.org/web/packages/VennDiagram/index.html
Ensembl
ActhBl是脊椎动物基因组的基因组浏览器,它支持比较基因组学、进化、序列变异和转录调控的研究。Actubl注释基因,计算多重比对,预测调节功能并收集疾病数据。集成工具包括BLAST、BLAT、BIOART和所有受支持物种的变异效应预测器(VEP)。
了解更多:
http://www.ensembl.org/index.html
piRNApredictor
用于预测piRNA序列信息,该软件不依赖基因组数据来鉴定非模式生物piRNA,在线软件给出的piRNA高精度预测结果,对表观遗传学、调控网络与piRNA功能的进一步研究有重要理论意义和应用价值。目前,在线软件piRNApredictor已被国外科研机构用于猪的piRNA研究中。
了解更多:
https://omictools.com/pirnapredictor-tool
RNAfold web server
用于预测RNA分子二级结构,输入我们需要研究的RNA序列,系统通过计算,运行结果将会显示不同的结构表示,我们可以根据自己的需要进行选择。
了解更多:
http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi

NRED
提供人和小鼠的长链非编码RNA在芯片数据的表达信息。这也是John mattick实验室构建的网站。
了解更多:
http://jsm-research.imb.uq.edu.au/nred/

LncRNADisease database
长非编码RNA疾病数据库(lncRNA and disease database, LncRNADisease),这一数据库收录了160多种和长非编码RNA有关的疾病,并集成了一个生物信息学工具用以预测新的人类长非编码RNA和疾病的关系。
了解更多:
http://www.cuilab.cn/lncrnadisease
CyrRNADB数据库
CyrRNADB数据库是一个全面收录人类circRNA的数据库,它是免费提供的,该circRNA数据库的最新版本包含32914个注释的外显子环,并且可以是大规模研究circRNA的宝贵资源,特别是在人类中。
了解更多:
http://202.195.183.4:8000/circrnadb/circRNADb.php

UniProt蛋白序列与功能
UniProt为科学界提供一个全面、高质量、可自由获取的蛋白质序列和功能信息资源。
LNCipedia
对人类的长链非编码RNA的序列和结构全面的注释。
了解更多:
http://www.lncipedia.org/db
ExcOctA外泌体数据库
外泌体是由大多数细胞类型分泌的内吞细胞来源的30~150 nm的膜囊泡,ExcOctA是一个外泌体数据库,提供了在多个生物体中的外来体中识别的内容。

Vesiclepedia胞外囊泡数据库
Vesiclepedia是一个专门收录胞外囊泡研究的数据库,涉及到的物种有33个,来源研究538个,包含了凋亡体,微泡,外泌体等在内的囊泡中发现的miRNA/mRNA/蛋白 /脂类。
了解更多:
http://www.microvesicles.org/
EVpedia
EVpedia是一个收录了原核和真核生物胞外囊泡研究的数据库。该数据库包含不同囊泡研究(包括外泌体)中发现的miRNA/mRNA/m蛋白/脂类/代谢产物及其对应的marker。除此之外,该数据库还可以进行数据分析和功能注释。
了解更多:
http://student4.postech.ac.kr/evpedia2_xe/xe/
The Functional lncRNA Database
一个哺乳动物lncRNA数据库。要搜索一个特定的lncRNA,在搜索框中输入其名称下面,选择合适的品种。或者,你可以浏览所有lncRNAs一次。目前,该数据库包含来自人,小鼠和大鼠的lncRNAs。
了解更多:
http://www.valadkhanlab.org/database
lncRNAdb
提供有生物学功能的长链非编码RNA的全面注释。这是长链非编码RNA研究领域的大牛John mattick实验室构建的网站。
了解更多:
http://student4.postech.ac.kr/evpedia2_xe/xe/

NONCODE
提供对长链非编码RNA的全面注释,包括表达和该团队开发的ncFANs计算机软件预测的lncRNA功能,这是非编码RNA研究的知名数据库。
了解更多:
http://www.microvesicles.org/
GeneCards v3
GeneCards是一个可搜索的集成的人类基因数据库,提供了简洁的基因组相关的信息,对所有已知和预测的人类基因。
了解更多:
http://www.genecards.org/

NCBI/ BLAST
序列分析常用工具。BLAST发现生物序列之间的相似区域,该程序将核苷酸或蛋白质序列与序列数据库进行比较,并计算统计学意义。
了解更多:
https://blast.ncbi.nlm.nih.gov/Blast.cgi
Linc2GO数据库
该数据库旨在提供人类lncRNA的全面功能注释,整合了microRNA-mRNA以及microRNA-lncRNA相互作用数据以基于“ceRNA假说”产生lncRNA功能注释。
了解更多:
https://omictools.com/linc2go-tool
lncLocator: long non-coding RNA
由上海交大模式识别与生物信息学研究组开发,用于预测lncRNA在亚细胞中的定位。
了解更多:
http://www.csbio.sjtu.edu.cn/bioinf/lncLocator/

UCSC Genome Browser
UCSC Genome Browser网站包含有人类、小鼠和大鼠等多个物种的基因组草图,与ENCODE同步更新,并提供一系列在线分析工具,用户能够通过它可靠和迅速地浏览基因组的任何一部分,并可得到与该部分有关的基因组注释信息,可免费下载。
了解更多:
http://genome.ucsc.edu/
TCGA
TCGA是美国国家癌症研究所(NCI)和国家人类基因组研究所(NHGRI)合作数据库,已产生了33种癌症的关键基因组变化的全面、多维图谱。TCGA数据集已经公开,包括两个以上的基因组数据,并且该基因组信息有助于癌症研究团体改善癌症的预防、诊断和治疗。
了解更多:
https://cancergenome.nih.gov/
circNet
整合circRNA-miRNA-mRNA的互作网络。
了解更多:
http://circnet.mbc.nctu.edu.tw/
circRNA检索数据库
用于检索circRNA信息。
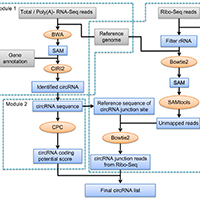
CircPro
CircPro是自动化高通量数据分析流程,它能够检测circRNAs,预测其蛋白质编码潜力,并发现Ribo测序数据的连接读取。
了解更多:
http://bis.zju.edu.cn/CircPro/
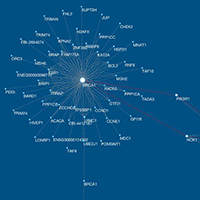
cystoscape网络图
Cytoscape是一个专注于开源网络可视化和分析的软件,它可以建构分子交互作用网络,以可视化的方法将分子间的互作关系展现出来。
了解更多:
http://www.cytoscape.org/index.html
catRAPID
大规模预测RNA与蛋白质相互作用的算法。
了解更多:
http://big.crg.cat/gene_function_and_evolution/services/catrapid
弦图(Chord Diagram)
弦图是为了表示一组具有相互关系的单位之间的联系,通常连线表示具有联系,连线的粗细表示权重。可以用于展示基因融合、共表达、蛋白互作等信息。
了解更多:
http://mkweb.bcgsc.ca/tableviewer/visualize/
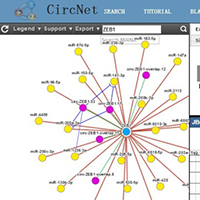
ceRNA功能研究数据库:circNet
circNet 是ceRNA功能研究数据库,circNet利用464个RNA-seq测序数据,进行新circRNA预测及基因组注释,并计算已知的及新预测的circRNA表达情况,构建circRNA-miRNA-gene调控网络。
了解更多:
http://circnet.mbc.nctu.edu.tw/
聚类图
聚类分析是一种在高通量数据中常用的无监督分类方法。通过特征集计算样本之间的相似性,构建相似性矩阵,然后根据一定的原则进行聚类,最终实现将相同或相似类型的样本聚类在一起,可以直观的观察样本之间的差异。
了解更多:
http://www2.heatmapper.ca/expression/
推荐微生信,0代码在线绘制110+款科研图!