 蛋白质互作网络是由不同蛋白通过彼此之间的相互作用构成,参与生物信号传递、基因表达调节、能量和物质代谢及细胞周期调控等生命过程的各个环节。在进行基因机制研究中,寻找互作蛋白是一个深入探究基因功能的过程,寻找未知蛋白充满艰辛,而通过数据库预测互作蛋白能够为我们的研究拓展思路,提供方向,让我们在科研道路上更加轻松。下面就为大家推荐四个可以用来预测相互作用蛋白的网站!
蛋白质互作网络是由不同蛋白通过彼此之间的相互作用构成,参与生物信号传递、基因表达调节、能量和物质代谢及细胞周期调控等生命过程的各个环节。在进行基因机制研究中,寻找互作蛋白是一个深入探究基因功能的过程,寻找未知蛋白充满艰辛,而通过数据库预测互作蛋白能够为我们的研究拓展思路,提供方向,让我们在科研道路上更加轻松。下面就为大家推荐四个可以用来预测相互作用蛋白的网站!
一:STRING网站
1.STRING数据库是比较经典的蛋白互作数据库。数据库使用简单。
2.输入基因名称后,可以获得蛋白互作网络图,其中圆圈节点之间的直线代表该直线连接的两个蛋白之间的相互作用关系,点击直线可查看蛋白的详细信息。
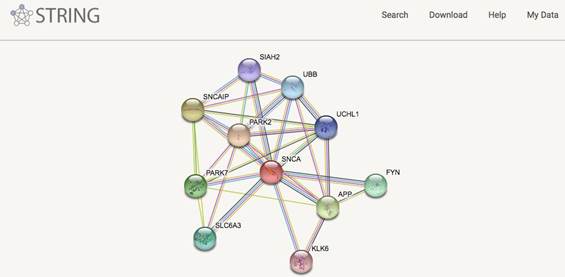
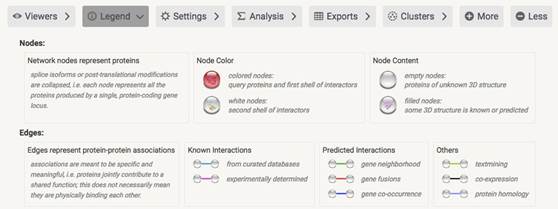
3.网站下方提示 不同颜色的直线代表不同的相互作用关系证据来源。包括蛋白质之间的直接物理相互作用,也包括蛋白质之间的间接功能相关性。它除了包含有实验数据、从PubMed摘要中挖掘的结果和综合其他数据库数据外,还有利用生物信息学的方法预测的结果。
二:Genemania网站。
1. Genemania也是比较常用的蛋白互作网站,网站结果类似cytoscape app,但是无需下载安装软件,直接在线使用,非常方便。

2.在网站左上方可以选择查询物种以及基因名称,同时点击下方不同的图标能够设置网络图展示形式:列表式、环状等。
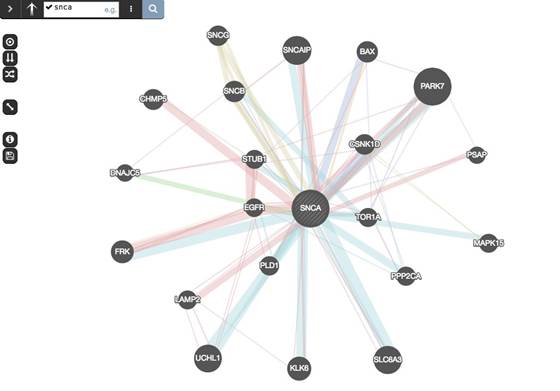
3.网站右侧可以设置网络边的来源,所应用的生物信息学方法有:物理互作、基因共表达、基因共定位、基因富集分析以及网站预测等等,通过这种方法能够设定检索范围。
三:unihi 网站
1. unihi是一个用于检索,分析和可视化人类分子相互作用网络的数据库。其主要目的是为广泛的生物学和医学研究人员提供一个全面且易于使用的网络调查平台。UniHI目前包括基因,蛋白质和药物之间近350,000个分子相互作用,以及许多其他类型的数据,如基因表达和功能注释。

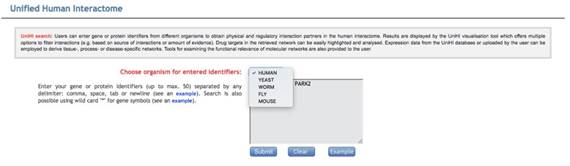
2.网站支持输入单基因或者多基因名称,寻找与之互作蛋白。

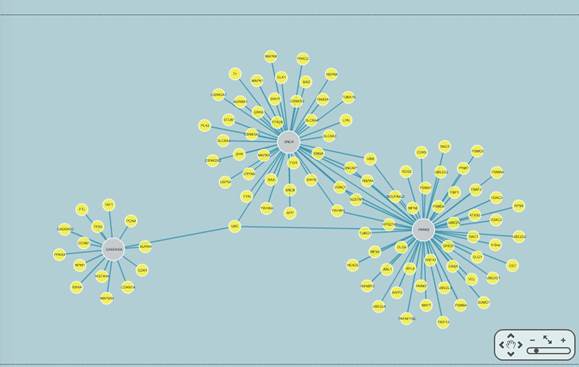
3.网站输出的蛋白互作网络图,以简洁直观的方式展现PPI网络图。

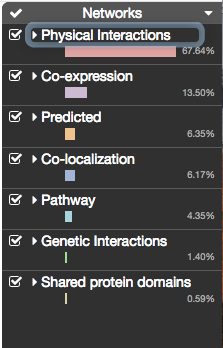
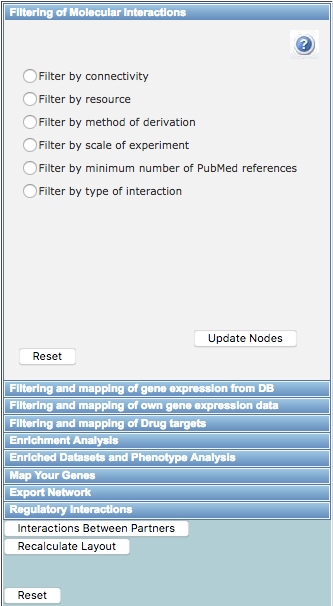
4.网站右侧栏可以通过分子作用、生物学途径、数据库预测、富集分析、药物靶标等进行限定。
四:Hitpredict网站
1. Hitpredict网站收集了IntAct, BIOGRID 和 HPRD数据库中的由高通量实验或者是小规模实验得到的蛋白质互作关系,综合了三大数据库的内容,数据准确全面。此外,还有根据互作得分估计的蛋白质相互作用。

2.目前为止,网站已经收集了人类,小鼠等9个物种的蛋白质-蛋白质相互作用关系,搜索栏选择物种(例如:人类),输入蛋白名称(例如:gene symbol:NASP; GENE ID: 4678)进行预测。
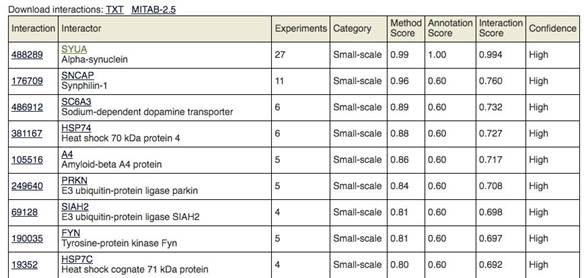
3.搜索结果显示该蛋白的基本信息,可能与之互作的蛋白以及这些基因的GO注释。

4.与前面几个网站不同的是网站预测结果会议列表形式展现,并给出互作评分以及关联性高低。根据分值(Likelihood)判断高低置信度。
5.当然,蛋白互作网络图仍是必不可少的,与Genemania网站类似,点击下方不同的图标 能够设置网络图展示形式:列表式、环状等。

您好,我想问一下,如何预测一个植物蛋白与几十个病原菌效应子之间有无互作关系?
您好,我想问一下,我研究的是一个新菌,测定了转录组现在想找到关键调控因子,我一开始看文献里有人通过差异基因使用string进行蛋白质或作,但是我输入进去发现说是库里没有这个物种,这种情况我还可以怎么解决呢
您好,请问用STRING、GENEMANIA、BIOGRID分别查找与某基因/蛋白后,发现在BIOGRID里面得到的分子与前两个里面的没有任何交集,这种情况是否正常呢?BIOGRID的结果是否可信呢?
请问下昆虫和病毒的蛋白质互作预测应该怎么做,上面这些网站好像都没有收集这些物种
看了进哥哥这篇文章大受启发!感谢!获得了200多个互作蛋白应该怎么筛选呢,想取交集,但是string数据库就得到了十几个,筛选的数量决定后面的工作量【哭泣】
string预测有多个依据,包括分子结合,表达相关等等,可以根据这些选定感兴趣的预测方式,此外,一般也可以设置阈值,以缩小范围或增加预测数目
您好,最后一个Hitpredict的蛋白互作网络图显示不出来,我找了找也没找到怎么打开这个图,可以指导一下吗?
您好 这个我也不清楚诶,试了一下也不行,可能网络原因。当然表格有数据,你可以用cytoscape自己绘制网络图
这个网页界面太神奇了吧
哈哈,神奇在哪?
可以拖动收集这些线条啊,我在这里玩了半天了