CRISPR-Cas9系统是目前最流行的基因组编辑技术,可以实现目的基因的敲除、插入和突变,该技术是一种由RNA指导Cas蛋白对靶基因定性修饰技术。科学家为了让Cas蛋白定向剪切DNA序列,在CRISPR工作机理的基础上,人为重组了一段目的基因的sgRNA(small-guide RNA),在sgRNA的引导下,Cas9蛋白可以实现对目的基因的定向切割。本文主要介绍,目的基因sgRNA设计和如何将sgRNA构建到CRISPR-Cas相关载体。
1. 目的基因sgRNA设计
目前设计sgRNA的网站比较多,张锋老师实验组总结了sgRNA设计工具如:https://zlab.bio/guide-design-resources,其中synthego(https://www.synthego.com/products/bioinformatics/crispr-design-tool)是小编比较喜欢用的设计工具。不过该网站只能设计人、果蝇和小鼠基因的sgRNA。以人的基因TP53为例,介绍TP53基因sgRNA的设计过程。
1.1 打开synthego网站sgRNA设计工具,点击Launch Design Tool
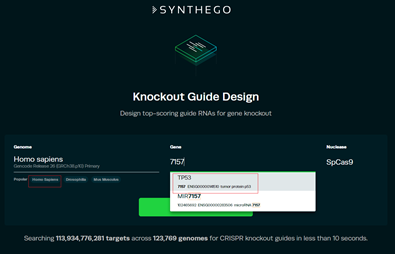
1.2 选择种属人Homo sapiens,人的TP53基因的ID号为7157,选择TP53基因,点击search;
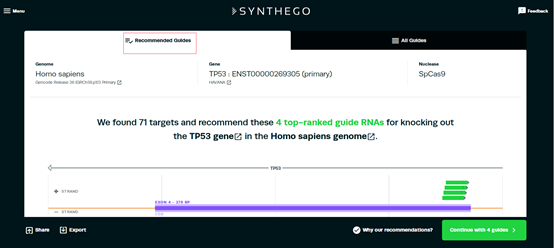
1.3 搜索结果分两部分:推荐的sgRNA和所有的sgRNA。往下滚动滚轮,会看到四条推荐的sgRNA的序列。
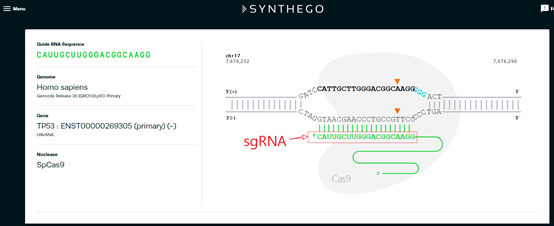
1.4 点击其中一条sgRNA序列,会看到sgRNA在TP53基因上的具体作用位置;
2. sgRNA重组构建
以lentiCRISPRv2载体为例,说明sgRNA是如果构建到该载体中。首先,将设计好的sgRNA中的U碱基改成T碱基,如TP53基因的sgRNA序列为5’-CAUUGCUUGGGACGGCAAGG-3’,修改后的碱基为5’-CATTGCTTGGGACGGCAAGG-3’,该序列的反向互补序列为3’-GTAACGAACCCTGCCGTTCC-5’。正向序列和反向互补序列退火成双链后就可以连接到载体中,但是需要在序列两端添加BsmBI酶切后的同源序列,如下图所示:
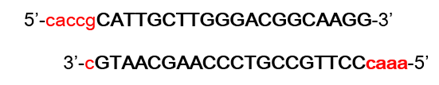
在生工生物合成这两条互补的序列后,退火成双链就可以连接到用BsmBI酶切后的LentiCRISPRv2载体中。sgRNA载体构建具体的操作过程如下。
2.1 LentiCRISPR载体酶切和去磷酸化,用BsmBI在37℃酶切30min;

2.2 琼脂糖凝胶纯化酶切的LentiCRISPRv2载体
LentiCRISPR载体的大小为14,873bp,酶切后产生两个片段,一条大的为目的片段,小片段大小为1885bp。在LentiCRISPRv2载体中有两个BsmBI酶切位点,两个位点之间的片段为1885bp,酶切连接后sgRNA正好在U6启动子下游,连接后的sgRNA下游就是gRNA scaffold序列,与sgRNA一起转录出来发挥gRNA的引导功能。
2.3 磷酸化和Oligo退火成双链DNA;

反应条件如下:37℃ 30min → 95℃ 5min,然后按照5℃/min降温到25℃。
生工生物可以提供磷酸化修饰的引物,可以选择在Oligo的5’磷酸化修饰,就不需要用T4 PNK磷酸化Oligo链了。
2.4 稀释退火的Oligo链;
上一步退火后的双链DNA,按照1:200用水稀释。
2.5 双链Oligo与酶切的载体连接(室温连接10min)

2.6 转化到Stbl3细菌;进行后续筛选。
您好,我想问一下,u626和u629为什么要连入敲除载体
没明白意思,这俩是什么?
请问,我用赛默飞的T4 PNK和T4 连接酶的buffer可以嘛。
当然可以,这只是举例,我用的是这个而已
如果按照这个方法 转化后 菌落老是做不出来 是什么原因呢?
您好,请问sgrna序列可以直接使用网站里(比如chopchop)给的target序列吗?
请问大佬有没有做过 一次混合构建针对一个基因的5条sgrna质粒pool,如果有的话请问具体流程是什么样,是怎么验证pool的丰度的呢,谢谢解答
进哥,想问下你推荐的这个网站有文章引用过吗
老师您好,请问如果我做基因敲入knock in,把够好的LentiCRISPRv2载体和doner质粒共转进目的细胞,然后筛选是不是就可以了,应该不用包病毒吧?