miRNA是近年来研究的热点,掌握miRNA数据库,不仅有利于研究,同时也能够极大地节省科研时间。
以下是目前网上搜罗的一些数据库,相信不少小伙伴都会用到,但同时也有许多不方便的问题,比如,搜索或下载受限,数据库取交集,网站服务器更新,预测信息冗余。
本期介绍的工具不仅是解决了以上问题,还能做出高大上的miRNA-mRNA网络图,是不是很心动呢?
一个高通量实验数据CLIP-Seq(或称为HITS-CLIP,PAR-CLIP,iCLIP)和mRNA降解组测序数据支持的microRNA靶标数据库,包含了miRNA-mRNA,miRNA-lncRNA,miRNA-circRNA,miRNA-ceRNA 和RNA-protein等的调控关系。整合和构建多个流行的靶标预测软件的交集和调控关系。
网址:http://starbase.sysu.edu.cn/
众所周知的microRNA基因注释数据库。目前miRBase只提供了microRNA的靶标的预测软件的链接(如:PicTar)。
网址:http://mirbase.org/index.shtml
整合CLIP-Seq和ChIP-Seq的数据探讨microRNA的转录和转录后调控,构建转录因子->microRNA->靶标的调控网络。
网址:http://deepbase.sysu.edu.cn/chipbase/
一个收集已被实验验证的microRNA靶标数据库。
网址:http://microrna.gr/tarbase/
一个整合的microRNA靶标数据库。整合多个靶标预测软件的调控关系。
网址:http://mirecords.biolead.org/
基于靶mRNA序列的进化保守等特征搜寻动物的microRNA靶基因。是预测microRNA靶标假阳性率较低的软件。而且是microRNA领域大牛Bartel实验室开发的。网址:http://www.targetscan.org/
基于microRNA或microRNA靶标联合作用等特征开发的搜寻动物的microRNA靶基因。假阳性率也较低。是microRNA领域大牛Rajewsky实验室开发的。
网址:http://pictar.mdc-berlin.de/
基于靶位点的可接性和自由能预测microRNA的靶标。是著名的生物信息学家Segal实验室开发的。
网址:http://genie.weizmann.ac.il/pubs/mir07/mir07_data.html/
基于序列特征预测microRNA的结合位点。是几个流行的microRNA靶标预测软件的其中一个。IBM公司的研究团队开发的。
网址:http://cbcsrv.watson.ibm.com/rna22.html/
是著名的MemorialSloan-Kettering 癌症研究中心的研究人员开发的软件和数据库。miRanda的最新版本又叫mirSVR。
网址:http://www.microrna.org/microrna/home.do/
EMBL-EBI的Enright 实验室开发的microRNA靶标数据库。
网址:http://www.ebi.ac.uk/enright-srv/microcosm/htdocs/targets/v5/
整合实验证实的microRNA靶标的数据库。
网址:http://mirtarbase.mbc.nctu.edu.tw/index.html/
整合microRNA表达、靶标和疾病相关信息的数据库。
网址:http://mirgator.kobic.re.kr:8080/MEXWebApp/
动物的microRNA基因及其靶标的数据库。
网址:http://mirnamap.mbc.nctu.edu.tw/
动物microRNA靶标预测和功能注释数据库。
网址:http://mirdb.org/miRDB/
一个基于miRNA-target配对自由能预测microRNA的靶标。
网址:http://bibiserv.techfak.uni-bielefeld.de/
microRNA基因和microRNA靶标数据库。
网址:http://www.diana.pcbi.upenn.edu/miRGen.html/
整合多个以上microRNA数据库(mirwalk2相关tips点这里)。
http://www.umm.uni-heidelberg.de/apps/zmf/mirwalk/
-
下载Cytoscape,http://www.cytoscape.org。
下载与操作系统匹配的版本(支持Mac OS X、Windows 64bit、Linux),下载后安装过程与常规软件相同。
2. 在Cytoscape中下载CyTargetLinker。
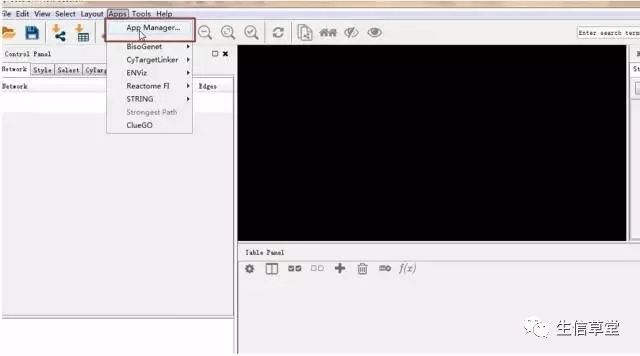
4. 安装完毕后,下载miRNA数据库。
CyTargetLinker | CyTargetLinker website and tutorials
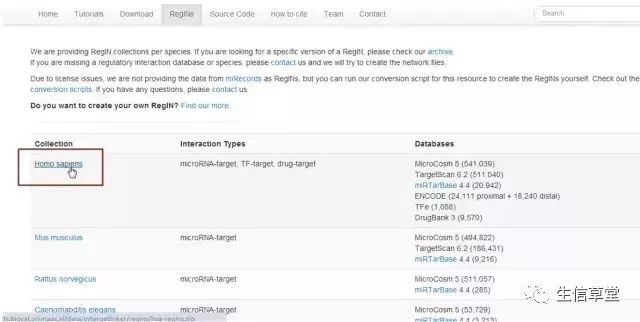
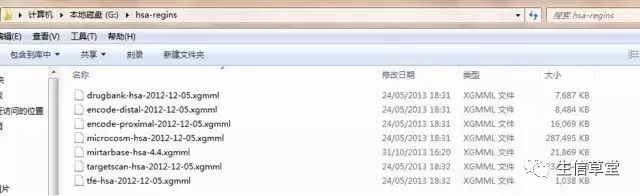
5. 解压下载文件hsa-regins。
6. 输入数据,新建txt文件。可以输入基因名称或miRNA名称,或者共同输入。
输入基因和miRNA个数不受限。
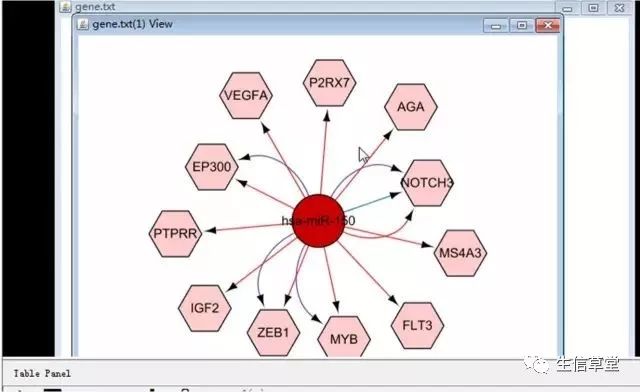
这里我们以TP53和miR-150为例,预测 TP53和miR-150是否存在调节关系,同时探索其他TP53的靶基因或者miR-150调控的基因。
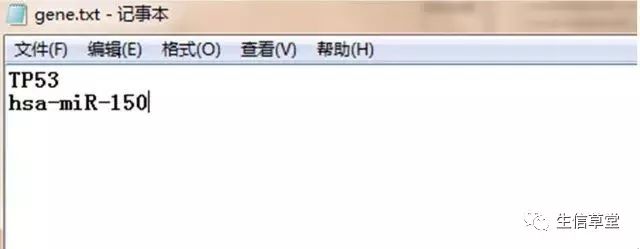
7. 打开Cytoscape,导入 txt文件。
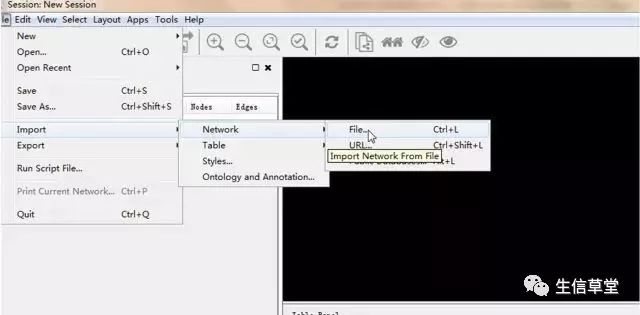
8. 导入后选择高级选项。
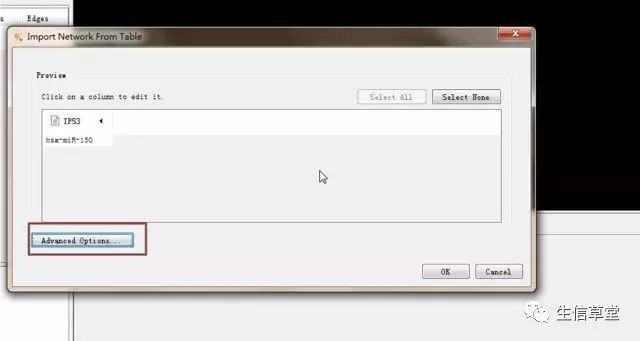
9. 勾选掉如下图的选项。
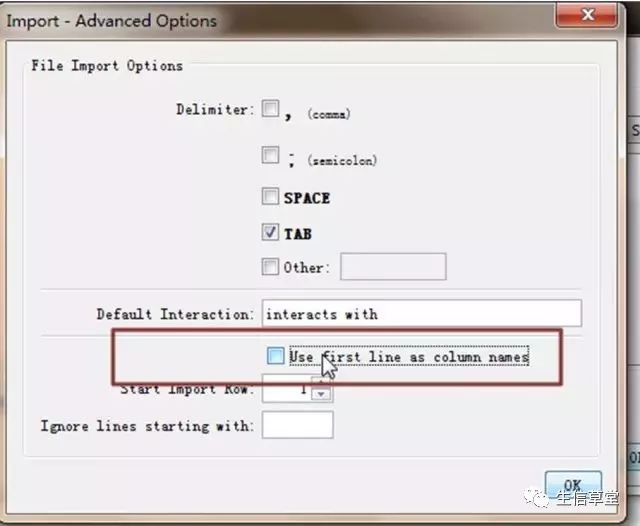
10. 单击如图column1和source node,后选择ok。
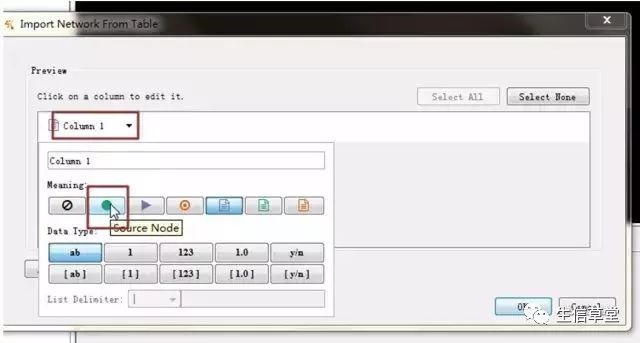
11. 忽略以下提示,选择是,即可。

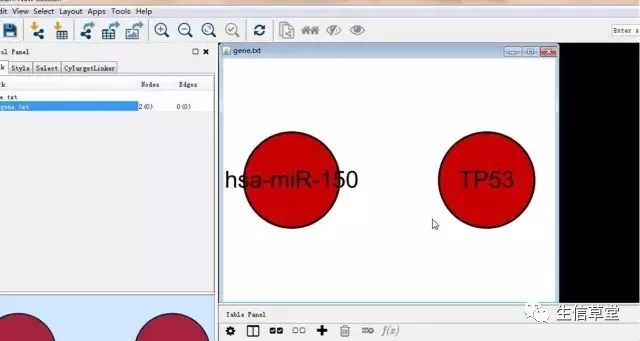
12. 如下图,导入成功。
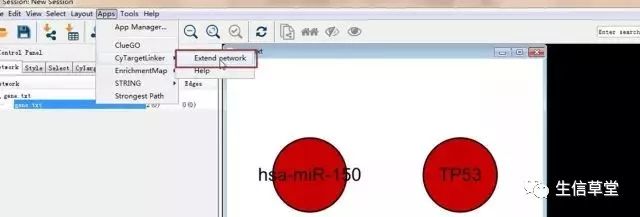
13. 点击安装CyTargetLinker中的拓展网络。
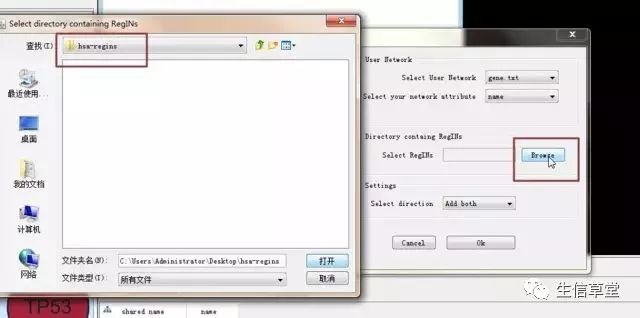
14. 加载数据库,即下载的hsa-regins文件,点击ok。
15. 勾选掉不是miRNA的数据库。
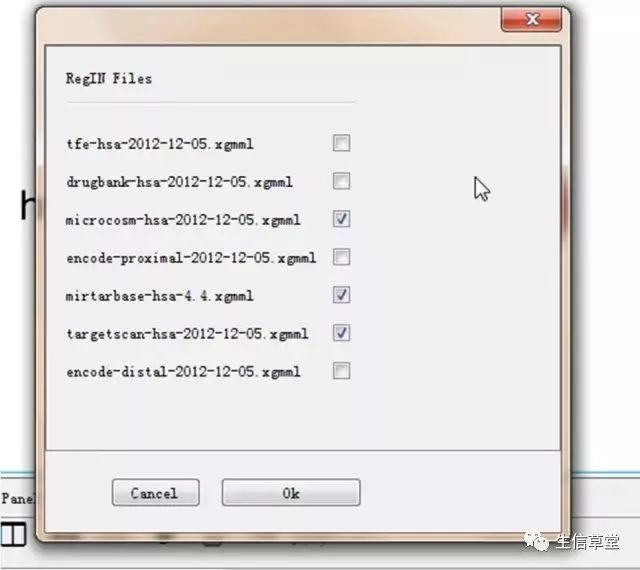
16. 等待运行结果。
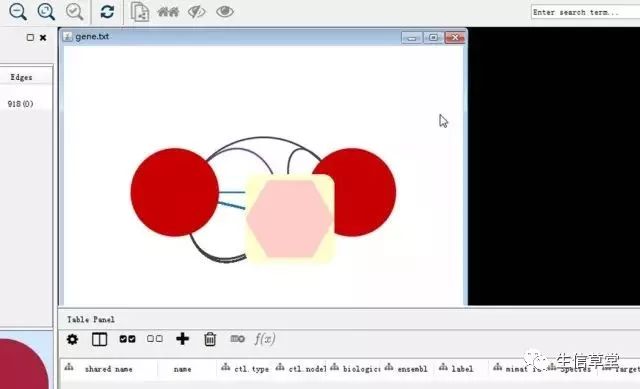
17. 出现以下如图结果,即运行成功。
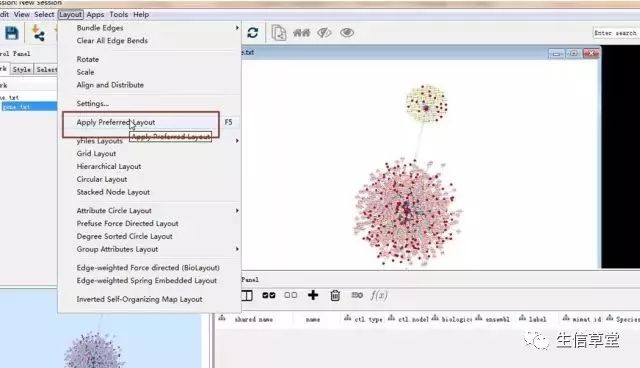
18. 重新布局。点击如图选项,重新布局。
19. 据库来源,p值,实验支持的类型,文献来源pmid号。
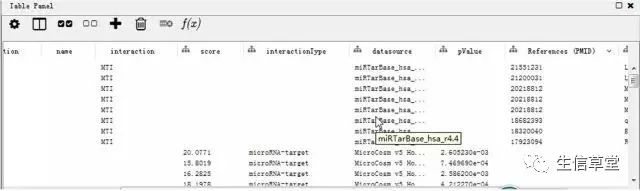
20. 选取高质量的预测信息。
在进行miRNA靶基因预测时往往会得到大量的预测信息,我们根据是否实验支持,文献报道,p值高低来缩减我们研究范围。
这里只选取实验支持的结果来构建网络。
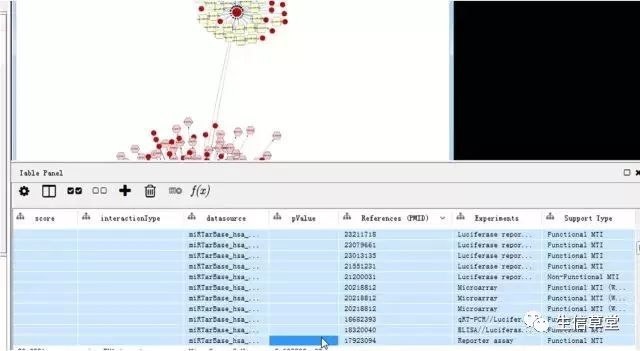
21. 在edge table中选择好要实验支持的edge,点击如下图的选项。
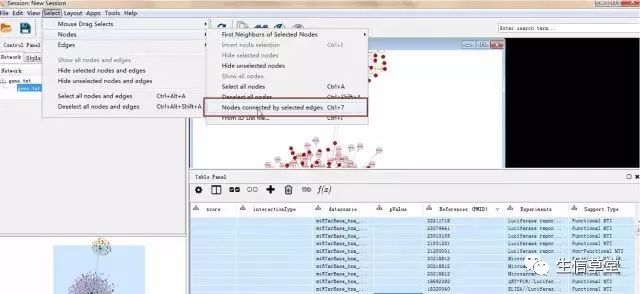
22. 构建高信度的网络。
点击如图的选项。
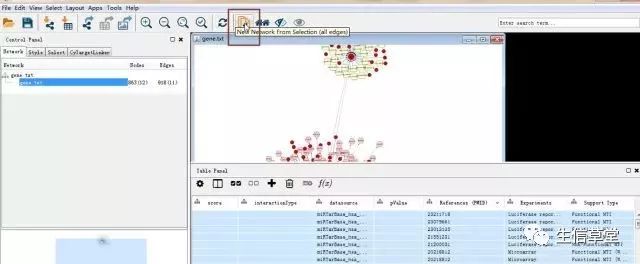
23. 生成了原网络的高信度的miRNA-mRNA子网络。
你好,老师,第四步下载miRNA数据库网站打开好像不是下载数据库的界面,请问可以再发一遍下载miRNA的数据库网址连接吗
https://cytargetlinker.github.io/pages/linksets
这个网址变了好多次,这个应该没问题了
想请问下如何通过下载的TargetScan数据然后利用Cytoscape作图
“4. 安装完毕后,下载miRNA数据库”请问用的是哪个miRNA数据库呢,可以提供下链接吗?
楼主提供的下载miRNA的数据库链接似乎有误,应该是这个链接http://projects.bigcat.unimaas.nl/cytargetlinker/regins,不过我一直打不开,不知道楼主是否有下载好的文件,是否可以分享一下
底下评论回复有放最新的链接:https://apps.cytoscape.org/apps/cytargetlinker
github也有:https://github.com/CyTargetLinker/
可以github下载安装 也可以直接软件里面Apps–>搜索这个插件安装更方便
您好,请问您mirna的数据库如何下载下来的呀
你下载了吗,我也是一直打不开
老师,您好,步骤的第四条:安装完毕后,下载miRNA数据库http://projects.bigcat.unimaas.nl/cytargetlinker/regins/ 这个网址打不开,请问您能再发一个网址吗?谢谢老师!
不好意思 刚刚看到 https://apps.cytoscape.org/apps/cytargetlinker
github也有:https://github.com/CyTargetLinker/
好的,谢谢老师!
“4. 安装完毕后,下载miRNA数据库”请问用的是哪个miRNA数据库呢,可以提供下链接吗?