- 1.在 UCSC 下载 hg19 参考基因组;
- 2.从 gencode 数据库下载基因注释文件,并且用 IGV 去查看感兴趣的基因的结构,比如TP53,KRAS,EGFR 等等。
- 3.截图几个基因的 IGV 可视化结构
- 4.下载 ENSEMBL,NCBI 的 gtf,也导入 IGV 看看,截图基因结构
- 5.了解 IGV 常识
来源于生信技能树:http://www.biotrainee.com/forum.php?mod=viewthread&tid=1750#lastpost
1 UCSC下载hg19参考基因组
测序得到的是几百bp的短read, 相当于把拼图打散了给你。如果没有参考基因组,从头(de novo)组装等于是重走人类基因组计划的老路,也就是打散了拼图,却不告诉你原来是什么样子,那么任务将会及其艰巨。
还好人类基因组已经组装好了,我们只需要把我们测得序列回贴(mapping)回去,毕竟人与人之间的差距只有不到1%差异, 允许mismatch就行。
因此第一步就是要去UCSC(http://genome.ucsc.edu/index.html)下载hg19参考基因组(文献要求)
那下载哪个基因组呢?先了解一下:
https://bitesizebio.com/38335/get-to-know-your-reference-genome-grch37-vs-grch38/
hg19、GRCH37、 ensembl 75 这 3 种基因组版本应该是大家见得比较多的了,国际通用的人类参考基因组,其实他们储存的是同样的 fasta 序列,
只是分别对应着三种国际生物信息学数据库资源收集存储单位,即 NCBI,UCSC 及ENSEMBL 各自发布的基因组信息而已。有一些参考基因组比较小众,存储的序列也不一样,比如 BGI 做的炎黄基因组,还有 DNA 双螺旋结构提出者沃森(Watson)的基因组,还有 2016 年发表在 nature 上面的号称最完善的韩国人做的基因组。前期我们先不考虑这些小众基因组,主要就下载 hg19 和 hg38,都是 UCSC 提供的,虽然 hg38 相比 hg19 来说,做了很多改进,优点也不少,但因为目前为止很多注释信息都是针对于 hg19 的坐标系统来的,我们就都下载了,正好自己探究一下。也顺便下载一个小鼠的最新版参考基因组吧,反正比对也就是睡个觉的功夫,顺便分析一下结果,看看比对率是不是很低。(http://www.bio-info-trainee.com/1985.html)
基因组各种版本对应关系:http://www.bio-info-trainee.com/1469.html
# 下载USCS版本的hg19$ mkdir /mnt/f/rna_seq/data/reference && cd /mnt/f/rna_seq/data/reference
$ mkdir -p genome/hg19 && cd genome/hg19
$ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg19/bigZips/chromFa.tar.gz & tar -zxvf chromFa.tar.gz# 解压,得到所有染色体的信息# 将所有的染色体信息整合在一起,重定向写入hg19.fa文件,得到参考基因组$ cat *.fa > hg19.fa# 将多余的染色体信息文件删除,节省空间$ rm -rf chr*备注:用wget下载速度实在龟速,下下来显示需要N天,最后我用迅雷下载,用时大概3h。
这个时间我们可以去下UCSC看看…….
2 下载基因组注释文件
然而参考基因组是一部无字天书,要想解读书中的内容,需要额外的注释信息协助。因此第二步,就是去gencode数据库(http://www.gencodegenes.org/)下载基因组注释文件
这里有基因组版本对应信息
https://www.gencodegenes.org/releases/19.html

Release 19(GTF_GFF3 files).png
下载基因组注释文件
cd /mnt/f/rna_seq/data/reference/genome/hg19
wget ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/release_26/GRCh37_mapping/gencode.v26lift37.annotation.gtf.gz
$ wget ftp://ftp.sanger.ac.uk/pub/gencode/Gencode_human/release_26/GRCh37_mapping/gencode.v26lift37.annotation.gff3.gz
$ gunzip *.gz && rm -rf *.gz这个下载速度还是完全可以接受
3 IGV(Integrative Genomics Viewer)Integrative Genomics Viewer下载和使用
igv有几个版本
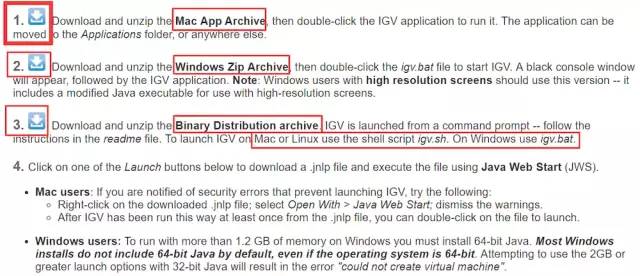
igv各个版本.png
-
1 下载windows版本,安装到本地进行可视化
下载地址:http://data.broadinstitute.org/igv/projects/downloads/2.4/IGV_Win_2.4.13.zip
安装后解压缩,
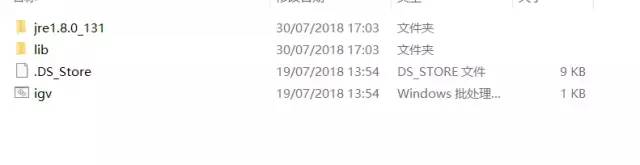
igv24_hm.png
igv.bat打开,稍等便可打开。
-
2 下载linux版本,命令行打开
# 进入IGV官网,并下载相应的软件包,有Windows,Mac,和LINUX,这里我下载Linux二进制包$ cd ~/src
$ wget http://data.broadinstitute.org/igv/projects/downloads/IGV_2.3.97.zip
$ unzip IGV_2.3.97.zip && mv IGV_2.3.97 ~/biosoft# 添加环境变量$ vim ~/.bashrc
PATH=$PATH:~/biosoft/IGV_2.3.97
$ source ~/.bashrc# 运行IGV,Linux直接运行igv.sh可以开启IGV窗口,但是会比较慢,要耐心等待。$ igv.sh