检测甲基化状态的方法比较多,目前被认为金标准的是亚硫酸盐的测序法(bisulfite sequencing PCR,BSP)。
BSP原理
用亚硫酸氢钠修饰处理基因组DNA,所有未发生甲基化的胞嘧啶(C)被转化为尿嘧啶(U)而甲基化的胞嘧啶则不变。
基因组DNA经亚硫酸盐处理后,设计BSP引物扩增目的片段,此时尿嘧啶(U)全部转化为胸腺嘧啶(T),最后对PCR产物进行测序就可以判断CpG位点是否发生甲基化。
BSP实验流程:
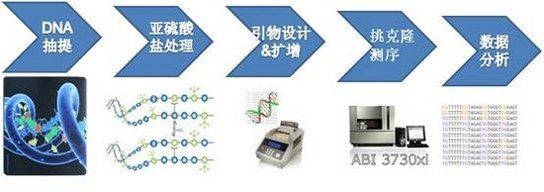
DNA抽提和亚硫酸盐处理都可参考相应的试剂盒进行操作
引物设计网站网址如下:http://www.urogene.org/cgi-bin/methprimer2/MethPrimer.cgi
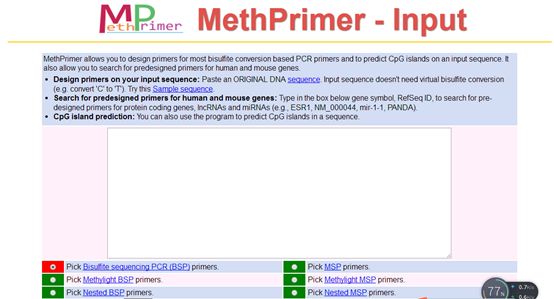
网址引物设计界面如上图,可选择设计什么类型的引物,此处选择设计BSP引物。
该网站可提供CpG岛的预测等功能,所以可选择通过预测CpG岛后用于引物设计的选项,如下图:
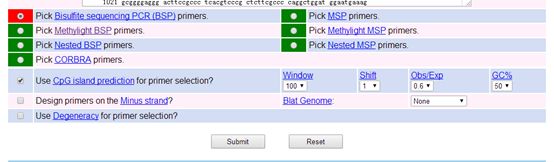
其他参数选择默认值即可,仍以人的GAPDH启动子序列为例
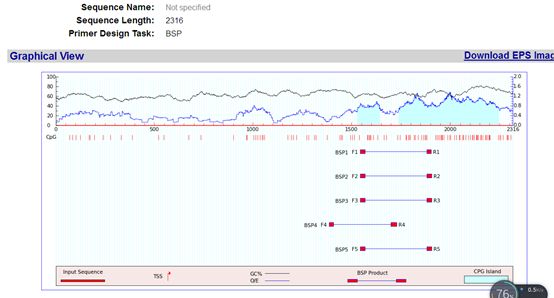
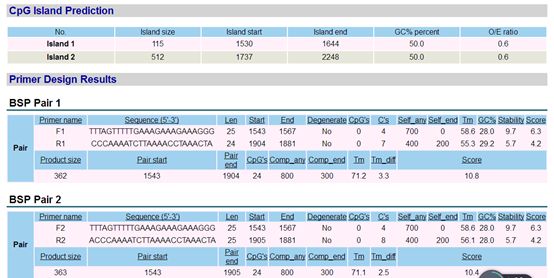
设计结果如上,预测到有2个CpG岛,根据测的结果反馈设计的引物如上图
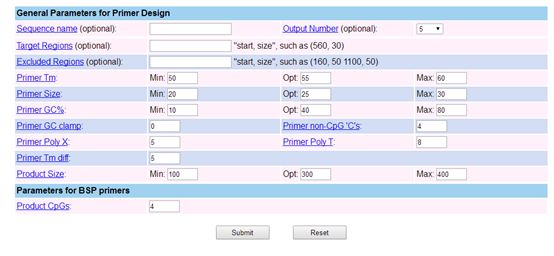
另外可根据自己的需要调整引物设计的相关参数,如下图
另外预测结果中还给出了亚硫酸盐处理前后序列的变化情况、引物对应的位置以及CpG位点的位置情况:


另外在设计引物中还有相关注意事项:
用于BSP实验的DNA是经过亚硫酸盐处理,将未甲基化的C碱基转变为U碱基,而甲基化的C碱基不变,从而将甲基化的差异转变成碱基上的差异,所以在后续进行PCR扩增引物设计的时候需要注意:
1、引物设计不能含有CpG位点,以避免造成发生甲基化和未发生甲基化DNA的差异。
2 、引物扩增的片段能包含尽量多的CpG位点,BSP扩增片段长度300-500bp,包含的CpG位点越多则引物的打分越高。
BSP引物设计完成后应进行PCR扩增直接测序或者通过TA克隆测序分析甲基化频率。
BSP直接测序结果
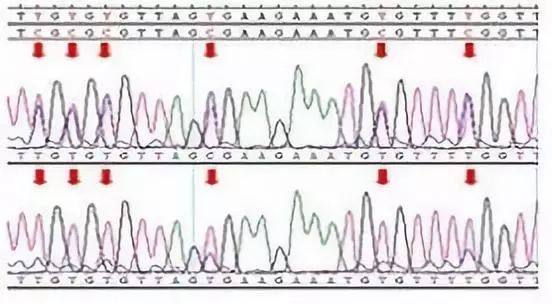
BSP点状图

空点表示为未发生甲基化的CpG位点,实点表示发生甲基化的CpG位点
BSP柱状图

您好,亚硫酸盐处理前采用物理破碎DNA后在转化,最后得到的文库峰图有拖尾现象是什么情况呢?是PCR循环多了吗?input DNA投入100ng,建库的。
您好,我想问一下亚硫酸氢盐处理后的DNA 对于某个基因某个片段进行测序发现有的C全部未转化,有的序列C是部分转化为T,而有的是全部转化为T,我这边认为全部没转化的可能是高甲基化也可能是DNA没处理完全的问题,而这种全部转化为T的就是甲基化程度低吗?还是说会有其他原因导致的呀?
同一片段,甲基化程度高的测序测通了,甲基化程度低的测到中间断了(只要甲基化程度低的都这样),这是什么原因呢
老师好,您觉得BSP引物设计好后有必要用blast检测特异性吗?普通PCR+琼脂糖凝胶跑一下看看条带位置即可吗
你好,看文献里面计算单基因甲基化水平,做的柱状图。请问根据单克隆测序序列,可以用哪个网站或者软件计算甲基化水平,分CG,CHG和CHH,然后再作图。
进哥哥,BSP引物设计完成后应进行PCR扩增直接测序或者通过TA克隆测序分析甲基化频率。这两种测序方式有对结果的影响大不?
请问有bsp法测序出来结果,进行数据分析,计算甲基化程度的网址吗?之前用的那个不能用了
老师您好,请问BSP作为半定量法,挑菌测序需要挑菌多少个比较合适呢?我看有人说至少20个才能保证准确。
分析是用R分析吗?
老师,您好,我这边将样本进行亚硫酸氢盐处理之后pcr条带不明显,未处理的pcr之后条带比较亮
重亚硫酸盐处理之后会损失不少,如果确定有条带,可以增加循环数,没有的话就重新设计引物
老师,您好,我将未进行亚硫酸氢盐处理的DNA样本和亚硫酸氢盐处理后的DNA样本进行pcr,电泳条后都有条带,只是未处理的条带比较亮
你好,不好意思 刚刚看见,可能由于之前迁移服务器的原因导致的,其实还是可以显示的 右击那个没有显示的小方格,加载映像即可
当然 我看看能否修复一下
这个方法羟甲基化的胞嘧啶也会保持不变吗?5mc,5hmc,5fmc都会被识别成5mc?
你好,羟基化和甲基化(5mc,5hmc)都不会被转化,5fC和没有任何修饰的C就被转化成U
您好,请问如果是TA克隆测序,根据测序结果,怎么分析三种环境的甲基化水平呢,或者总甲基化水平,想用柱状图分析,类似定量的方式
可以的 有专门的用于BSP seq的R语言分析包,可以做到半定量,我抽空做个小程序分享给大家
好的,是不是可以根据测序序列,看碱基的变化来统计发生甲基化位点
可以的
请问根据测序序列,怎么统计分析甲基化水平啊,可以出一片关于bsp数据分析的文章吗(根据他人的文献)
OK,最近有空整一下,也抽空做个小程序进行分析
您好,麻烦问一下你亚硫酸氢盐处理的DNA样本pcr后电泳条带和未处理的DNA样本pcr电泳条带之间有什么不同吗
你好,请问这高小程序的名称是什么?在你的主页中搜索不到。
重亚硫酸盐转换后的DNA跑胶为什么会没条带?
重亚硫酸盐处理之后DNA会被破坏打断,所以原来基因组的那一条带不会存在 在低分子量位置有弥散条带就是处理之后的
如果进行过了WGBS的话,DMR在该基因的外显子和内含子上,那么这个DMR如何验证呢,DMR在内含子上的话,这个基因是否有关注的必要呢,麻烦老师了
您好,对于确定位点的验证,建议BSP测序验证,比较简单直接;
内含子区可能价值不大,最好还是启动子区的,减少试错可能性,当然时间足够可以同时做做
非常感谢您的答复
麻烦问一下后续的分析图是从那个网站分析的,谢谢。
点状图吗 一个比较老的软件BiQ_Analyzer
很全面!