之前已经介绍过Snapgene的安装与使用,我们在进行分子生物学设计的时候经常用到这个工具:

这样:
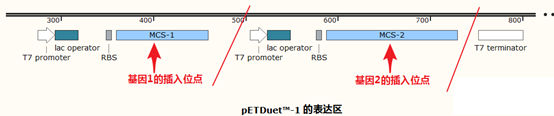
还有这样:
下面介绍一下如何利用SnapGene设计SNP位点的RFLP检测和扩增测序方案:
序列获取
软件给出的结果一般都是以下形式,给出的只有突变所在基因组位置或者区间信息。
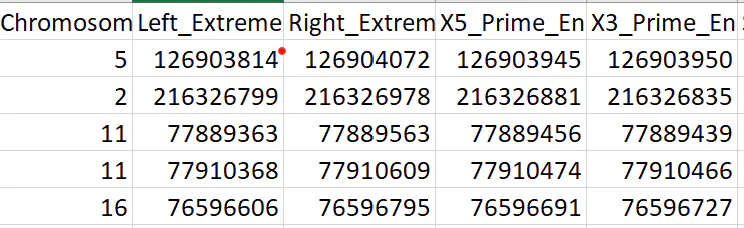

那么该怎么去设计PCR验证实验的引物呢?
首先,我们这里对snv取鉴定到的位点的上下游各400bp,indel的话是区间位置的上下游200-300bp。
拿上图的snv举例,它的Start和End分别是48556379和48556379,那么各加400bp,则位置变为:48555979和48556779。同时要注意这里注释出来时ABBCA13这个基因。
那么接下来就先在NCBI的Nucleotide数据库检索这个基因:
然后选择第一个检索出来的GENE的链接:
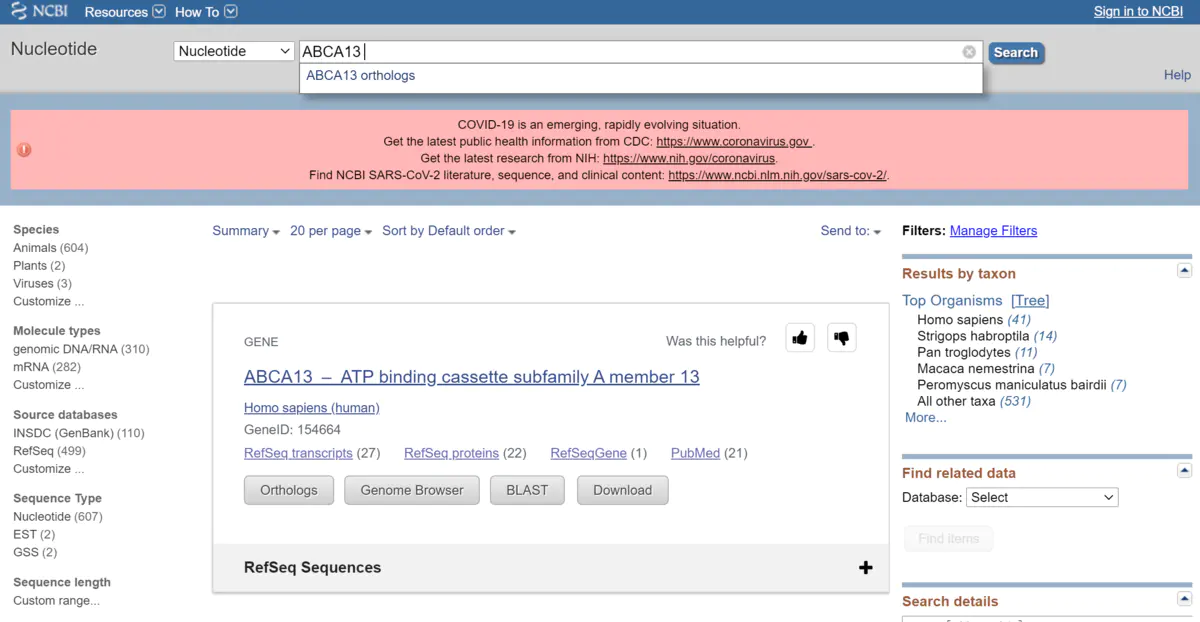
再把页面下拉到“Genomic Regions, transcripts, and products”这边,点击右边的“FASTA”
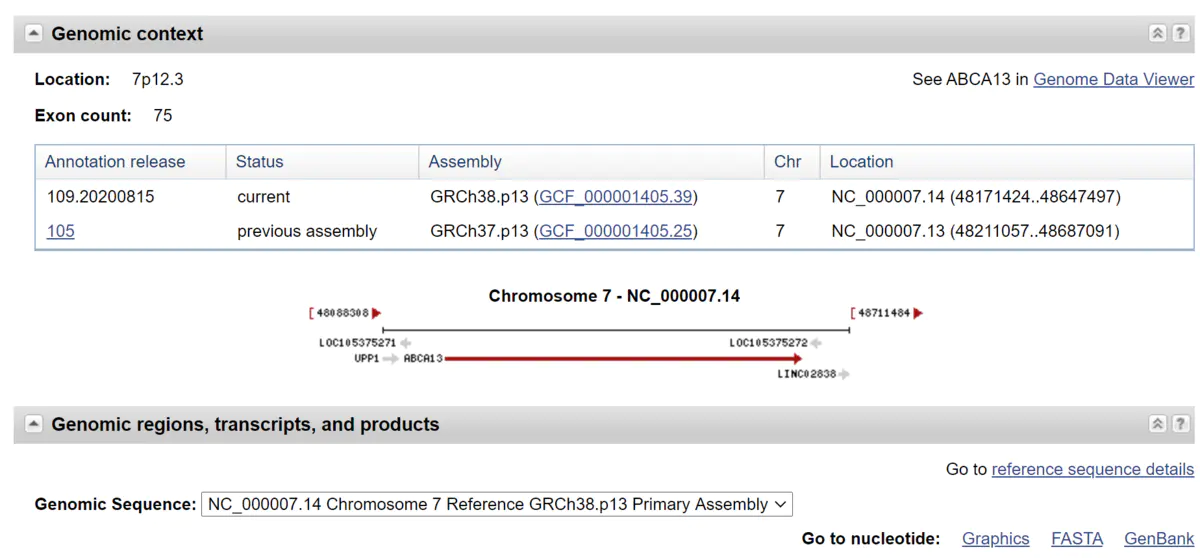
这时候可以看到右边有一个”Selected region”
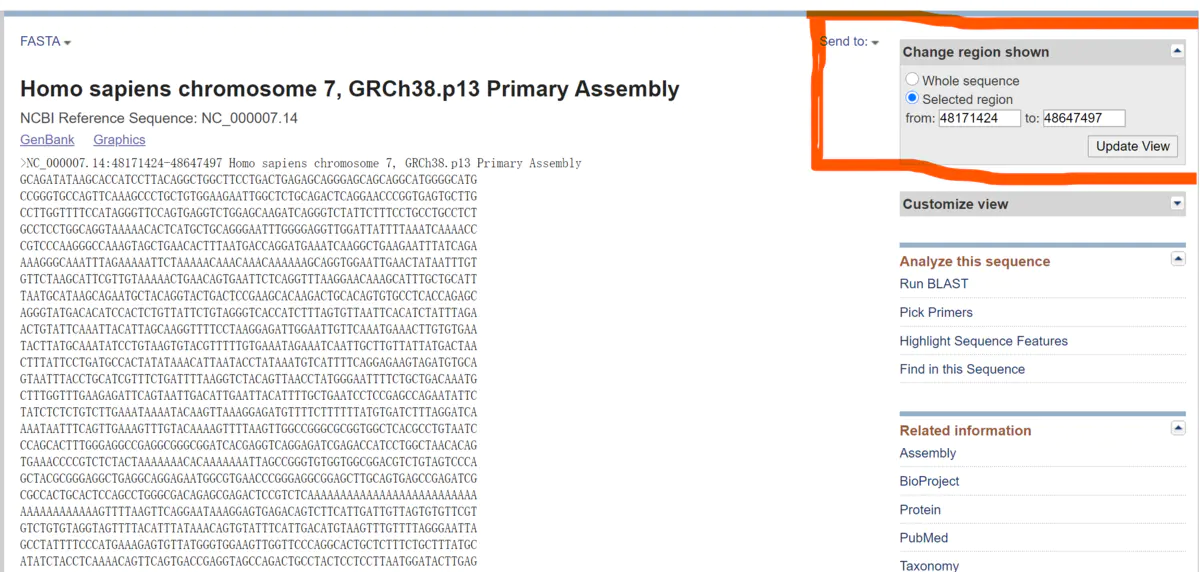
最后再把刚才取好的上下游400bp扩展后的基因组位置输入到这个区间,就得到这段的序列了,然后根据序列使用Primer premiere设计验证引物即可。

扩增出序列之后进行Sanger测序,验证SNP或Indel位点。
RFLP检测
RFLP是指DNA片段的限制性酶切片段多态性,SNP的限制性片段长度多态性(restriction fragment length polymorphism,缩写RFLP)检测流程如下:
1. 选择合适的限制性内切酶,该酶能够特异性识别SNP位点的碱基种类,当SNP位点碱基变化时,能够使该酶失去原有的识别位点;
2. 设计扩增引物,将包含SNP位点的一段DNA扩增下来,且保证其中只有一个该酶的识别位点;
3. 使用限制性内切酶对产物进行酶切、电泳,通过产物带型来确定SNP的碱基种类。
因此,该方案的核心有两个:限制性内切酶的选择及扩增引物的设计。
一、 限制性内切酶的选择
由于SNP位点出现的随机性,很难一眼就看出该序列位于什么酶的识别位点,而使用SnapGene软件,就非常容易了。
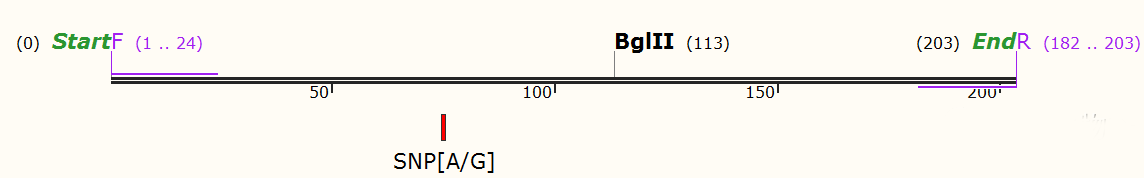
举个例子,检测RS2267668 [A/G]多态性:
【1】将SNP位点分别设置为A和G,得到两条序列,将序列都放入SnapGene,开启左上角酶切位点按钮,建议最初选择所有的(All Commercial)

【2】 对比两者,发现当SNP位点碱基为G时,多出一个酶切位点:Hpy188I。这就意味着,可以使用该酶进行该SNP位点基因型的检测。
限制性内切酶选择结束,简单而高效~
二、 设计扩增引物
从序列的酶切位点分布看,这个酶有四个识别位点:
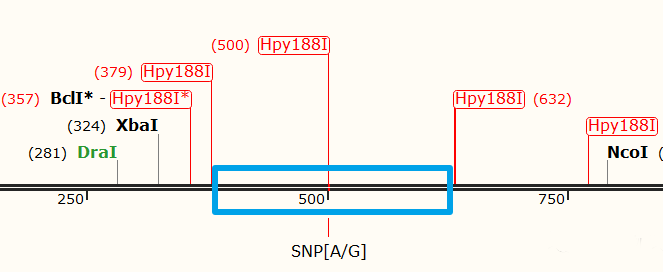
本着RFLP的引物设计原则,应该保证扩增产物中只有一个识别位点,因此可以在上述蓝色方框范围内使用Primer premiere进行引物设计,使产物长度为200~1000bp,且酶切位点左右序列长度差异>100 bp以保证电泳带型明显。
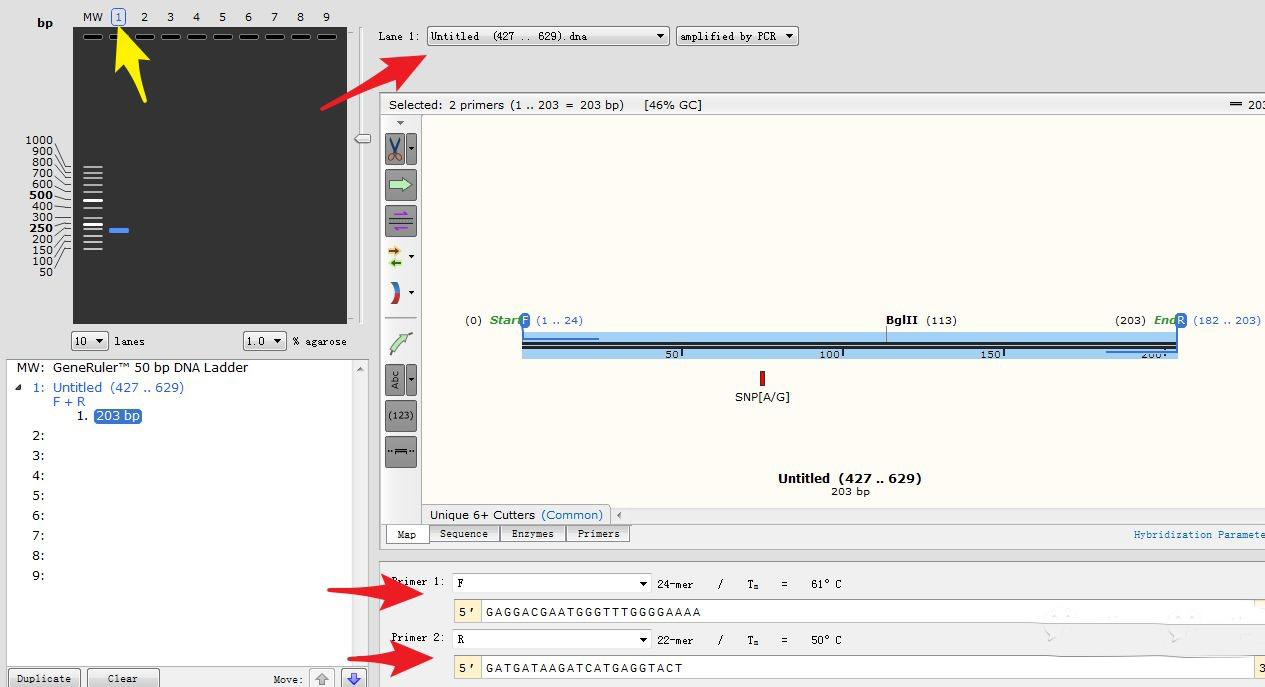
引物设计好之后,将F/R引物的扩增产物新建一个SnapGene文件,准备进行PCR电泳预测:
点击“Tool”标签栏中的“模拟DNA电泳”,弹出新的页面,其中:
点击电泳图中的MW,选择需要用到的Marker:
点击泳道1,选择使用F和R引物扩增该条带:
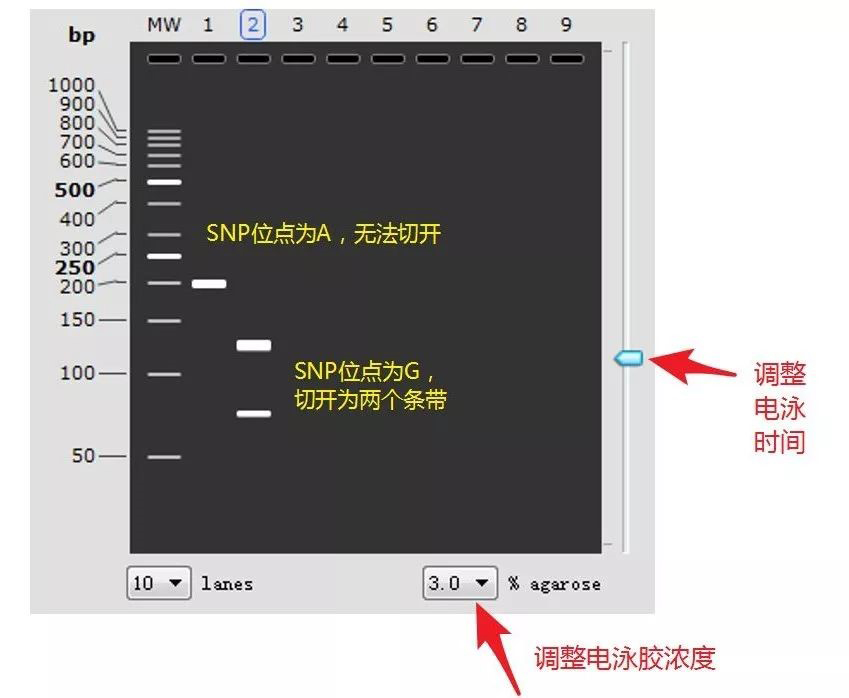
点击泳道2,选择使用Hpy188I酶切扩增条带:
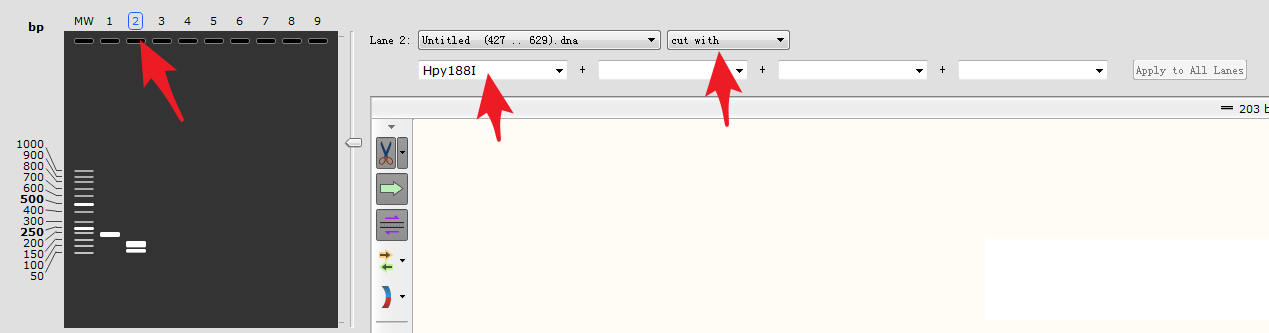
最后调整一下胶浓度和电泳时间,就得到了完美的模拟电泳条带图,以他为目标,放手去做吧!
王博士,您好!拜读了您的这一篇文章后,我通过您的方法筛选到了目的SNP位点出现的核酸内切酶,并且也通过了在SNP位点出现的内切酶位点处进行PCR扩增后额外加入内切酶的方法,在电泳图上获得了您所绘制的最后一张模拟电泳图,非常感谢您的分享。但是我有一个问题,想请教您一下:我想通过在设计引物的过程中引入内切酶序列的方法直接鉴别出未突变序列和突变序列,因此我在原引物的5’端都引入了SNP位点出现的新核酸内切酶和保护碱基,但是均不能达到预期的效果,该引物在未突变序列和突变序列上跑出了相同bp位置的条带。必须通过先PCR扩增再额外加NEB的内切酶方式才能达到一张电泳图鉴别两条序列的效果,因为我是一名刚进入分子研究领域的小白,望您给予指导建议,多有打扰,望您体谅!
您好,不好意思,没太明白你的意思 如果还没有解决的话可以加微信讨论一下,导航栏我的简历有手机号码