为什么要分割GTEx数据
GTEx数据库(https://gtexportal.org/home/datasets)中有人体各个器官和组织的转录组。之前的有关ACE2教程,都是用这个数据库中的数据完成的。当然数据是开放下载的,Xena研究机构提供了标准化以后的数据(下载链接是https://toil.xenahubs.net/download/gtex_RSEM_gene_tpm.gz, https://toil.xenahubs.net/download/GTEX_phenotype.gz )这个数据下载解压以后有大小有3G+,一般笔记本是无法打开的,用内存大一点的服务器读入也需要一段时间。
教程里使用的是肺组织的数据,那么这个数据是如何分割的呢?
代码
setwd("G:\\GTEx/split")
GTEx_phenotype <- read.delim(file="G:\\GTEx/GTEX_phenotype.gz",header=T,as.is = T,row.names = 1)
GTEx_Tpm<-read.delim(file="G:\\GTEx/gtex_RSEM_gene_tpm.gz",header=T,as.is = T,row.names = 1)
GTEx_phenotype_split<-split(GTEx_phenotype,GTEx_phenotype$X_primary_site)
GTEx_Tpm_split<-list()
colnames(GTEx_Tpm)<-stringr::str_replace_all(colnames(GTEx_Tpm),"[.]", "-")
GTEx_Tpm_split<-lapply(GTEx_phenotype_split,
function(x){
xxxx<-GTEx_Tpm[,colnames(GTEx_Tpm)[colnames(GTEx_Tpm) %in% rownames(x)]]
write.csv(xxxx,file=paste(x[2,1],".csv",sep=""))})
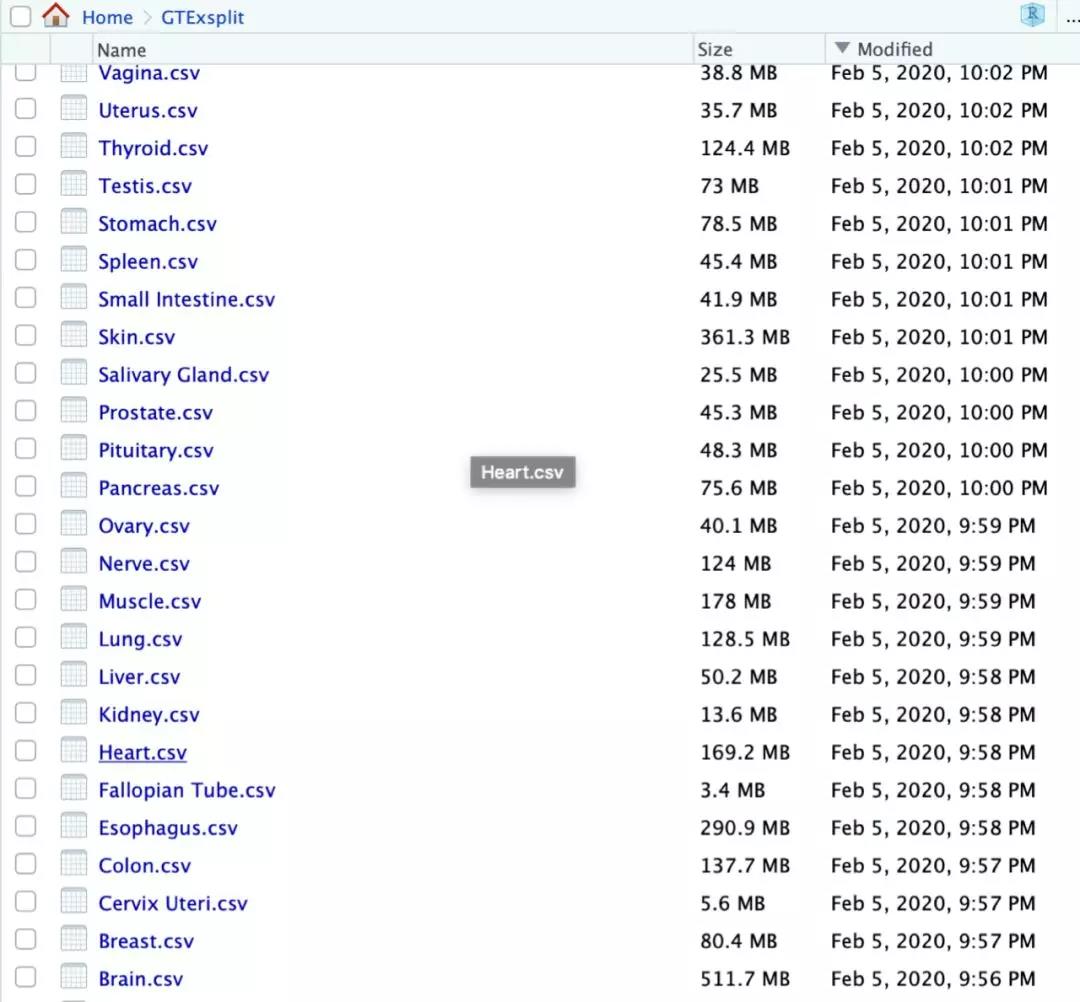

就得到了上面这些文件
当然用同样的方法,我们还可以分割泛癌转录组数据
fastSave::load.pigz(file="tcga_gtex_tpm.RData")
TCGAsample_split<-split(XenaSampleClin,XenaSampleClin$Tumor)
TCGA_TpmOS_split<-list()
TCGA_TpmOS_split<-lapply(TCGAsample_split,
function(x){
xxxx<-TCGAxenaExpOS[rownames(x),]
write.csv(xxxx,file=paste(names(x),".csv",sep=""))})
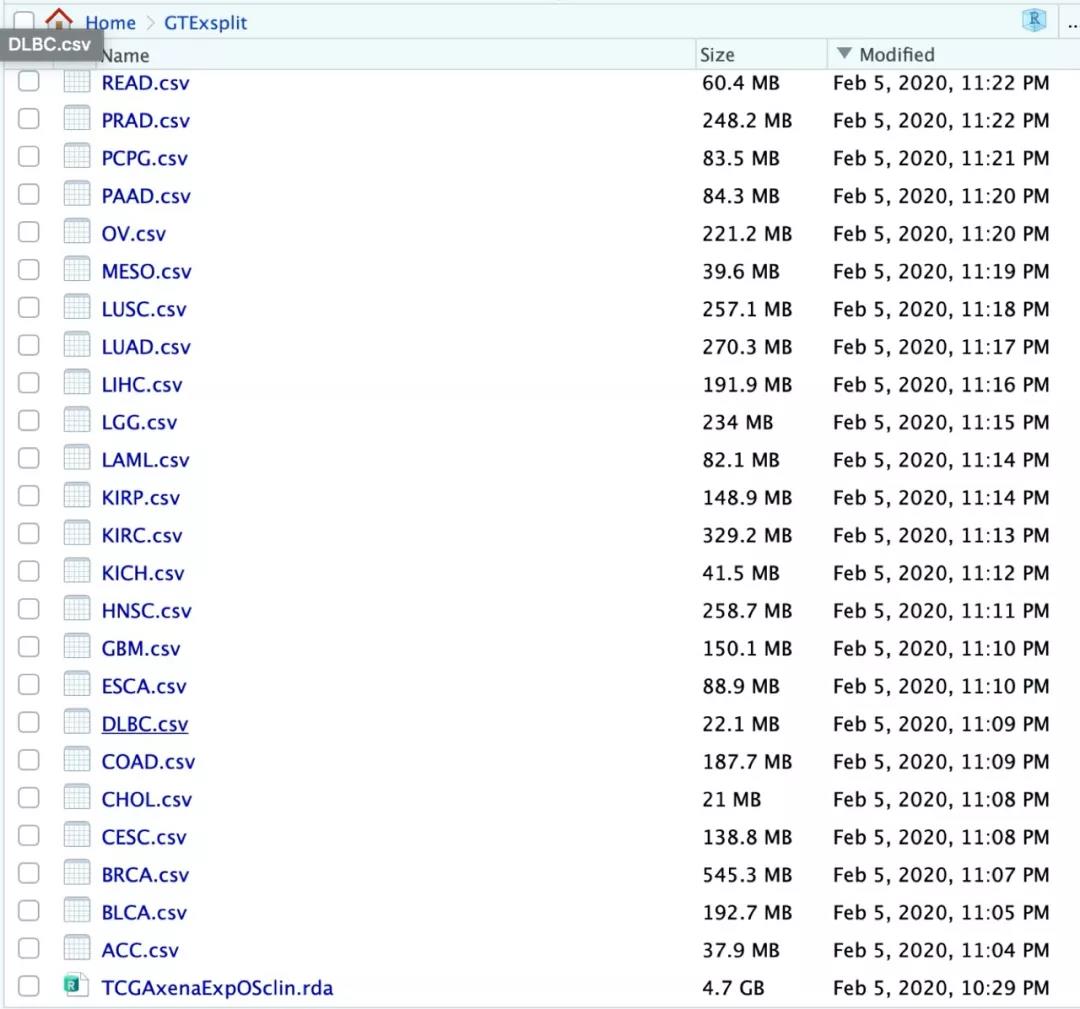
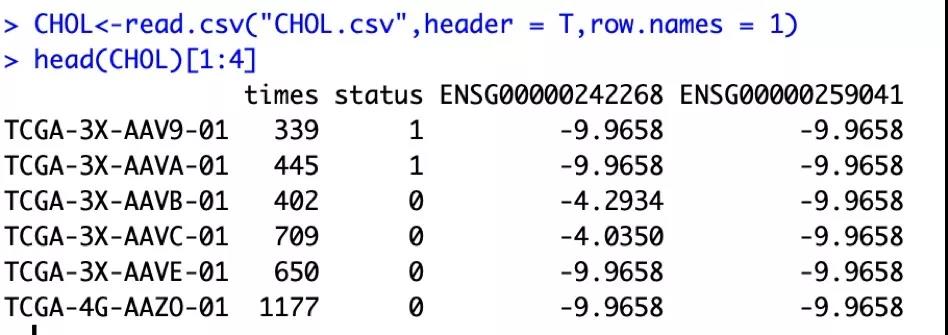
对了,这个泛癌数据是是带有生存时间与生存时间的数据。
素材:
代码不难,用split+lapply就实现了
分割好的数据:
https://pan.baidu.com/s/17blyTZb-Kni8u9yqIsweyA?pwd=eqsp
提取码:eqsp
进哥,为啥fastSave 系统说没有呢
需要提取TCGA-GTEx-TARGET-gene-exp-counts.deseq2-normalized.log2.gz这个文件中的表达谱数据,试用上面的代码,电脑内存8G,文件太大了读不进R
读取不了就没有办法诶 可以使用emeditor软件读取 手动拆分一下文件 把tcga gtex target先拆开来
您好,请问下我按您的代码分割保存数据后,为什么再用R读取数据后会出现数据都聚集在第一列,而其他列却没有对应的数据。但我用Excel打开这个数据表时,却是正常排列的。
设置的分隔符不对 也就是sep参数 CSV是, txt是制表符\t
你看一下
好,排序正常了,感谢进哥!!
我分割出来的文件,第一行是1,2,3,4,5.。。。。。,不是基因名,请问如何改成第一列输出基因名
文中代码少了一个参数,读取GTEx数据需要设置行名为基因名