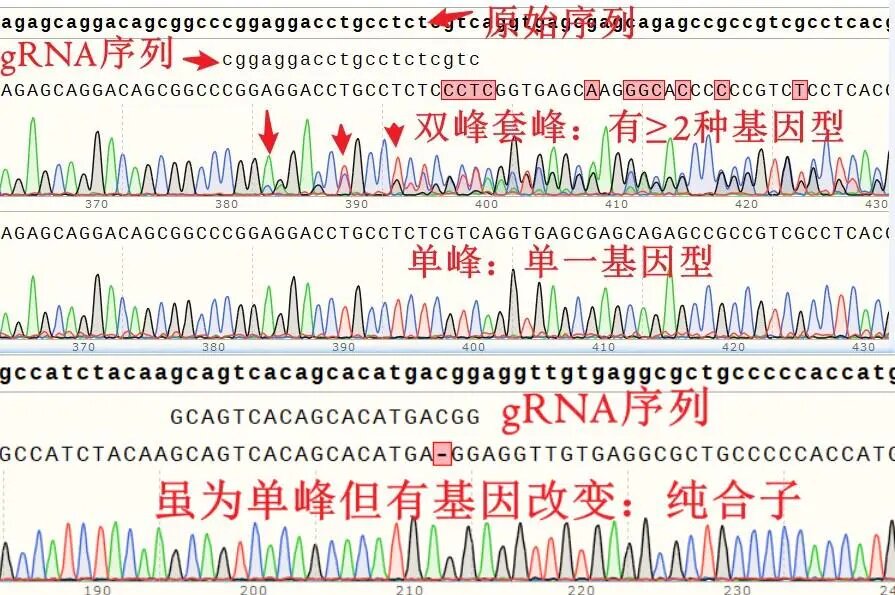
1. CRISPR-Cas9基因编辑结果可能出现的峰图
我们以依赖NHEJ修复的CRISPR-Cas9基因敲除为例,可能出现的峰图如下图:
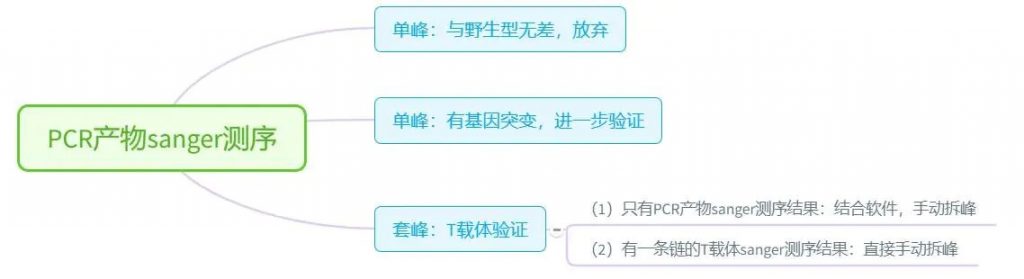
依据PCR产物峰图结果,我目前的工作流程如下:
2. 为什么要手动拆峰?
我们需要最终的基因型sanger测序结果,用于文章发表。在一切顺利的情况下,其实不花多少时间。但如果T载体连接失败或者总是只测到单边序列,急于知道基因编辑结果时,可以选择手动拆峰。
我目前的手动拆峰方法是来自于DZ师兄的言传身教,如果大家有什么新的方法,可以在评论中指出,感谢~我们的手动拆峰方法要求一定要有以下两个结果:
(1)PCR sanger测序结果:通过实验获得
(2)其中一条链的序列:通过实验获得,或者通过工具获得。
3. Poly Peak Parser:拆峰工具
PolyPeakParser: Run Poly Peak Parser in sangerseqR: Tools for Sanger Sequencing Data in R:https://github.com/jonathonthill/sangerseqR
如果已有实验结果获得了其中一条链的序列,我们可以直接手动拆峰。当T载连接实验总是失败时,我们也可以依据PCR产物sanger测序峰图,使用网页Poly Peak Parser推导其中一条链的序列。该网页工具的界面和使用方法都非常简单,在推荐给大家之前,我使用自己的测序结果验证过,目前没发现有错的结果,可以十分精准的推测出二倍体细胞在基因编辑后的其中一条链的序列。
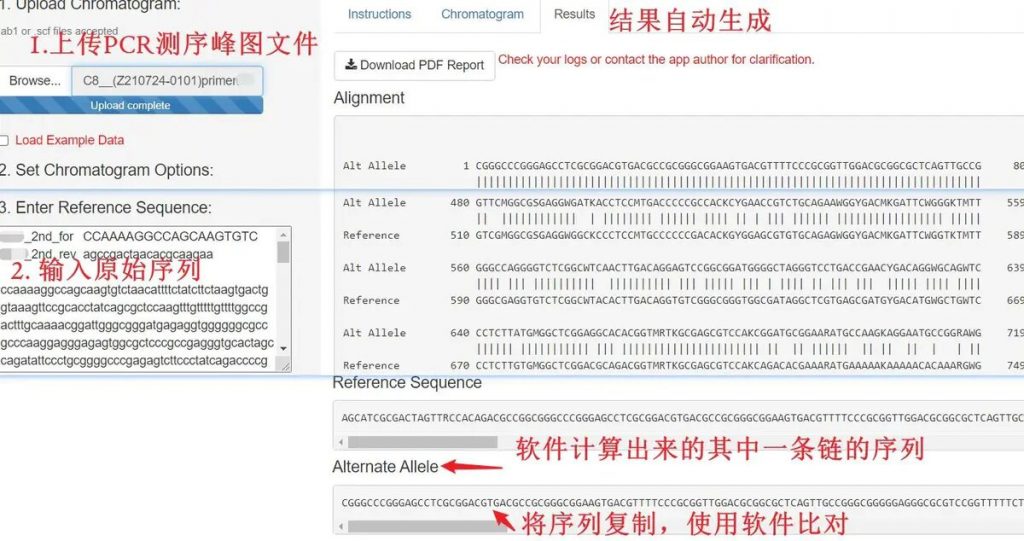
Poly Peak Parser
4. 手动拆峰
工作思路如下:
(1)PCR序列:读取PCR测序结果的上峰图为PCR序列1,将套峰下峰的序列读出为PCR序列2
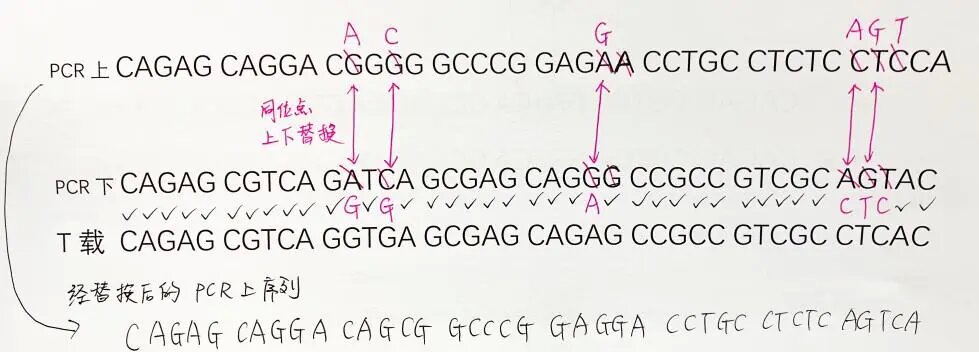
(2)T vector测到的/软件分析出的其中一条链的序列,我们先称之为T序列:摘取T序列和PCR序列1,2进行比对,摘取的位置从gRNA位点前(双峰出现前5-10个碱基)开始,一直到T序列突变位置结束后5-10个序列。通过同位点的上下替换,使得其中一条PCR序列和T序列一致,则剩下的PCR序列则为另外一条链的序列结果。
用文字描述非常难以理解,我们用过实际样本进行进行推测吧。
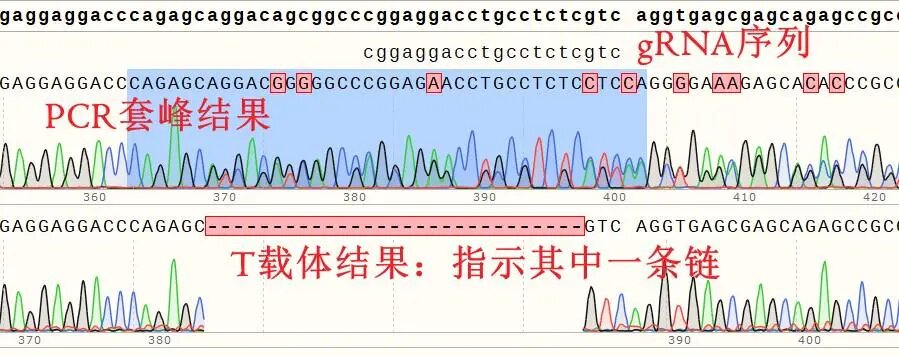
将PCR上峰和下峰序列读出
我们在双峰出现位点前几个碱基开始读取序列(图中蓝色选取部分)如下:
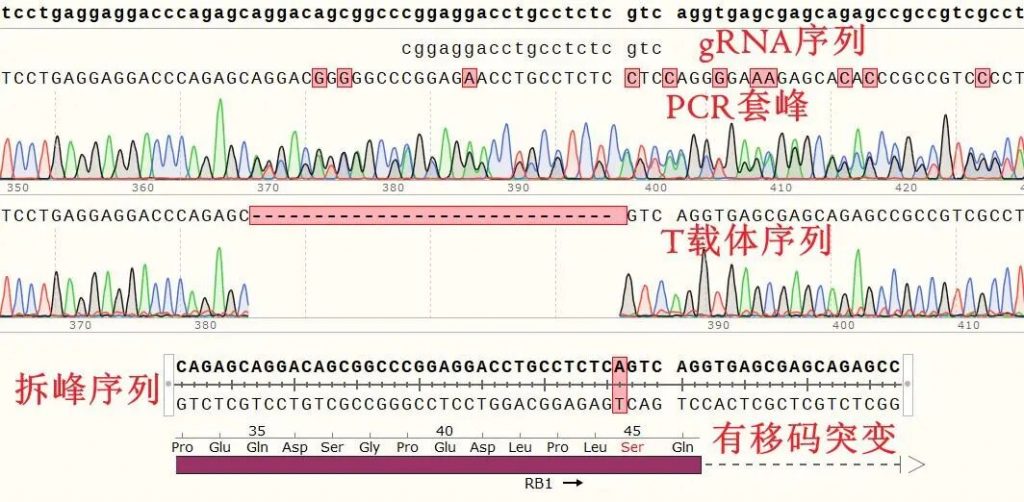
将T载体序列和PCR上峰,PCR下峰序列抄下来比对。通过同位点碱基替换使得PCR下峰序列和T在序列一致,经替换后的PCR上峰序列,则为另外一条链的结果。将该序列保存为DNA序列,加入到软件中比对可看到,另外一条链的基因编辑结果为一个A碱基的插入。结合两条链的结果,都没有出现3的倍数的碱基缺失或插入,初步认定该细胞为成功的敲除细胞系。
final result
以上拆峰适用于二倍体的单细胞克隆细胞系。如果发现拆峰结果不匹配或者拆峰无法顺利进行(上下没有可替换的碱基),则说明该细胞可能为多倍体或者多克隆细胞系。
链接:https://www.jianshu.com/p/4be28c7c61ca
来源:简书