前言
提起“克隆”、“载体构建”这两个词,似乎总会同时提到“限制性内切酶”。没错,在过去数十年,用限制性内切酶产生黏性末端,并通过碱基互补配对,连接两个甚至更多片段的克隆方法,是载体构建的经典方案。
近年来,“无缝克隆”逐渐受到科学家的欢迎。相比之下,无缝克隆操作更加简单,灵活性更强,同时几乎不受序列的限制,一次可定向组装高达10个片段的dsDNA。这篇文章将带大家认识无缝克隆的原理、优势和应用。

一、无缝克隆的原理
首先要强调的是,无缝克隆有两个主要的流派:Gibson assembly和Golden Gate assembly。由于Golden Gate assembly依赖于Type IIS限制性DNA内切酶(如BsaI,BbsI等),一旦载体不携带相应的识别序列,这种方法就走不通了。而假设通过PCR引入Type IIS内切酶识别序列,那便不是真正意义上的“无缝”了。因此本文所提的“无缝克隆”,特指Gibson assembly流,不涉及任何Golden Gate及其他流派。
目前,无缝克隆比较著名的商品化名字有NEB家的Gibson Assembly和NEBuilder HiFi DNA Assembly,Clontech家的In-Fusion,以及Invitrogen家的GeneArt等。但无论叫什么名字,其基本原理都和早期的Gibson assembly的是一样的。
与传统的双酶切克隆一般,无缝克隆同样需要依赖于dsDNA的黏性末端进行互补配对,并利用DNA连接酶催化形成磷酸二酯键修复缺口(nick)。但是,无缝克隆并不使用“内切酶”来制造黏性末端,而是通过“外切酶”来产生的。
(Tips 1:“内切酶”是在核酸链内部进行“一刀两断”剪切的酶,而“外切酶”是在核酸链的末端、即外部进行“逐个碱基”剪切的酶)
在无缝克隆中使用的外切酶是T5核酸外切酶,它能沿着5’→3’方向,降解dsDNA,从而产生黏性末端。
(Tips 2:T5外切酶同时具有ssDNA外切酶活性,但并不降解超螺旋dsDNA。)
读到这里大家应该能想到,假设要拼接的两个片段的末端,是一模一样的碱基序列,那么通过T5外切酶,就能产生几乎一致的黏性末端了。没错,无缝克隆的原理便是如此。如下图所示,假如我们需要将3个片段按照1→2→3的顺序拼接起来,只需要保证1的末尾和2的开头,以及2的末尾和3的开头,具有同样的序列即可(我们可称之为同源臂)。这个同源臂序列,并不是额外添加进去的,而是载体或者拼接片段本身携带的。也就是说,我们可以通过PCR的方法,在外源拼接片段的两端加上线性化载体的序列即可。
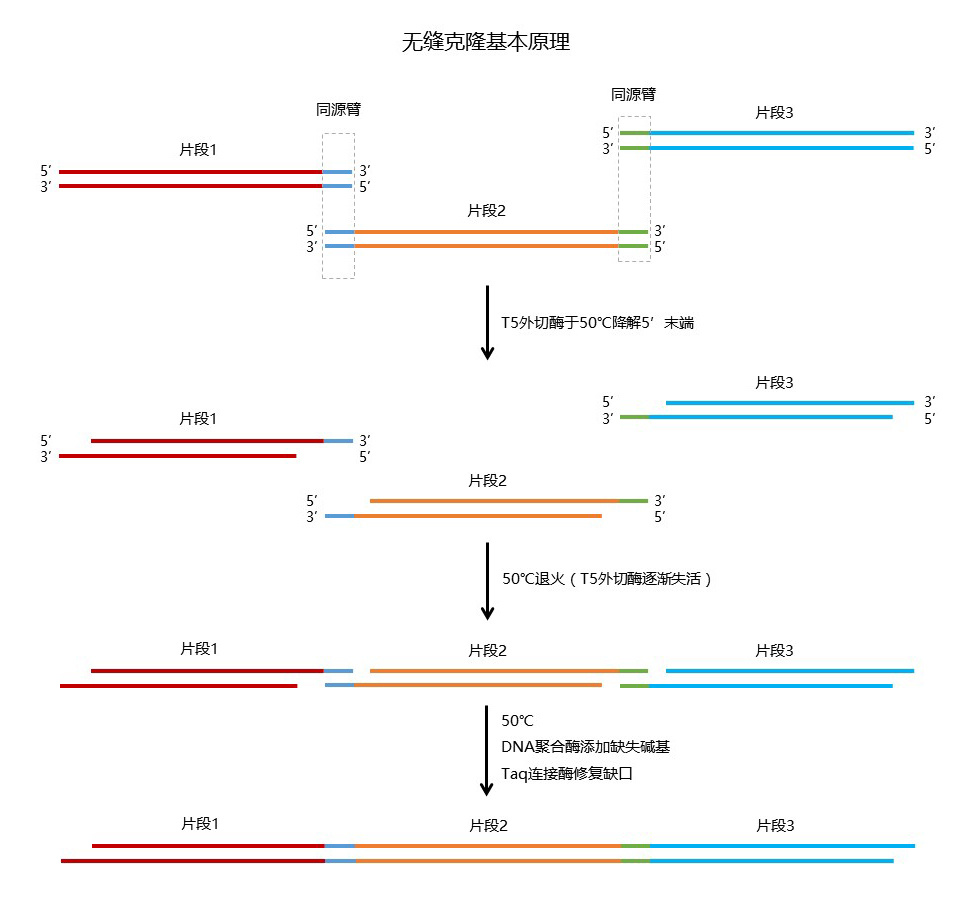
但是,T5外切酶并不能保证将每一个片段的5’末端都降解成一样长的黏性末端,退火之后,极可能存在缺失的碱基。这时候,就需要借助DNA聚合酶,也就是我们平时用于PCR扩增的酶进行修复。最后一步,便是借助Taq DNA连接酶,催化形成磷酸二酯键,将所有缺口补齐。
有心的读者会发现,假设T5外切酶一直降解dsDNA的5’末端,岂不是最终会将其变成ssDNA?这样如何进行下游的拼接?难道仅仅依靠DNA聚合酶的5’→3’合成双链的反应与之竞争?
无缝克隆的另一高明之处,就是利用高温。T5外切酶的最适温度是37℃,然而无缝克隆是在50℃下进行的,加之添加的酶单位较少,因此整个反应体系很快便会失去外切的活性,而DNA聚合酶的活性得以维持。而为了保证DNA连接反应顺利进行,无缝克隆采用的是热稳定的Taq DNA连接酶,而非传统的T4 DNA连接酶。同时,为了保证黏性末端能够在50℃下进行互补配对,同源臂的长度一般>15 bp。
二、无缝克隆的优势
不受片段序列的限制、实现任意组装,是无缝克隆的最大优势。
传统的酶切克隆,需要克隆载体的多克隆位点携带可用的单一酶切位点。假如这些酶切位点在外源片段之中同时存在,克隆就会受到限制。而无缝克隆不依赖于限制性内切酶,仅靠同源臂进行互补配对。因此每次进行片段拼接时,仅需通过引物制作同源臂,而不需要像以往一般仔细地检查PCR出来的外源片段内部是否同时会被内切酶消化。与此同时,我们也不再需要购买各式各样的内切酶,只要一个试剂盒就可解决所有克隆实验。
无缝克隆第二个优点是可以同时无缝地拼接多个片段。传统的酶切克隆,由于酶切位点有限,我们能组装的片段数量是有限的。同时,由于酶切位点之间存在着一定的空间距离,片段与片段之间不可避免地会产生间隔,或者说“缝隙”。此外,酶切克隆通常一次只能拼接2-3个片段。随着拼接片段的增多,传统方案的效率会急剧降低,通常只能分多次拼接。
(Tips 3:过去有一种提高酶切连接多片段的方法,是在连接后,在载体和外源片段的边界上设计引物,进行一轮PCR,扩出全长连接产物,跑胶纯化后再进行二次连接。然而这种方法耗时耗力,且长片段的PCR容易突变或失败,同时PCR的引物还引入了更多的无意义序列。)
而无缝克隆可轻松拼接4-5个片段,最高可达10个,而且只需要将所有片段混在一个管子里,进行一步反应。
节省时间,是无缝克隆的第三个优势。传统的酶切克隆,首先需要进行PCR,胶回收,然后酶切载体和外源片段,再过柱纯化,随后连接,最后转化。而如果需要进行多片段组装,部分步骤还需重复几轮。每多一个片段,可能要多花费三天到一周的实验时间。而无缝克隆仅需PCR和胶回收,随后即可直接进行多片段拼接。拼接10个片段所花的时间,跟拼接2个片段的几乎是一样短的。
常见问题答疑:(视频转自碧云天的公众号)
三、应用举例
例一:无缝克隆在CRISPR中的应用。
CRISPR介导的基因敲除依赖于Cas9等内切酶,以及帮助内切酶定位到指定位点的gRNA。在一个细胞系中同时敲除多个基因,虽可以通过转染多个gRNA表达载体来实现,但这种方案存在随机性,并不能保证每个细胞都能获得所有的gRNA载体。而通过无缝克隆,我们可将多个U6启动子和gRNA序列(包括spacer和scaffold)元件串联起来,实现一个质粒表达多个gRNA。
(Tips 4:目前还有其他多种multiplex CRISPR的方案,各有优缺点。请自行查阅文献,本文不赘述。)
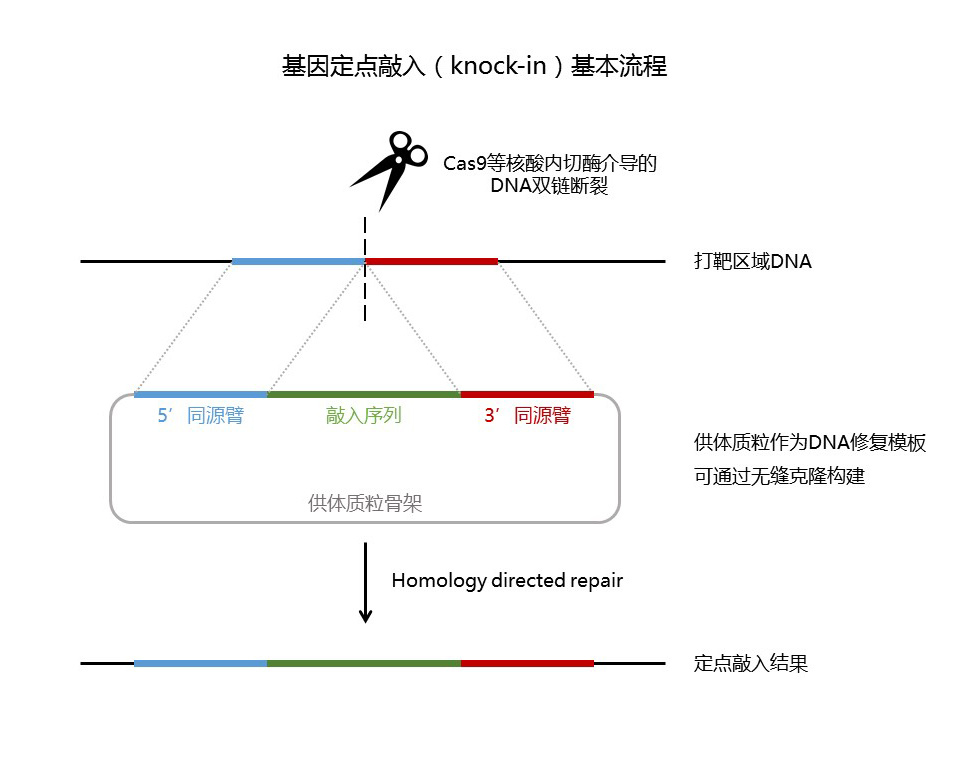
基因定点敲入(knock-in),需要在打靶区域DNA产生双链断裂,并以含有上下游两段同源臂外加插入基因序列的供体作为修复模板,实现外源序列的插入。简而言之,供体质粒需要含有5’同源臂 + 插入序列 + 3’同源臂,以及基本质粒骨架(含有ori、抗性基因等基本元件,方便生产供体质粒)共计4个部分构成(见下图)。通过无缝克隆,我们可以轻松地将这4部分片段通过一步反应拼接出来。
例二:用无缝克隆构建多顺反子表达载体。
将EGFRvIII基因、P2A多顺反子元件、iRFP720基因拼接到pLVX-TetOne载体中(如下图),实现Tet-on诱导表达EGFRvIII基因,同时还能表达iRFP720基因,用于小动物活体荧光成像。
(Tips 5:有关多顺反子元件的介绍,请阅读本专栏的另一篇文章 多顺反子表达元件——一次满足你N个愿望)
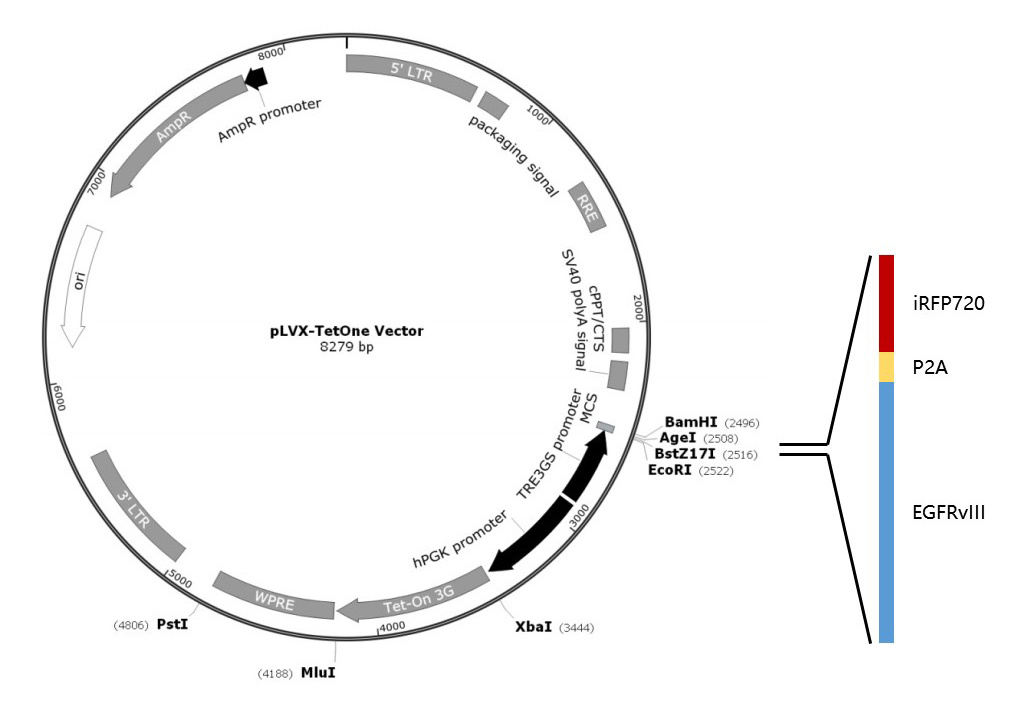
假如使用双酶切克隆,你会发现,只有AgeI和BstZ17I酶切位点可用于EGFRvIII基因克隆,因为另外两个酶切位点同时存在于该基因内部。而P2A元件和iRFP720基因必须和EGFRvIII基因处于同一个读码框,我们没有办法使用AgeI进行后续的克隆了。
本司机的做法是,直接用PCR构建出各个部件,然后通过无缝克隆一步组装。这几个部件分别是:
1、pLVX-TetOne载体的一半;
2、pLVX-TetOne载体的另一半;
3、EGFRvIII;
4、P2A + iRFP720。
将pLVX-TetOne通过PCR拆成两半的原因是:质粒超过8 kb,直接通过PCR线性化整个质粒的突变几率,比扩增两个4 kb的片段要略高一些。同时,由于两个4 kb片段在跑琼脂糖时,可以和8 kb的环状PCR模板区分开来,因此可以直接切胶回收,而不需预先进行DpnI去除环状质粒,即可实现零背景的转化。
(Tips 6:线性化质粒也可以通过单酶切或双酶切来实现。)
P2A元件非常短,仅有66 bp,很可能被T5外切酶彻底降解。不过,我们只需通过两轮PCR,在扩增iRFP720基因的上游引物的5’端引入部分P2A序列(每次引入33个碱基),即可获得P2A + iRFP部件。
(Tips 7:有关无缝克隆的引物设计,可直接使用所购试剂盒厂家的线上工具。我个人比较喜欢用NEBuilder HiFi DNA Assembly,传送门 NEBuilder Assembly Tool。使用之前,可以用Word等文本编辑软件,将所要拼接的序列按5’→3’依次排好,用不同颜色区分标注,然后再挨个粘贴到线上设计工具中即可。此外,该试剂盒似乎通过调整酶浓度和buffer的离子浓度,克服了T5外切酶对ssDNA的活性,从而可以用ssDNA oligo桥接任何dsDNA片段。具体内容请参考NEB公司的宣传页面。)
四、目前存在的问题
吹了半天,无缝克隆其实也不是完美的。细心的读者可能已经发现,前面提到,P2A太短无法直接拼接。是的,无缝克隆目前无法组装小片段,因其可能会被T5外切酶彻底降解。Addgene blog指出,用于Gibson assembly的片段最好不低于200 bp,而NEB官方的线上设计软件,强制输入片段的长度必须大于75 bp,并提示最好不低于110 bp。
(注:NEBuilder试剂盒号称可用短至60 nt的单链oligo桥接两个dsDNA,或者在桥接的同时在两者之间引入短片段,而其他试剂盒做不到,具体机制正如前文所提的T5外切酶对ssDNA的活性。但本人并未亲测是否真实可靠。)
其次,由于无缝克隆依然依赖于前后两条同源臂黏性末端的互补配对,假设黏性末端内部恰好形成了稳定的二级结构(如发夹结构),无缝克隆的效率便会显著降低。虽然这只是个概率的问题,但万一实验失败,在洗完脸后,不妨检查一下同源臂及临近序列会不会产生稳定的二级结构。(注意:反应温度是50℃,也不算低了,通常只有脸太脏的时候才会这么倒霉。)
最后,无缝克隆中所使用的连接酶,是有所谓的“保真性”的。DNA连接酶所实现的功能,是催化形成磷酸二酯键,将两个脱氧核苷酸连接起来。这同时也意味着,我们并不希望见到,含有错配互补序列的片段,也被连接起来。经典酶切克隆法使用的是保真度低、即对错配容忍度极高的T4 DNA连接酶。但由于酶切克隆所产生的黏性末端是完全一致的,因此基本不可能出现问题。而无缝克隆虽然借助了序列完全一致的同源臂,但T5外切酶并不能保证仅消化同源臂的部分。再加上在互补配对时,长链黏性末端极易随机产生不完美的配对,因此导致拼接错误。DNA连接酶的保真度,是由酶的类型,以及反应条件(温度、缓冲液pH、盐强度等)决定的。不同无缝克隆试剂盒的厂家,可能有不同的配方,至于哪个最适合你,就只能靠自己摸索了。而至于DNA聚合酶的保真性,由于需要合成的碱基数少,并非传统的PCR应用,因此一般不会在这个环节翻车。
附上进哥做的基于碧云天教程的分子量计算工具:
你好,视频无法观看
你好 已经更新视频 该视频源于碧云天公众号