蛋白质的糖基化(glycosylation)是目前已知最为复杂的蛋白质翻译后修饰,由8种单糖参与构成的各种复杂寡糖链与蛋白质连接形成。蛋白质糖基化分析包括:蛋白质是否发生了糖基化及糖基连接形式、糖基化位点分析和糖链结构分析。
蛋白质糖基化分析的方法主要有两种:内切糖苷酶分析N-连接寡糖和糖基化抑制剂。
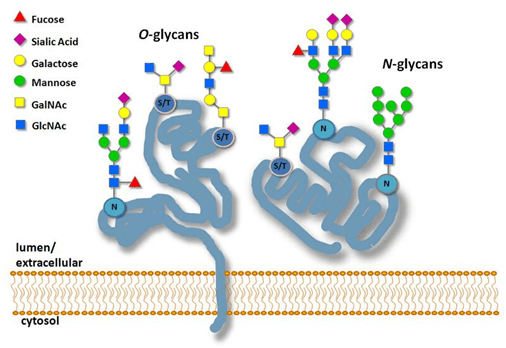
蛋白质糖基化的基本原理是:寡糖中的N-乙酰葡糖胺与多肽链中天冬酰胺残基的酰胺氮连接,形成N-连接糖蛋白;寡糖中的N-乙酰半乳糖胺与多肽链的丝氨酸或苏氨酸残基的羟基相连则形成O-连接糖蛋白。糖蛋白可分布于细胞表面、细胞内分泌颗粒和细胞核内,也可被分泌到细胞外构成细胞外基质。真核细胞内大约50%的蛋白质属于糖蛋白(glycoprotein)蛋白质糖基化所形成的糖链一般含有1015个单糖基,理论上计算会产生大抵的异构体。
- 内切糖苷酶分析N-连接寡糖
简介
如果蛋白质分子中有糖链存在,在体外使用可去除蛋白质中的N-连接寡糖的内切糖苷酶(endoglycosidase)、糖胺酶(glycosamidase)或者可去除O-连接寡糖的O-糖苷酶O-glycosidase)处理样品,然后和未经处理的样品共同进行SDS-PAGE,不含糖链的蛋白质的电泳迁移率会高于糖基化蛋白。因此,对这些酶的敏感性可用于检测N-连接寡糖或O-连接寡糖的存在。
原理
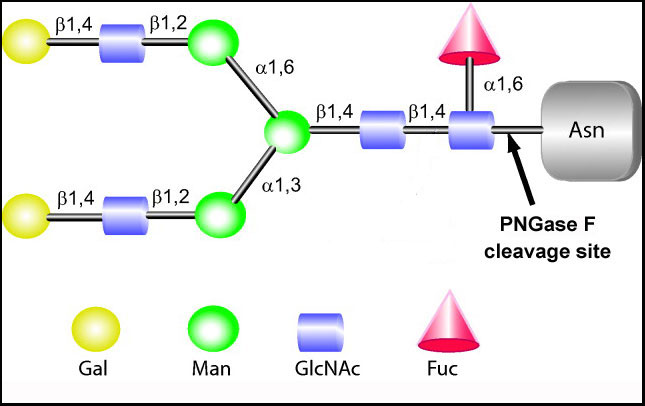
内切糖苷酶分析N-连接寡糖的基本原理是:N-糖苷酶和肽N-糖苷酶(Glycopeptidase F,GPase F)可以将糖链从天然或变性蛋白质的肽链骨架上完整地切割下来,去除所有的N-连接寡糖链。切割作用发生在最靠近天冬酰胺的糖与氨基酸的连接处。
试剂:
1、蛋白质A-琼脂糖珠
2、0.1mol/L的2-ME/0.1%SDS
3、0.5mol/L Tris-HCl(pH8.6)
4、10%NP-40
5、200-250mU/ml PNGase F
6、0.5mol/L Tris-HCl
步骤
内切糖苷酶分析N-连接寡糖的基本过程可分为如下几步:
A、[35S]-甲硫氨酸示踪标记细胞,裂解细胞后,用特异性抗体沉淀拟检测糖基化的目标蛋白,用蛋白质A-琼脂糖珠收集抗原抗体复合物。
B、在微量离心管中,向结合着抗原抗体复合物的琼脂糖珠中加入20-30ul 0.1mol/L的2-ME/0.1%SDS,充分混匀,90°C 3~5min,冷却后于1000g离心1min,将含有变性蛋白质的上清移至洁净的微量离心管内。
C、取10ul变性蛋白质上清,依次加入3微升0.5mol/L Tris-HCl(pH8.6)、5ul H2O、2ul 10%NP-40、5ul 200-250mU/ml PNGaseF。每加1种试剂后要混匀。对照管内以5ul 0.5mol/L Tris-HCl代替酶。
PS:建议摸索浓度,我的经验使用终浓度≥500 U/ml的PNGase F,并延长温育时间至12-16小时以确保完全去糖基化。此外本步骤缓冲液根据说明书选择,可以50 mM磷酸钠缓冲液(pH 7.5)或HEPES缓冲液(pH 7.0-8.0)等.
D、37℃温育过夜。
E、加入2.5ul10X SDS样品缓冲液,90℃5min,使PNGaseF失活。
F、SDS-PAGE分离蛋白质,进行western blot。电泳迁移率的增加表明存在N-连接寡糖链。
注意事项
可以用pH7.0的磷酸钠或HEPES缓冲液代替Tris-HCl,不能使用含钾盐的缓冲液,因其引起SDS沉淀。
2. 糖基化抑制剂用于鉴定糖蛋白
原理
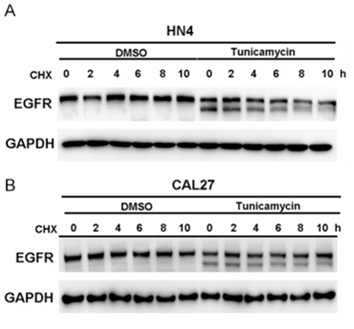
衣霉素是一种N-糖基化抑制剂,它通过阻断N-乙酰葡萄糖胺-1-磷酸转移酶(GPT)的活性,从而抑制N-连接糖基化的第一步,也就是将糖链转移到新生多肽的Asn-X-Ser/Thr序列上。因此,当细胞被衣霉素处理后,目标蛋白的糖基化会被抑制,导致其分子量减少,这在WB结果中应该会显示为条带位置的下移。。
细胞处理与分组
A.细胞培养
选择目标蛋白表达的细胞系(如HEK293、HeLa等),常规培养至对数生长期。
B.衣霉素处理
实验组:添加衣霉素(终浓度1-5 μg/mL,需预实验优化),处理12-48小时。
对照组:等体积DMSO(溶剂对照)处理相同时间。
可选阳性对照:取部分细胞裂解液,用PNGase F(去N-糖基化酶)37℃处理1小时。
C.蛋白样品制备
裂解细胞:RIPA裂解液(含蛋白酶抑制剂)冰上裂解30分钟,离心取上清。
定量:BCA法测定蛋白浓度,调整至等量。
变性:加入5×SDS上样缓冲液,95℃煮沸5分钟。
D.Western Blot检测