rm(list=ls())
options(stringsAsFactors = F)
load("D:/R/R TCGA/survival_input.Rdata")
load("D:/R/RTCGA/TCGA-KIRC-miRNA-example.Rdata")
dim(expr)
dim(meta)
# 可以看到是 537个病人,但是有593个样本,每个样本有 552个miRNA信息。
# 当然,这个数据集可以下载原始测序数据进行重新比对,可以拿到更多的miRNA信息
# 这里需要解析TCGA数据库的ID规律,来判断样本归类问题。
group_list=ifelse(as.numeric(substr(colnames(expr),14,15))< 10,'tumor','normal')
exprSet=na.omit(expr)
## 必须保证生存资料和表达矩阵,两者一致
all(substring(colnames(exprSet),1,12)==phe$ID)
#表达矩阵列名的1到12位等于phe的ID吗
## 挑选感兴趣的基因构建coxph模型
# 2015-TCGA-ccRCC-5-miRNAs-signatures
# Integrated genomic analysis identifies subclasses andprognosis signatures of kidney cancer
# miR-21,miR-143,miR-10b,miR-192,miR-183
#hsa-mir-21,hsa-mir-143,hsa-mir-10b,hsa-mir-192,hsa-mir-183
e=t(exprSet[c('hsa-mir-21','hsa-mir-143','hsa-mir-10b','hsa-mir-192','hsa-mir-183'),])
e=log2(e)
colnames(e)=c('miR21','miR143','miR10b','miR192','miR183')
dat=cbind(phe,e)#合并生存资料和选出来的基因表达情况
dat$gender=factor(dat$gender)
dat$stage=factor(dat$stage)
library(survival)
library(survminer)
colnames(dat)
s=Surv(time, event) ~ gender + stage + age + miR21+miR143+miR10b+miR192+miR183
s=Surv(time, event) ~ miR21+miR143+miR10b+miR192+miR183
model <- coxph(s, data = dat )
summary(model,data=dat)
options(scipen=1)
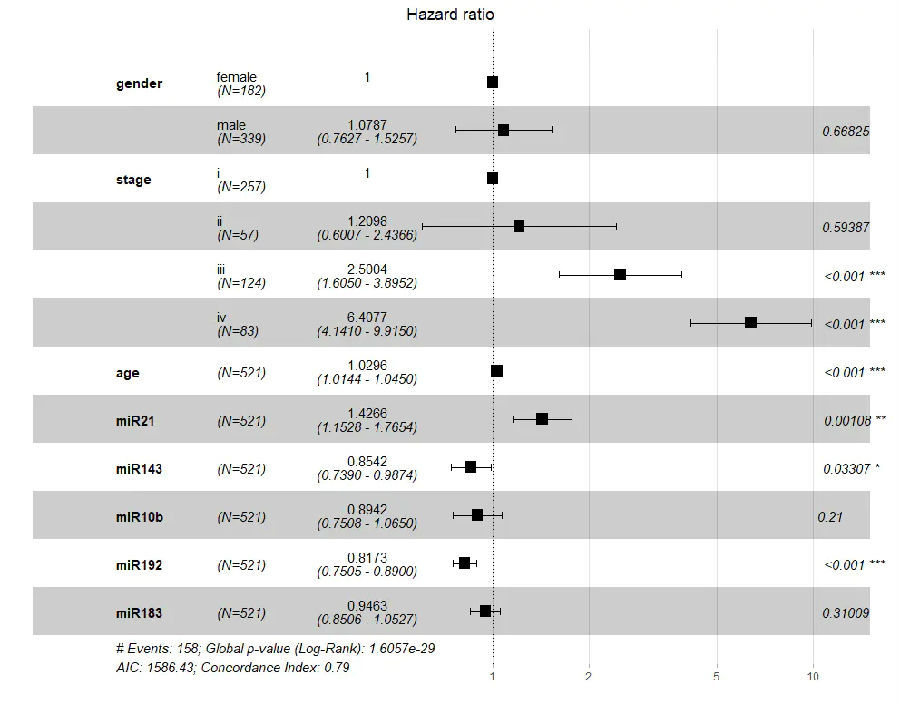
ggforest(model, data =dat,
main = "Hazardratio",
cpositions= c(0.10, 0.22, 0.4),
fontsize =1.0,
refLabel ="1", noDigits = 4)#画森林图
fp <- predict(model)#预测模型
summary(model,data=dat)
library(Hmisc)
options(scipen=200)
with(dat,rcorr.cens(fp,Surv(time, event) ))
#用一部分预测一部分,可以用其本身,也可以用其他的数据,也可以把原来的数据分为两组
# 用于计算生存分析中的COX模型预测值与真实之间的区分度(discrimination),也称为Harrell'sconcordanceindex。
# C-index在0.5-1之间。0.5为完全不一致,说明该模型没有预测作用
# 1为完全一致,说明该模型预测结果与实际完全一致。
# 若要找到最佳模型,我们可以进行变量选择,可以采用逐步回归法进行分析