聚类是基因表达数据分析中的重要工具-转录本以及蛋白质水平。这种无监督的分类技术通常用于揭示隐藏在大型基因表达数据集中的结构。迄今为止应用的绝大多数聚类算法都会对数据进行严格划分,即每个基因或蛋白质都是完全分配给一个聚类的。如果聚类分离良好,则硬聚类是有利的。但是,对于基因表达数据而言,情况通常并非如此,因为基因/蛋白质簇
经常重叠。另外,硬聚类算法通常对噪声非常敏感。为了克服硬聚类的局限性,可以应用软聚类为研究人员提供一些优势。首先,它会生成可访问的内部集群结构,即,它指示相应簇代表基因/蛋白质的程度。这个额外的信息可用于精细搜索监管要素。其次,可以定义群集之间的整体关系,从而定义全局群集结构。此外,软聚类具有更强的抗噪性,并且可以避免对基因进行先验的预过滤。这样可以防止从数据分析中排除生物学上相关的基因/蛋白质。
下面介绍这个包的使用:
采用R包中自带的测试数据。
1.包的安装
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("Mfuzz")
browseVignettes("Mfuzz") #查看帮助文档(软件英文说明)
library(Mfuzz) #加载包
2.数据的前处理
#如果为测试数据,则输入如下代码
data(yeast)
#如果为真实数据,则输入如下代码
peach<-read.table("/Users/bcl/Desktop/foldchange.txt") #参数为地址加文件名。
count<- data.matrix(peach) #将数据框转换为数值矩阵。
peachs<-new("ExpressionSet",exprs =count) #它可用来生成 一个类的对象。第一个参数提供类的名称,字符串或描述类的对象。
2.1缺失值
#第一步:我们排除丢失超过25%的测量值的基因。注意缺失值应在基因表达矩阵中用NA表示。
#测试数据
yeast.r <- filter.NA(yeast, thres=0.25)
#真实数据
peachs.r<-filter.NA(peachs,thres=0.25)
#第二步:像许多其他聚类算法一样,模糊c均值也不允许缺失值,因此,我们用相应基因的平均值表达值替换剩余缺失值。
#测试数据
yeast.f <- fill.NA(yeast.r,mode="mean")
#真实数据
peachs.f<-fill.NA(peachs.r,mode="mean")
2.2过滤
#大多数已发表的聚类分析都包括过滤步骤,以去除表达的基因在低水平或仅表现出小的变化。
#测试数据:
tmp <- filter.std(yeast.f,min.std=0)
#真实数据:
tmp<-filter.std(peachs.f,min.std=0)
2.3标准化
#由于在欧几里得空间中进行聚类,因此基因的表达值为标准化后的平均值为0,标准差为1.
#测试数据
yeast.s <- standardise(yeast.f)
#真实数据
peachs.s <- standardise(peachs.f)
3基因表达数据的聚类
注:mac上要先装XQuartz,否则会报错:网站[https://www.xquartz.org/](https://www.xquartz.org/)
#测试数据
#cl <- mfuzz(yeast.s,c=16,m=1.25) #第一个参数为标准化后的数据,第二个参数代表要聚类的数目,第三个参数为模糊化参数。
#mfuzz.plot(yeast.s,cl=cl,mfrow=c(4,4),time.labels=seq(0,160,10)) #mfrow(4,4)把一个绘图区分成4*4,放16张图,time.labels=seq()表示横坐标从0--160,每格10.
#真实数据
cl<-mfuzz(peachs.s,c=6,m=1.25)
mfuzz.plot2(peachs.s,cl=cl,time.labels = c("2B","S5","S20","S30"),
cex.main=1.5,cex.lab=1.5,cex.axis=1.5,mfrow=c(2,3))
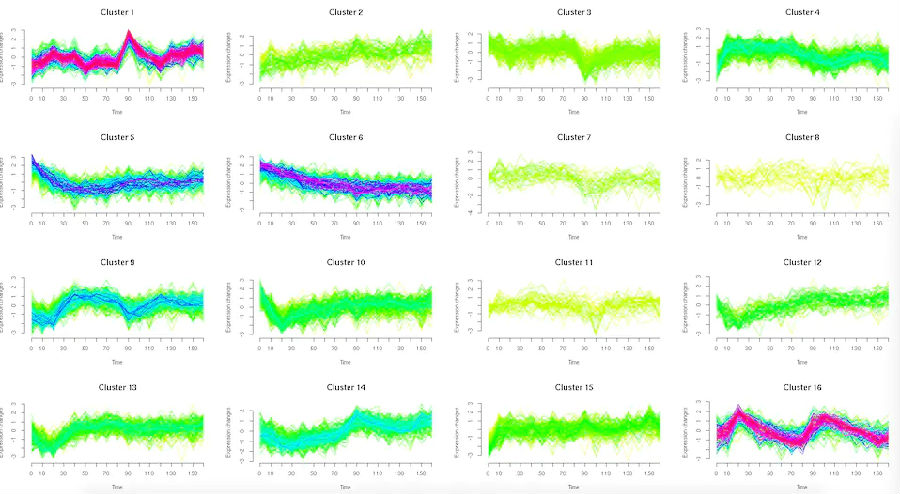
黄色或绿色的线条对应于成员价值较低的基因;红色和紫色线对应具有高隶属价值的基因。
3.1设置FCM聚类的参数
对于模糊c均值聚类,模糊参数m和聚类数c必须预先选择。对于模糊参数m,我们想选择一个值来防止随机数据聚类的值。请注意,模糊聚类可以通过以下方式进行调整,即随机数据不会聚类。这是硬聚类(例如k-means)的明显优势,硬聚类即使在随机数据中也可以检测到聚类。为此,存在不同的选择:可以使用函数partcoef进行测试,是否针对m的特定设置对随机数据进行了聚类。
#下面代码可得到模糊参数m值
#测试数据
m1 <- mestimate(yeast.s)
m1
#实际数据
m1 <- mestimate(peachs.s)
m1
设置聚类数c的最佳数量通常很困难,尤其是对于短时间序列以及聚类重叠的情况。在这里,可以测试c的范围,并且可以设置c的最大值,从而导致出现空聚类。而且,聚类质心之间的最小距离D min可以用作聚类有效性指标。在这里,我们可以测试众多c对应的D min。 我们期望D.min在达到最佳c之后下降的慢一点。另一种方式将用一定范围的簇号进行聚类,然后根据对它们的生物学相关性的评估来选择最佳的聚类,例如通过GO分析。
3.2聚类核心
隶属度值也可以指示向量彼此之间的相似性。如果两个基因表达载体对于特定聚类具有较高的隶属度,则它们通常彼此相似。 这是定义聚类核心的基础。 我们定义成员值大于选定阈值的基因属于聚类的α-core。 这使我们能够定义聚类内基因之间的关系。 与分层聚类类似,聚类的内部结构变得可访问。
设置α=0.7时候,平均聚类内变化会大大降低。因此,使用α—阈值可以充当基因过滤的后验。这与先前讨论的需要先验设置阈值的问题的过程形成对比。要提取属于聚类核心的基因列表,可以使用acore函数。
黄色或绿色线条对应的基因价值较低; 红色和紫色线对应
具有高价值的基因。
3.3提取某个cluster下的基因
# 查看每个cluster中的基因个数 cl$size # 提取某个cluster下的基因 cluster_gene<-as.data.frame(cl$cluster) # 查看基因和cluster之间的membership membership<-cl$membership
3.4Plots a colour bar
#BiocManager::install("marray")
mfuzzColorBar(col="fancy",main="Membership",cex.main=1)
4聚类稳定性
FCM参数m的变化还允许研究聚类的稳定性。 这里,我们将稳定聚类定义为仅随参数m的变化而显示出结构上微小变化的聚类。 稳定的聚类通常比其他聚类紧凑。 与之形成鲜明对比的是,如果m增加,弱聚类内部结构失去或消失了。
#测试数据 cl2 <- mfuzz(yeast.s,c=16,m=1.35) #m增加。 mfuzz.plot(yeast.s,cl=cl2,mfrow=c(4,4),time.labels=seq(0,160,10))
5全局聚类结构
软聚类的一个有趣的特征是聚类之间的重叠或耦合。偶联表明两个簇共有多少个基因。耦合度低的集群总体上表现出明显的差异模式。如果耦合较大,则聚类模式更相似。 因此,耦合为成对的聚类定义相似性度量。
#测试数据 O <- overlap(cl) Ptmp <- overlap.plot(cl,over=O,thres=0.05)
由于定义了聚类之间的关系,因此可以分析通过软聚类获得的全局聚类结构。与分层聚类相似,全局聚类结构可以按聚类数确定的不同分辨率进行检查C。 对于较小的c,仅获得数据中存在的主要聚类。
#测试数据 cl3 <- mfuzz(yeast.s,c=10,m=1.25) mfuzz.plot(yeast.s,cl=cl3,mfrow=c(3,4)) O3 <- overlap(cl3) overlap.plot(cl3,over=O3,P=Ptmp,thres=0.05)
如果c增加,则会出现具有不同模式的子集群,因为它们共享整个表达模式,所以从主集群派生的子集群通常会紧密耦合。最后,软聚类产生空的聚类,以进一步增加c。
cl4 <- mfuzz(yeast.s,c=25,m=1.25) mfuzz.plot(yeast.s,cl=cl4,mfrow=c(5,5)) O4 <- overlap(cl4) overlap.plot(cl4,over=O4,P=Ptmp,thres=0.05)
这个时间序列可以用于TCGA中多个样本对应不同的TNM分期进行聚类吗?