今天我们来介绍一个功能强大的网站Oncomir:http://www.oncomir.org/
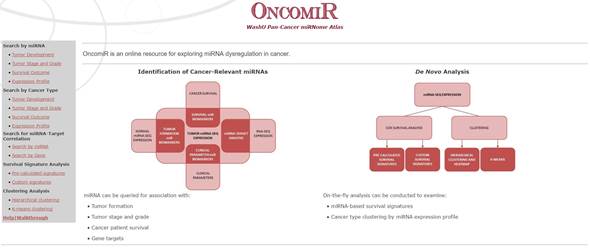
这是我们Oncomir的主页,下面我们来看网站的功能。
在数据库的左侧是不同的功能选项,我们看到首先是:
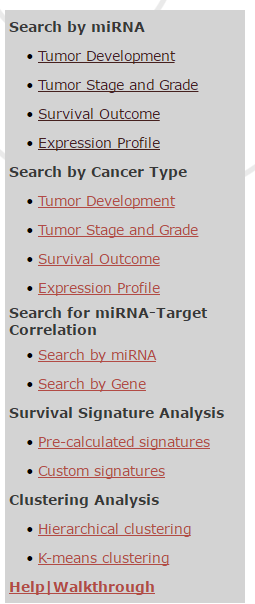
1. Search by miRNA: 根据miRNA进行搜索;
2. Search by Cancer Type:根据肿瘤类型进行搜索;
3. Search for miRNA-targetcorrelation:搜索miRNA和靶基因的相关性;
4. Survival SignatureAnalysis:生存分析;
5. Clustering Analysis:聚类分析。
第一个:根据miRNA进行搜索,
1) TumorDevelopment:查询某条miRNA在肿瘤组和对照组是否表达异常:
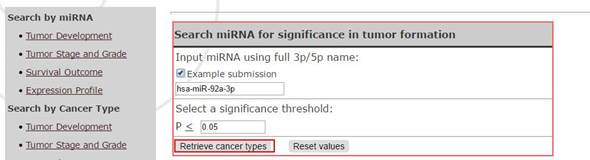
以案例的miRNA hsa-miR-92a-3p为例,单击Retrieve cancer types:

结果显示hsa-miR-92a-3p在14个肿瘤里面显著表达异常。
2)TumorStage and Grade:
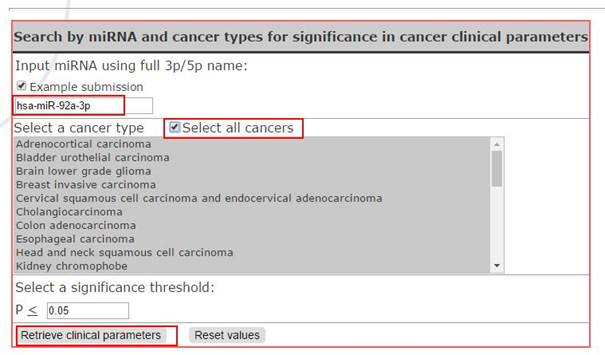
还是hsa-miR-92a-3p,所有肿瘤:
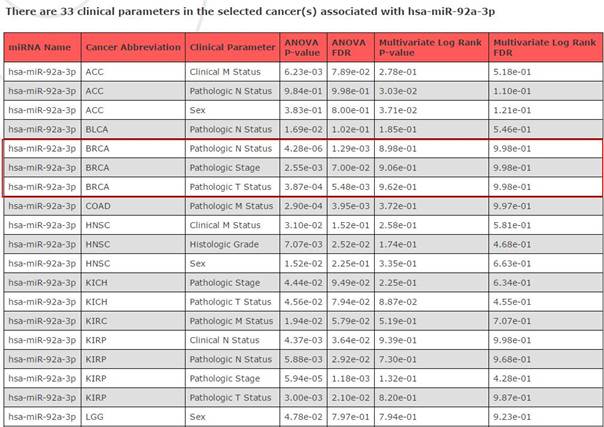
就可以看到miRNA在不同肿瘤里面的情况,比如与BRCA乳腺癌N、T和分级相关。
3) Survival Outcome:生存状态
这是结果:
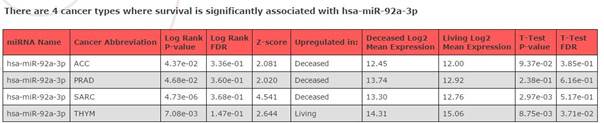
四个肿瘤里面的数据。
4) ExpressionProfile:

我们选择所有肿瘤:
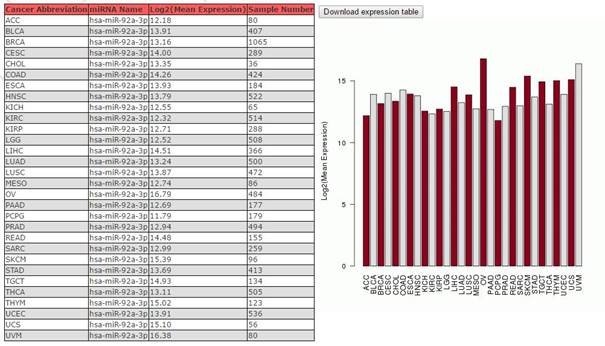
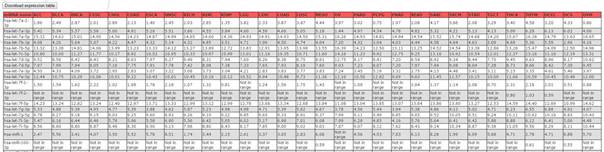
miRNA在所有肿瘤里面的表达数据。
第二个:根据肿瘤类型进行搜索。
1)TumorDevelopment,选择所有肿瘤:
结果:
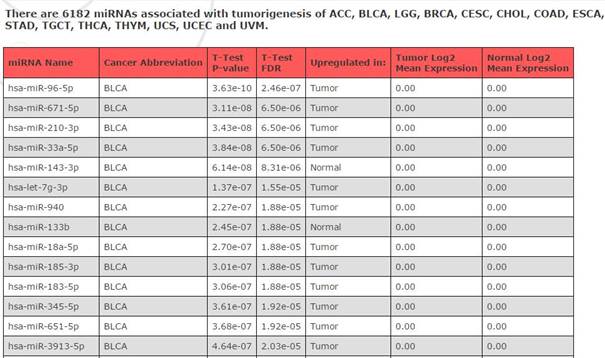
在各个肿瘤里面共有6182条miRNA表达差异(与正常相比),大家在做课题前就可以直接查询选miRNA了。
2)TumorStage and Grade。我们选择所有肿瘤的M分期:
结果:

这样我们是不是就可以找到与转移相关的miRNA了?
3)SurvivalOutcome:
结果:
 3820条miRNA在各肿瘤患者不同生存状态中的显著差异。
3820条miRNA在各肿瘤患者不同生存状态中的显著差异。
4) ExpressionProfile:
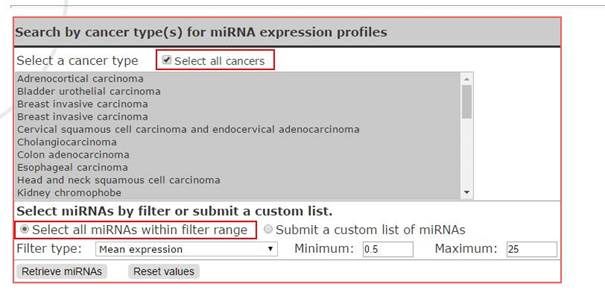
所有的miRNA在所有收录的肿瘤里面表达:
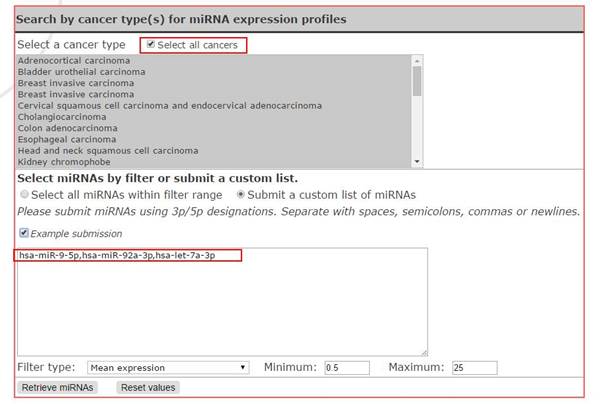
也可以自己提交名单:
结果:

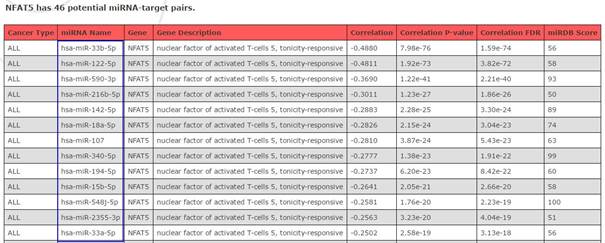
第三个:搜索miRNA和靶基因的相关性。
1) Searchby miRNA:根据miRNA搜索。

结果:

不仅有基因名字,还有靶基因表达与miRNA表达的相关性等数据。
2)Search byGene:根据基因搜索:
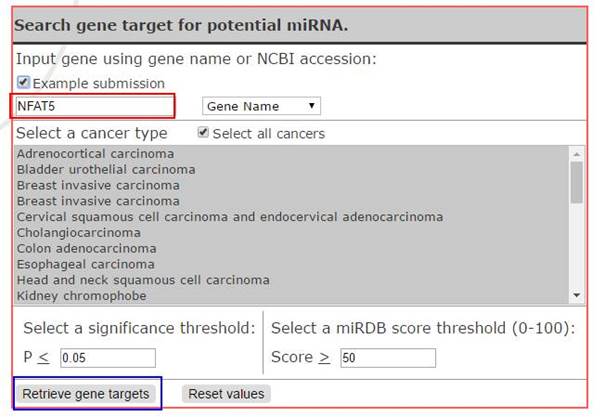
同样也可以实现:
第四个:生存分析。
1)Pre-calculatedsignatures:预计算的指标。

我们选择肺腺癌,经过计算的miRNA表达与生存之间的关系:

大家可以直接看到风险公式里面各miRNA前面的权重数值:
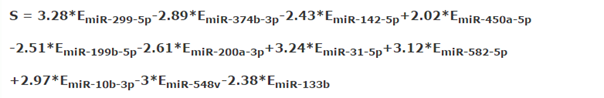
2)Customsignatures:自定义miRNA。
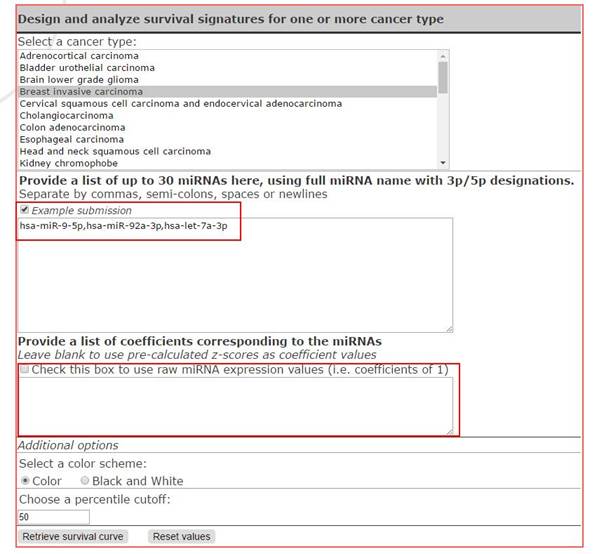
输入我们的miRNA和权重数值,不输入权重数值会直接默认计算好的指标:

这里大家可以直接通过多条miRNA的表达组合来对患者预后进行指征。
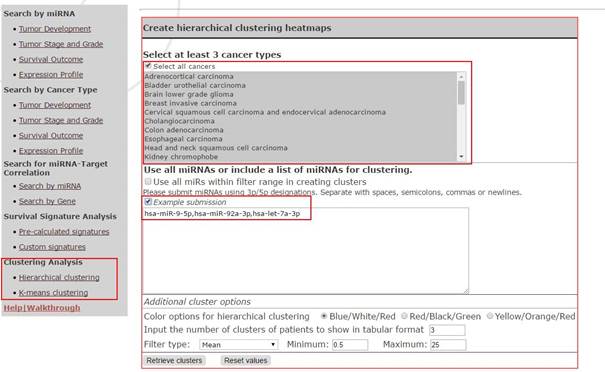
第五个功能:聚类分析。
有了miRNA在各肿瘤的表达后,我们可以对输入的miRNA进行分层聚类:
结果:
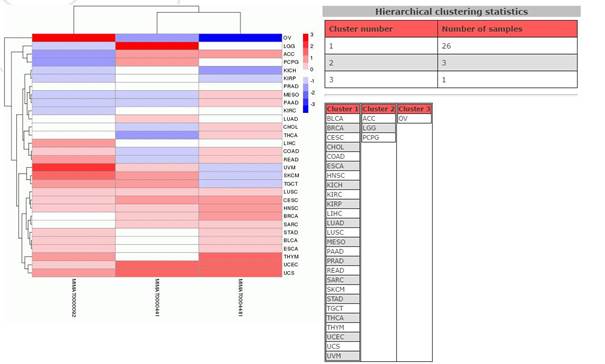
也可以对所有的miRNA进行分层聚类:
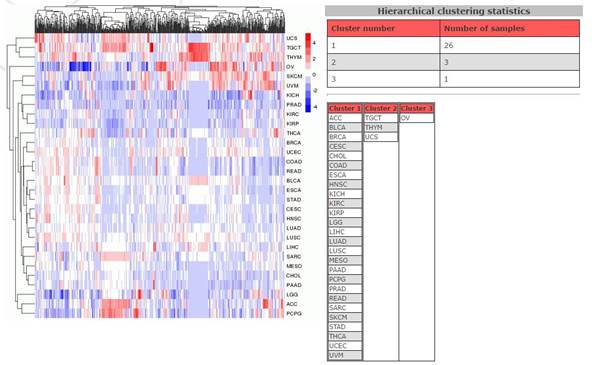
好了,这个网站的功能就介绍到这里了,大家使用的时候推荐chrome浏览器。
另外,吃水不忘挖井人,原文:OncomiR: An onlineresource for exploring pan-cancer microRNAdysregulation.Bioinformatics. 2017 Oct 3. doi:10.1093/bioinformatics/btx627.记得引用。