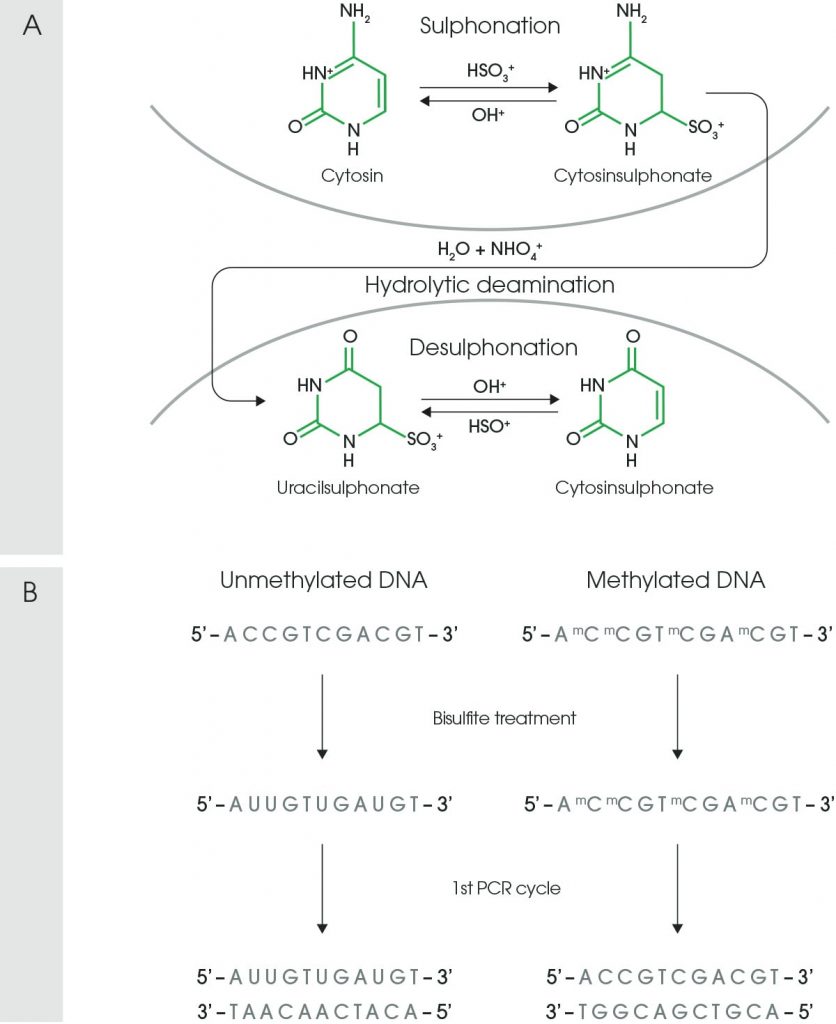
MSP原理:甲基化特异性PCR(MSP)其原理为:双链DNA变性解链后,在HSO3-作用下发生C→U转化,C若已有甲基化则无此改变;甲基化修饰只发生于5′→3′方向C-G相联结构的C上,因此在HSO3-作用后,DNA CpG岛若无甲基化,则序列中的改变为C→U,CG→UG,若有甲基化C不变,CG→CG,用不同的引物做PCR,即可检测出这种差异,从而确定基因有无CpG岛甲基化。从理论上说,DNA发生甲基化的MSP产物的序列与原序列比较,变化为C→T,CG→CG,相应其互补链的改变为G→A,CG→CG。然后根据目的基因修饰前后的改变,相应设计M和U引物,有时我们需要设计两轮引物!
上游3000 UCSC
DNA甲基化是哺乳类动物一个重要的基因外遗传信号.有研究表明,DNA甲基化与大多数肿瘤的发生相关,抑癌基因启动子CpG岛的高甲基化可引起抑癌基因表达沉默,细胞出现无节制生长,导致肿瘤的发生[1].目前,甲基化特异性PCR(methylation-specific PCR, MS-PCR)是研究DNA甲基化最为常用且准确度较高的方法之一,而理想的引物设计则是其成功的关键因素之一.国外互联网上有1个提供免费在线引物设计程序“MethPrimer”,它能够预测CpG岛,根据实验者要求设计硫化测序PCR(bisulfate-sequencing PCR, BSP)和甲基化特异性PCR(methylation-specific PCR,MSP)引物.本研究旨在介绍甲基化特异性PCR引物设计的原则及“MethPrimer”设计程序的使用方法和有关注意事项.
1、材料与方法
1. 1 序列的转换
DNA经亚硫酸氢盐硫化处理后,DNA双链中的“C”转化为“U”,通过随后的PCR,将“U”转化为“T”,但亚硫酸氢盐不能使已发生了甲基化的DNA的“C”发生上述转化,因此,根据经亚硫酸氢盐处理的DNA模板设计引物时,先输入感兴趣的DNA序列,程序将会显示2种序列:一种是输入的源DNA序列;另一种是硫化处理后的DNA序列,除了CpG岛上的5甲基胞嘧啶(5mC)之外,所有非甲基化的“C”都转换成了“T”,根据转化后的序列设计引物,进行BSP和MSP.
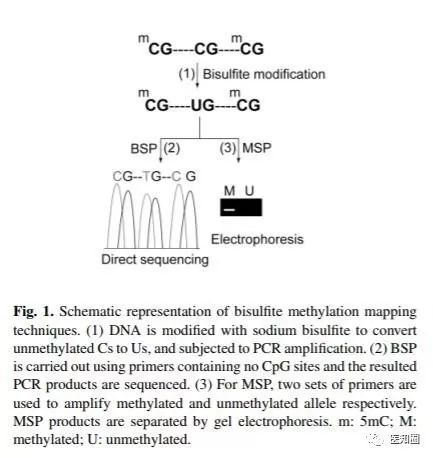
1. 2 CpG岛的预测[2]
哺乳类动物基因组中5% ~10%是CpG(二核苷酸),其中70% ~80%呈甲基化状态,称为甲基化的CpG(mCpG).CpG的聚集称CpG岛,已知在所有管家基因和一些组织特异基因的5′端调控区均有CpG岛呈高度甲基化,基因组中平均100kb有1个跨度为0. 5~5 kb的CpG岛.
“MethPrimer”设计程序默认的CpG岛跨度至少为200 bp,GC含量>50%,CpG出现频率>0. 6.因此,符合这些参数的区域都默认为CpG岛,我们根据任意1个CpG岛设计引物进行扩增.
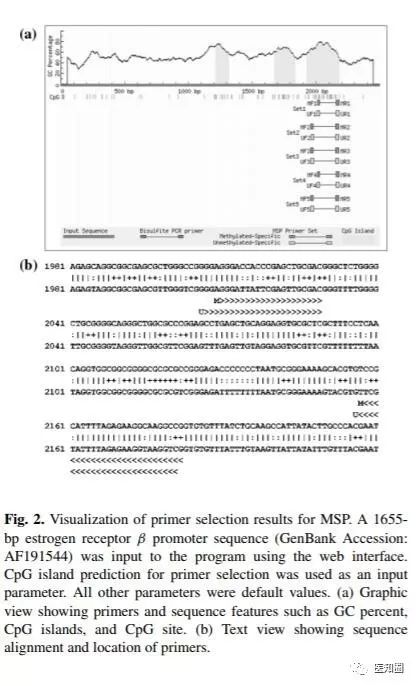
1. 3 BSP的引物设计原则
标准的PCR引物设计原则同样适用于硫化测序PCR(bisulfite sequencing PCR,BSP),除了标准PCR的一些参数外,“MethPrimer”应用3′端算法计算自身退火温度、末端退火温度、碱基对互补率、GC含量、解链温度(Tm),而且还提供了上游引物和下游引物的Tm区别,以及引物中最大允许的单核苷酸重复率.因为所有非甲基化的“C”都被转换成“T”,所以“MethPrimer”中默认的重复“T”为8个,而其他碱基重复数为5个.
DNA的完全硫化是很重要的,因此应选择含尽可能多的非甲基化CpG的“C”区域作为源序列,设计引物.对于BSP,引物设计的原则[1]:①为了区别甲基化DNA和非甲基化DNA,引物不应含有CpG位点;②引物扩增的产物应包含尽可能多的CpG位点.
1. 4 MSP的引物设计原则[3,4]
对于MSP需要设计2对引物,一对是针对于经亚硫酸氢盐处理的甲基化的DNA;另一对是针对于经亚硫酸氢盐处理的非甲基化的DNA.根据甲基化的DNA为模板的PCR扩增甲基化的DNA;根据非甲基化的DNA为模板的PCR扩增非甲基化的DNA.MSP引物设计的原则:①为了最大限度的区分甲基化与非甲基化,引物的3′端至少包含1个CpG位点,我们可以自己设定CpG的“C”距3′末端的最远距离.“MethPrimer”中默认值为3,即是在引物的最后3个碱基中,至少有1个是CpG的“C”.②引物序列中应包含尽可能多的CpG位点.③甲基化引物和非甲基化引物序列3′端应处于相同的CpG位点.如果,2对引物不在相同的CpG位点退火, PCR结果就不能准确反映样本DNA甲基化的情况.但是甲基化引物和非甲基化引物可跨越不同的长度,在起始位点和长度上也可以不同.一般非甲基化引物比甲基化引物长,因为受Tm值限制,由于非甲基化引物中的“C”转化成“T”,导致GC含量降低,从而引起Tm降低.④2套引物应有相近的Tm值,“MethPrimer”中默认2套引物Tm值相差不超过5℃,这种限制可使2个PCR反应在同一PCR仪中进行.
引物的成功设计对于PCR是至关重要的,硫化反应促使DNA链中的“C”转化为“U”,导致GC含量降低,使PCR反应后在序列中出现长的连续“T”,容易引起DNA链的断裂[5, 6].此外,纯化过程中的DNA的丢失,也是要考虑的另一个因素,这些因素又会影响下面的PCR的进行,这些都是设计PCR引物时需要考虑的.
一般情况下, PCR产物的长度大于300 bp时,就很难以硫化DNA为模板进行扩增.因此,“MethPrimer”程序设计PCR产物的长度在100~300 bp, 200 bp的长度比较合适.与标准PCR不同的是甲基化PCR引物的长度,甲基化PCR一般需要更长的引物,引物长度在30 bp左右,原因是硫化降低了模板DNA的GC含量,使序列中出现长的连续“T”,导致很难选择具有合适的Tm值及稳定的引物;另一方面,为了区别硫化处理和没有硫化处理以及未完全处理的DNA,需要引物有足够数量的“C”,这些都增加了选择稳定引物的困难.因此,为了获得更稳定的双链,选择更长的引物是有必要的,因为DNA的Tm值取决于引物的长度[7].一般情况下,甲基化PCR引物的长度在20~30 bp.“MethPrimer”默认的引物长度在20~30 bp, 25 bp为最适长度.
“MethPrimer”在设计引物时,不考虑输入序列中的重复元件和载体序列.因为,对于DNA甲基化的研究,我们一般注重有特点的序列或者序列中确定的区域,大部分这些序列或区域是启动子序列,因此载体序列不考虑;另一方面,输入的序列由程序模拟硫化处理发生了转化,这样重复的元件不再重复了,即使是引物位置所在的序列在转化前是重复序列,也不会影响引物的质量.
总之,“MethPrimer”是一个特异性的针对硫化PCRs(BSP和MSP)设计引物的程序,其操作简便、快速、准确,对于研究DNA甲基化是一个至关重要的工具.目前,“MethPrimer”正在进一步地发展和完善,使它可以设计出更多其他的硫化PCRs的引物.
进哥哥,能推荐查找甲基化基因的网站有哪些吗?
你好,请问如何验证甲基化PCR引物呢?
您好,请问设计出来的引物如何去验证呢?
老师您好,看了您讲解Msp的视频,有几个疑惑想请教您一下,Methprimer网站那个底下不是有一个参数Shift吗,是什么意思呀,我改成2了之后就显示了2个CpG岛,之前就显示就一个。然后我用那个甲基化的引物和原本的启动子序列比较最后显示未找到结合点。最后,Bsp实验是不是把整个启动子扩出来就行呀,然后送去测序,必须要克隆嘛
请问,我做WGBS实验将gDNA打断后进行连接C都甲基化的接头,然后使用接头设计的引物去PCR扩增,为什么一直扩增失败?
老师,您好,我去这个网站设计了MSP的引物,但是存在多个Tm值,下面有个product size 138,Tm69,和上面引物的Tm值相差很大。product size中的Tm值是什么意思呢?我该选择哪个Tm值去做PCR呢?
请问设计出来GC含量都比较高(约78%),怎么办呢?
老师好您好,“从理论上说,DNA发生甲基化的MSP产物的序列与原序列比较,变化为C→T,CG→CG,相应其互补链的改变为G→A,CG→CG。”这句话应该怎么理解?发生甲基化的C不是不会改变吗?
您好,以上是只能做5mC的引物设计么?请问是否有DNA 6mA甲基化的引物设计的方案呢?
进哥,引物设计出甲基化序列中只有一个CG,若想至少有3个CG,怎么设计?
那这个没有办法,是因为CG位点太少是吧,只有一个的话就是可能不能完全区分甲基化和非甲基化,试试看吧
你好,请问怎么查找CpG甲基化的检测靶点呢
您好 请问具体什么意思?是还不确定基因 还是确定基因不确定位点
对,就是确定基因,但是不确定甲基化位点,这种情况下您觉得是不是应该亚硫酸盐修饰后测序挑选位点才比较好?
是的,如果需要定位到一个点,需要bsp seq,不是的话,msp就够了
1.2CpG岛的预测中,“基因组中平均100个bp有1个跨度为0. 5~5 kb的CpG岛”,是否应该为基因组中平均100个Kb有1个跨度为0. 5~5 kb的CpG岛,否则基因组中全是CpG岛了
是的,我改一下。人类基因组CpG岛约为28890个,大部分染色体每1Mb就有5-15个CpG岛,平均值为每Mb含10.5个CpG岛:1Mb=1000kb有10个,也就是100kb有1个
您好,我也在MethPrimer上设计了某基因启动子区的CpG岛的引物,但是扩增效果非常差,可以加微信联系您吗?
你好!甲基化的C不是和羰基的C相对吗?图示是否有误?
您好,我设计引物时也出现了上游2000bp并没有发现CPG岛,然后我直接通过NCBI找到了CPG岛,但无法设计出引物,可否与您进行微信沟通?谢谢,我的微信:L_2317351
可以,不知道是否解决 如还需要讨论加我微信吧:18021308280
您好,我按照此法对启动子进行测序但无结果,请问原因会是什么呢,可不可以加微信联系您
可以的 当然
您好,VX:16628731513
您好,我在MethPrimer上设计了某基因启动子区的甲基化位点,但没有显示CPG岛,然后我调了预测条件参数(CG%),从50%调到了40%时有CPG到显示,但用对应的引物去扩增还是没有扩增出目的片段。是什么原因呢?
刚刚看到你的消息 请问是甲基化和非甲基化的都没有P出来吗?原因多种可能 要不加我微信讨论?
您好,这是我的VX:15154911223
好的,已查阅提供的材料,非常感谢!
你好,受益匪浅,请问如果想将荧光定量PCR和MSP结合的话,该怎么考虑内参的设计呢?
您好,实际上我也没做过这个,即所谓qMSP or ms-qPCR,我查了一下资料,bioprotocol上有一篇教程:Quantitative Methylation Specific PCR (qMSP) —BIO-PROTOCOL
此外,epigentek也有一款商业化产品