一、RIP(RNA immunopreciptation)
1.原理
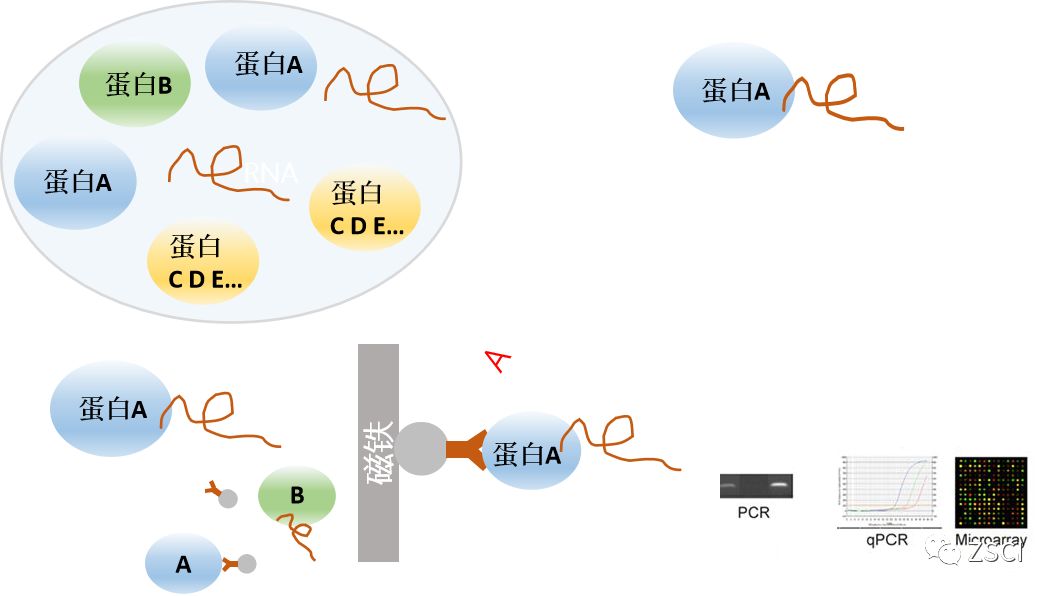
①RIP技术(RNA Immunoprecipitation,RNA免疫沉淀),是研究细胞内RNA与蛋白结合情况的技术,是了解转录后调控网络动态过程的有力工具,能帮助我们发现miRNA的调节靶点。
②利用抗体特异性结合目的蛋白的特征,将细胞内蛋白质-RNA 复合物沉淀下来,对蛋白-RNA复合物中的RNA 进行检测的方法。
③RNA-免疫共沉淀
利用抗体专一性特征,检测蛋白质-RNA相互作用。
2.RIP-qPCR 示例
①RIP-qPCR 研究细胞在有或无RA处理时 SIRT6蛋白上结合的lncRNA 。
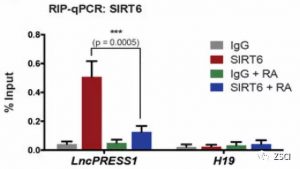
IgG或SIRT6抗体沉淀下复合物后,针对猜测的lncRNA设计引物检测是否有特定的lncRNA与SIRT6结合。
适用于验证与lncRNA结合的猜测的蛋白。
②RIP-microarray 研究6个细胞系内Ago2结合的miRNA 。
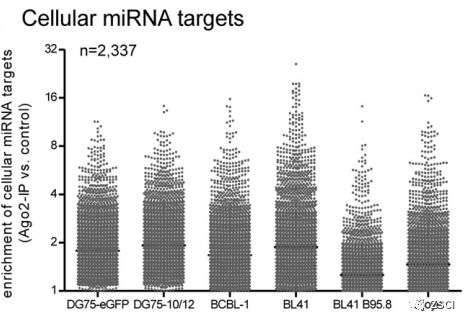
Anti-Ago2抗体沉淀下复合物后,分离纯化RNA,再进行microarray检测结合到Aho2上的miRNA。
适用于研究某个特定蛋白但是预期结合的RNA未知的情况。
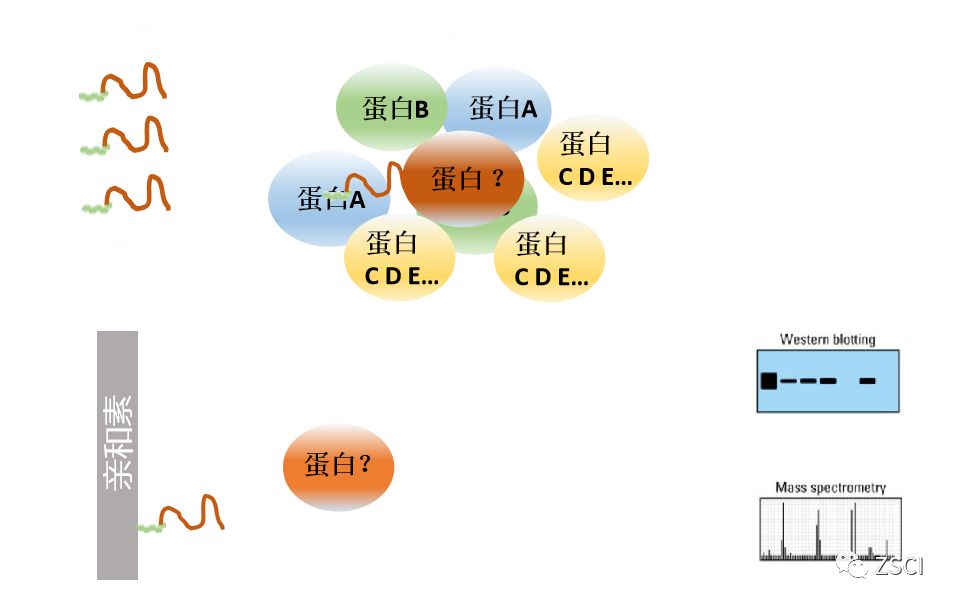
二、RNA-pull-down
1.RNA-pull-down原理
①RNA pull down, 即RNA拉下实验,是一种检测RNA与其结合蛋白(RNP) 之间的相互作用的技术。
其主要实验步骤为:
1、目标RNA生物素标记;
2、加入磁珠(通常用链亲和素酶标记)来富集RNA;
3、RNA结合蛋白孵育;
4、RNA结合蛋白洗脱;
5、进行WB检测或者MS检测(质谱检测)。
②在细胞外利用RNA-蛋白质相互作用拉下蛋白质。
2.RNA-pull-down 示例
①RNA-pull-down-WB 研究细胞内pre-miR-let-7a-1结合的蛋白质。
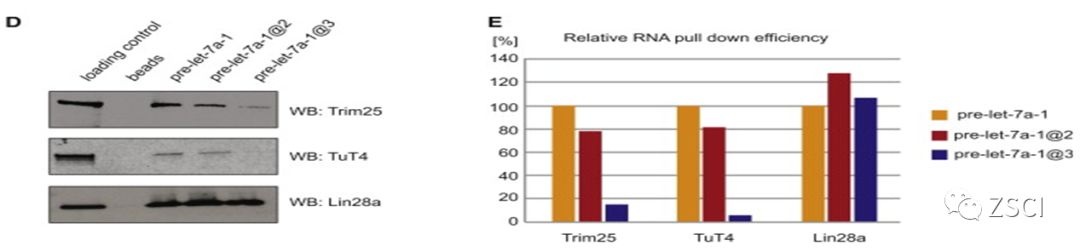
生物素结合的pre-miR-let7a-1将细胞内可与之结合的蛋白质拉下来,针对猜测的蛋白质进行WB验证。
适用于研究某个特定RNA与预期的某几个蛋白结合的情况。
②RNA-pull-down-质谱研究细胞内lncRNA TERRA结合的蛋白质。
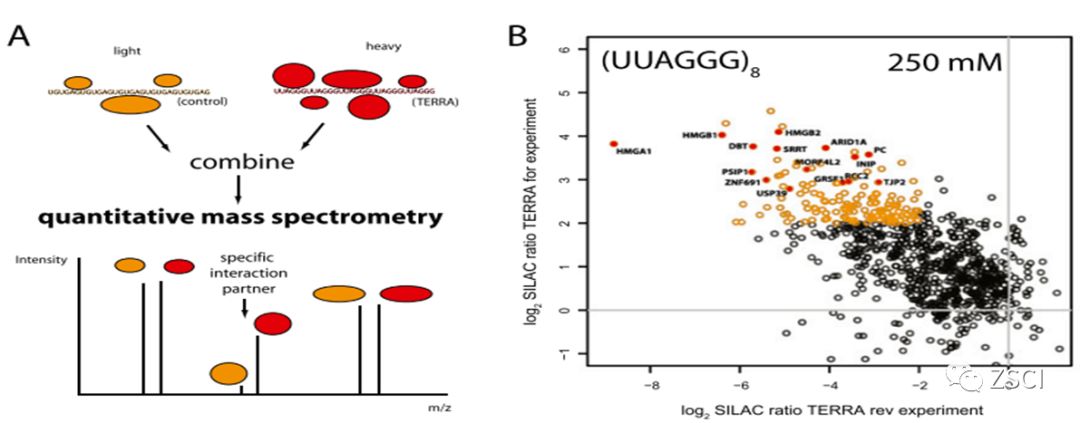
生物素标记的TERRA将细胞内可与之结合的蛋白质拉下来,蛋白纯化后进行质谱,得到可结合TERRA的所有蛋白的信息。
适用于研究某个特定RNA但是其结合蛋白质未知的情况。
三、Fluorescence In Situ Hybridization荧光原位杂交
1.FISH荧光原位杂交原理
①使用荧光标记的探针进入细胞,在互补配对、退火形成双链的基础上,将可与该探针结合的核酸(DNA.RNA)标记上荧光并显示位置。
②利用核酸互补配对特征,检测DNA、RNA位置或相对表达量。
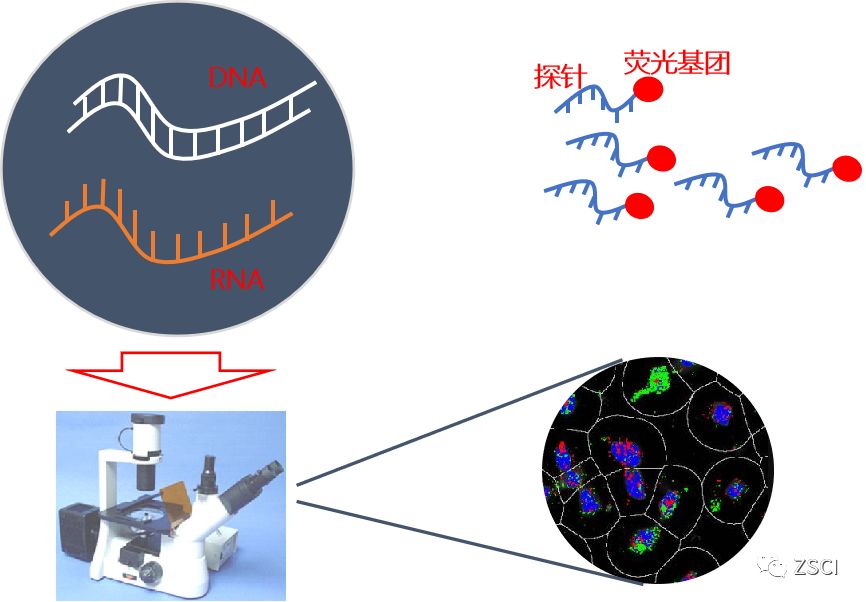
2.FISH荧光原位杂交示例
①DNA-FISH示例
针对DNA序列设计的荧光探针特异性标记基因。
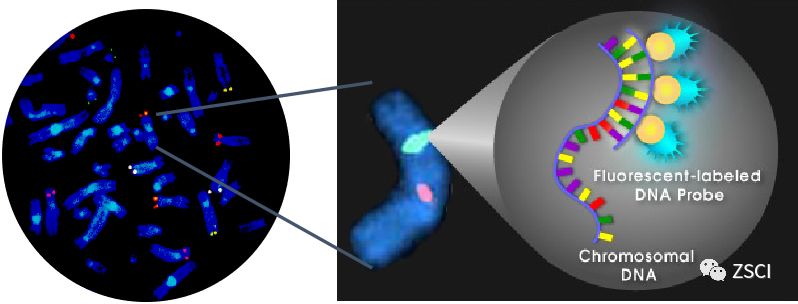
适用于研究某个特定基因在细胞内的特征。
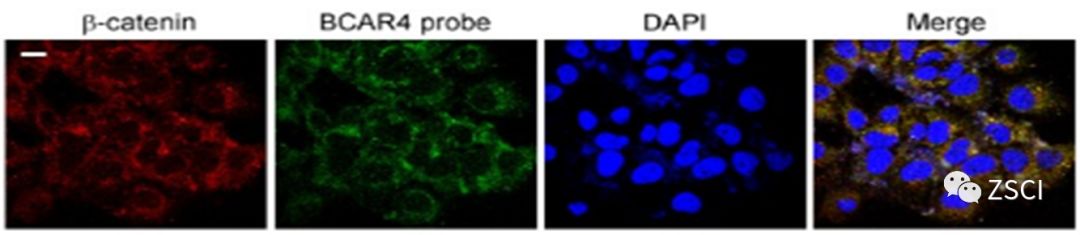
②lncRNA-FISH 示例
针对lncRNA BCAR4设计荧光探针,检测BCAR4定位。
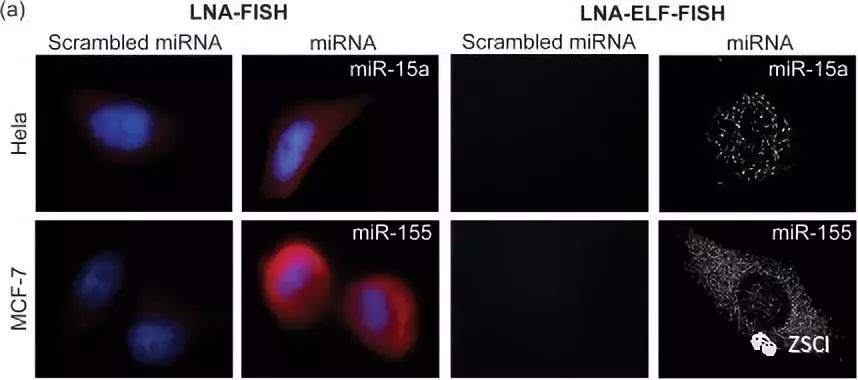
③miRNA-FISH 示例
针对miR-15a和miR-155设计荧光探针,检测miRNA定位和相对表达强度。