HOMER
安装旧版本的HOMER比较复杂,因为旧版依赖于调用其他几个工具:
- blat
- Ghostscript
- weblogo
Does NOT work with version 3.0!!!!
新版HOMER安装很简单,主要是通过configureHomer.pl脚本来安装和管理HOMER
| 1 2 3 4 5 6 7 8 9 | cd ~/biosoft mkdir homer && cd homer wget http://homer.salk.edu/homer/configureHomer.pl # Installing the basic HOMER software perl configureHomer.pl -install # Download the hg19 version of the human genome perl configureHomer.pl -install hg19 |
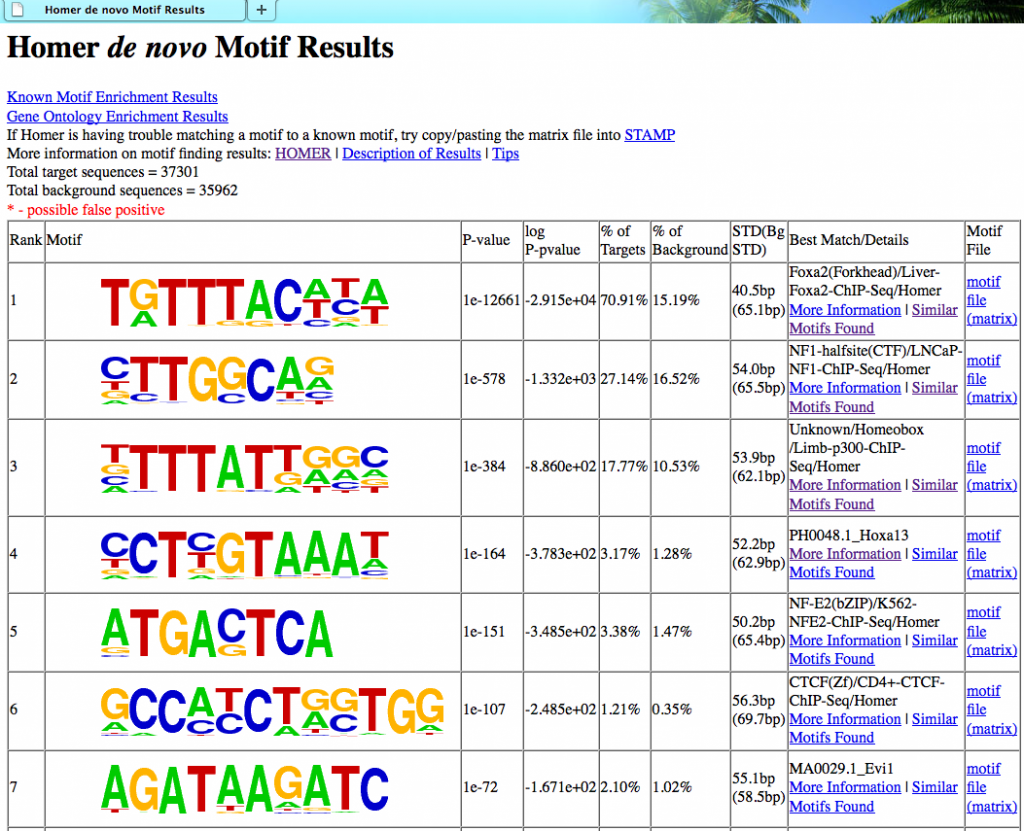
安装好后可以进行 Motif Identification
| 1 2 3 4 5 6 7 8 9 10 11 12 13 14 | # 提取对应的列给HOMER作为输入文件 # change # chr1 1454086 1454256 MACS_peak_1 59.88 #to # MACS_peak_1 chr1 1454086 1454256 + $ awk “{print $4″\t”$1″\t”$2″\t”$3″\t+”}” macs_peaks.bed >homer_peaks.bed # MeRIP-seq 中 motif 的长度为6个 nt $ findMotifsGenome.pl homer_peaks.bed hg19 motifDir -size 200 -len 8,10,12 # 自己指定background sequences,用bedtools shuffle构造随机的suffling peaks $ bedtools shuffle -i peaks.bed -g <GENOME> >peaks_shuffle.bed # 用参数”-bg”指定background sequences $ findMotifsGenome.pl homer_peaks.bed hg19 motifDir -bg peaks_shuffle.bed -size 200 -len 8,10,12 |
Usage: findMotifsGenome.pl <pos file> <genome> <output directory> [additional options]
注意:
<genome>参数只需要写出genome的序号,不需要写出具体路径bedtools shuffle中的genome文件的格式要求:1
2
3
4
5
6
> For example, Human (hg19):
> chr1 249250621
> chr2 243199373
> …
> chr18_gl000207_random 4262
>
可以使用 UCSC Genome Browser’s MySQL database 来获取 chromosome sizes 信息并构建genome文件
| 1 2 | > mysql –user=genome –host=genome-mysql.cse.ucsc.edu -A -e “select chrom, size from hg19.chromInfo” >hg19.genome > |
findMotifsGenome.pl 基本参数解释
# 基本选项
-mask 区分重复小写序列,可以添加到基因组之后,如 mm9r -bg 背景基因组位置 默认自动,用来删除与目标位置重叠的背景位置
-chopify 将大的背景区域切割为目标区域的大小
-len <#>[,<#>,<#>...] motif长度,默认8,10,12,值大于12可能会导致内存不足,从而减少序列分析数量或是分析短的序列区域
-size <#> 用于 motif 发现的片段大小,默认200
-size <#,#> -size -100,50 即获得中心-100到+50的序列
-size give 使用确定的给定区域
-S <#> 要优化的motif数量, 默认25
-mis <#> 全局优化,搜索#个不匹配的字符串,默认2
-norevopp 不搜索motif的反响链
-nomotif 不搜索de novo motif 富集
-rna 输出RNA motif logos 并且与RNA motif database 数据库自动比较,自动设置-norevopp
最后得到的文件夹里面有一个详细的网页版报告