双荧光素酶报告基因测试系统(DLR)
DLR 测试系统灵敏,方便,在一个系统中用于测量两个单独的荧光素酶报告基因,萤火虫荧光素酶及海洋海肾荧光素酶(Renilla reniformis) ,DLR测试系统可用于细胞裂解物及无细胞的翻译系统。
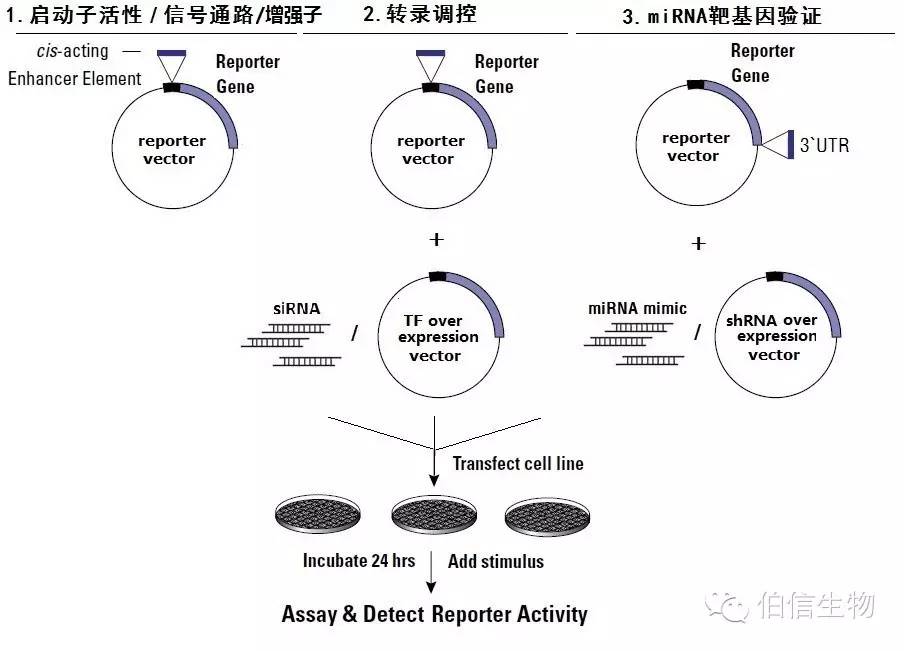
荧光素酶技术流程
1.启动子的预测
启动子是指RNA聚合酶识别并结合的DNA序列,并包括促进这一过程的调节蛋白的结合位点。启动子位于转录起始位点附近,而真核基因的转录起始位点通常需要实验进行确定。5’RACE是确定基因转录起始位点的经典方法。
在不知道转录起始位点时,我们也可以对启动子进行预测。预测基因启动子的软件有:NCBI promoter scan (http://www-bimas.cit.nih.gov/molbio/proscan/)等。
2.转录因子的预测
通常我们比较关心哪些转录因子可以调控我们感兴趣的基因,这就涉及到该基因的转录因子的预测和鉴定。
转录因子的预测软件有:
SignalScan http://www-bimas.cit.nih.gov/molbio/signal/
TF search http://www.cbrc.jp/research/db/TFSEARCH.html 等。
将待预测的上游调控序列输入进行分析,即可知道该序列中含有哪些转录因子的结合位点。(说明:这两个软件提供的预测结果不够全,有一些转录因子没有被包括其中。
3.报告基因质粒的构建
我们经常使用的质粒载体为PGL-Basic。
我们要将待分析的启动子序列插入到luciferase基因上游。
推荐使用kpnI,XhoI,BglII和HindIII这几个酶切位点,但不建议用XhoI和BglII双酶切,因为这两个酶切位点有重合。
该载体测序用RVP3(正向)和GLP2(反向)。
4.实验方法及结果分析
连续截短确定核心启动子。克隆基因的上游调控序列,做连续的截短,如构建-2000~-1500,-1500~-1000,-1000~-500,-500~0,0~500 一系列克隆,转入细胞,观察荧光素酶活性,以此判断哪一段序列对于该基因的转录活性起关键作用。例如:-2000~-1500和-1500~-1000有很高的活性,而-1000~-500活性很弱,则可判断核心启动子区在-1500~-1000。
特定转录因子对转录活性的分析。我们构建包含该转录因子的启动子序列(上游调控序列)的报告基因质粒,与转录因子的过表达载体(或RNAi)共转染细胞,观察转染前后报告基因活性的变化。如果转染后报告基因活性发生明显改变,则可判断该转录因子可能调控目的基因的转录。
您好,学长!请问我PGL3-basic用了Xho Ⅰ和nhⅠ,测序并没有切开,请问我现在换成kpnI,XhoI这两个酶切位点,可以切开嘛?
请问如果我要验证转录抑制的话用luciferase报告基因的方法怎么做呢?
楼主很厉害!分享的东西都很有用。想请教一下:对于一个序列已知的基因,比如IL1,也需要预测启动子序列吗?还是说我可以通过NCBI找到启动子序列,直接放在pGL3的luciferase序列的前面?
非常感谢!
序列已知的基因是指的是cDNA吗?这个不包含TSS序列,需要做luciferase试验时,需要首先预测转录因子结合靶基因TSS的位点,有另一篇文章有介绍工具预测与可视化转录因子结合位点的在线工具,得到结合位点之后一般pcr扩增包含结合位点和100-300侧翼序列插入报告基因质粒
要知道一般认为TSS是起始密码子上游1500-2000bp,不建议将整段插入pGL3,太大了
有其他问题可以微信继续讨论 我的简历有微信