今天为大家介绍一款预测和展示核酸(RNA和DNA)二级结构的在线工具。
其实早在去年9月份就有规划做这样一期教程,一年来一直没能找到一款出图“不丑”的工具,直到上周遇到了Mfold,看了首页的结构图后我心底就冒出一个声音:终于等到你!
Mfold 其实是计算分子生物学中最古老的在线工具之一,自1995年在华盛顿大学医学院上线以来持续工作至今。2003年7月在Nucleic Acids Research上发表的一篇介绍Mfold的文章已被引用了10520次。

我们一起看下别人家的文章是怎么用Mfold出图的吧。
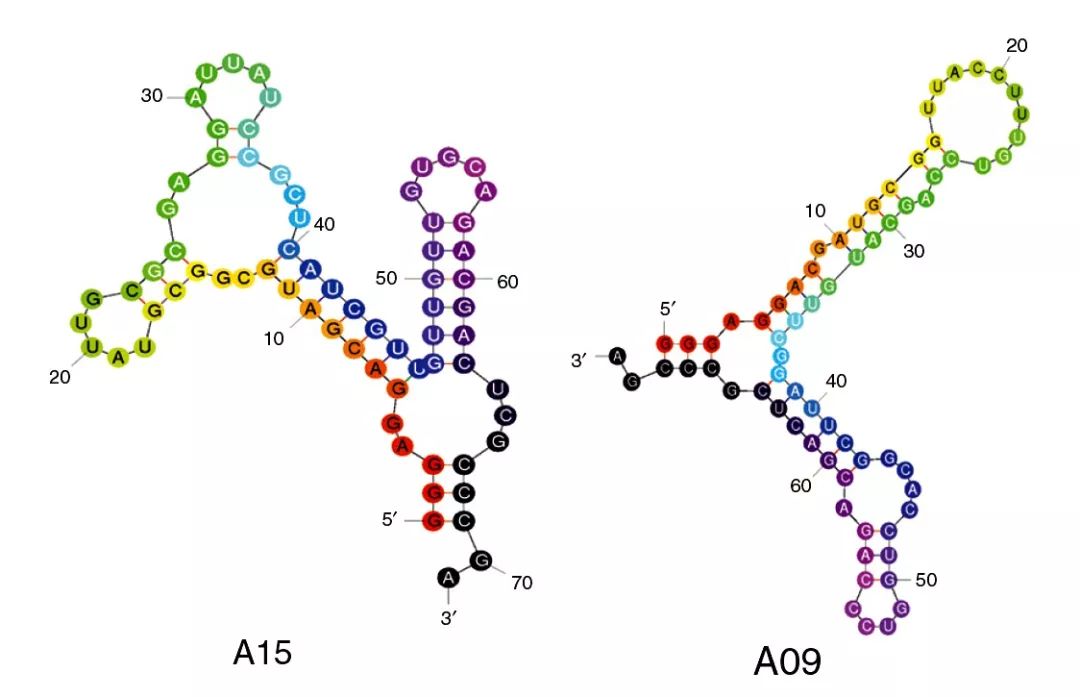
(Molecular Therapy-Nucleic Acids,2013)
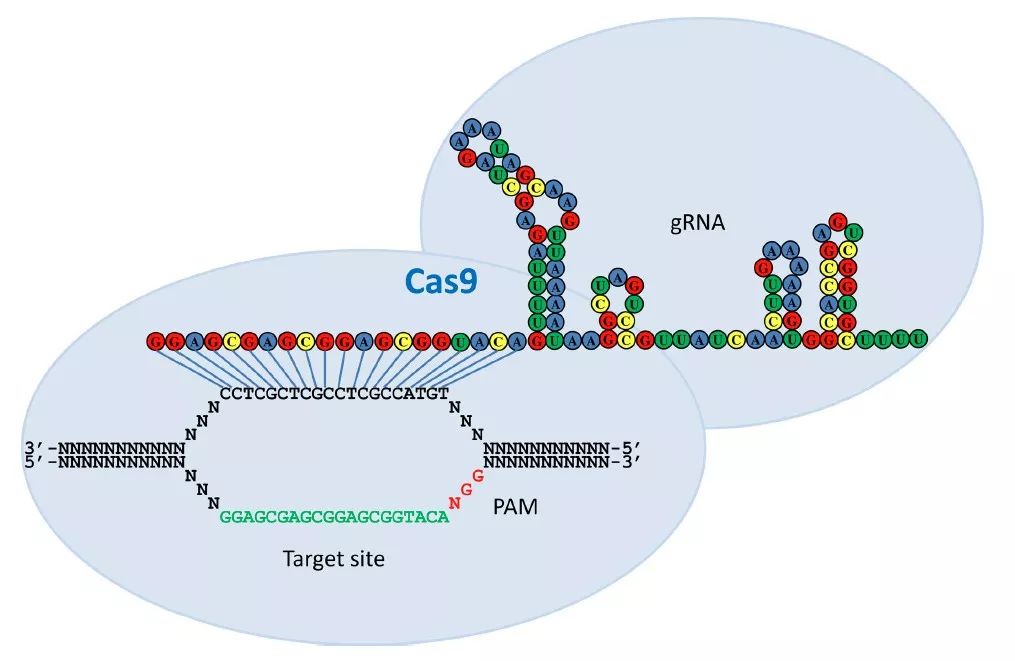
(Nature biotechnology,2013)
好啦,下面就开始看下如何使用Mfold吧。
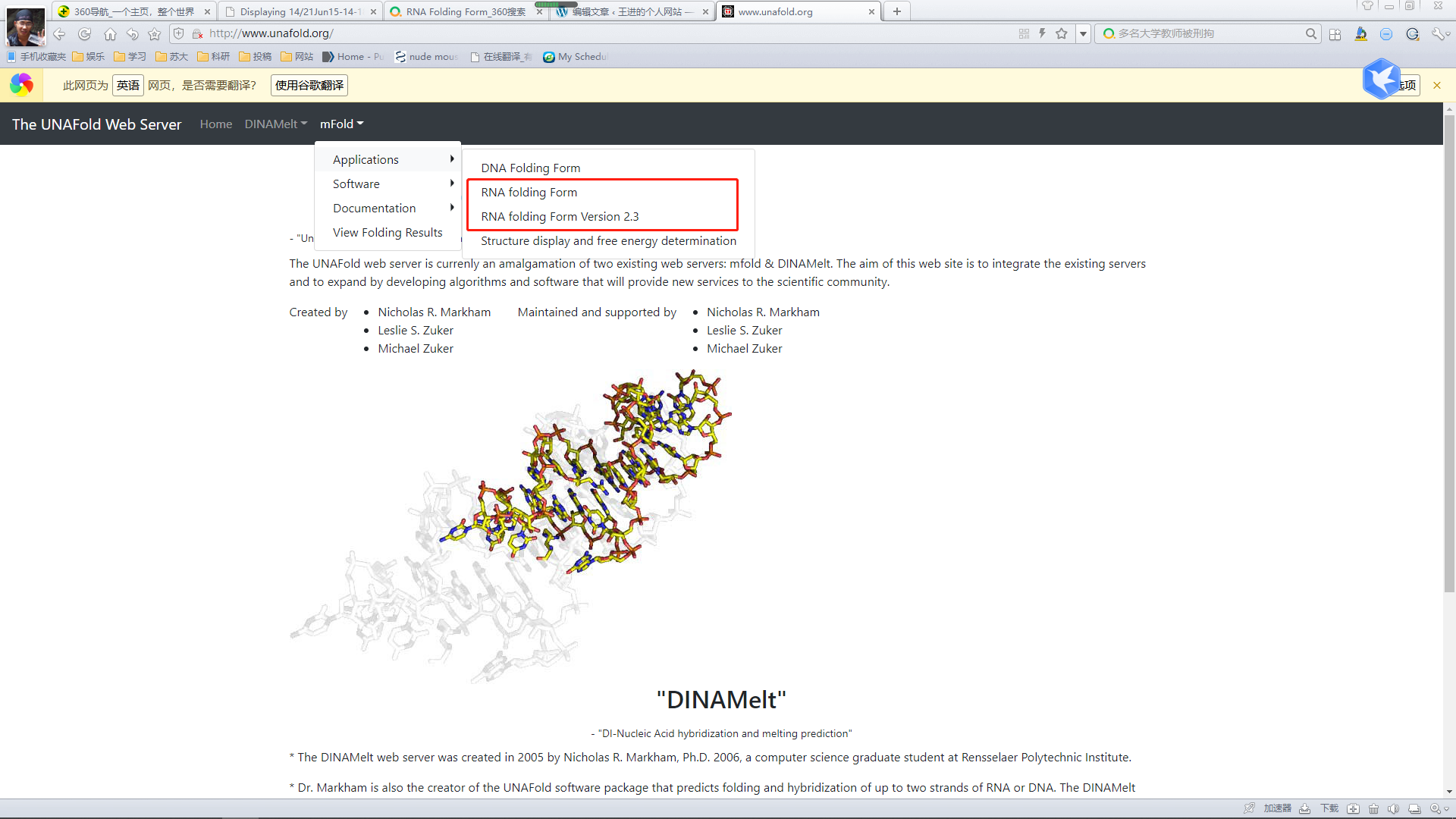
首先,打开Mfold的工具页面(旧网址:http://unafold.rna.albany.edu/?q=mfold),新网址:
RNA Folding Form:http://www.unafold.org/mfold/applications/rna-folding-form.php
RNA Folding Form V2.3:http://www.unafold.org/mfold/applications/rna-folding-form-v2.php
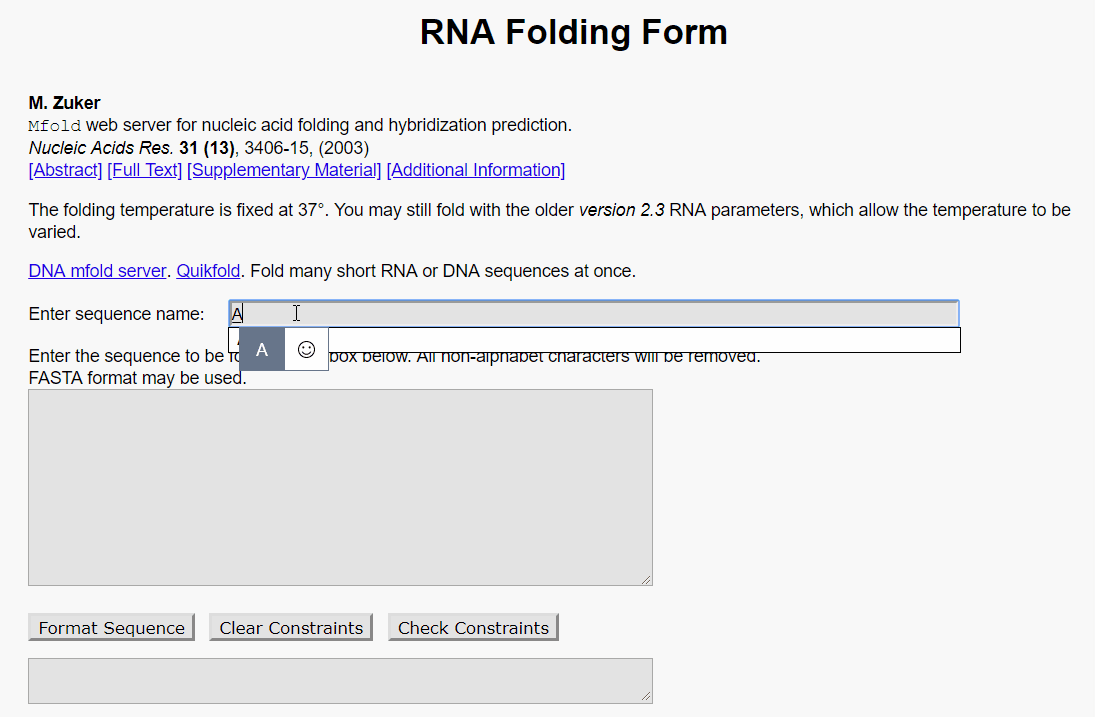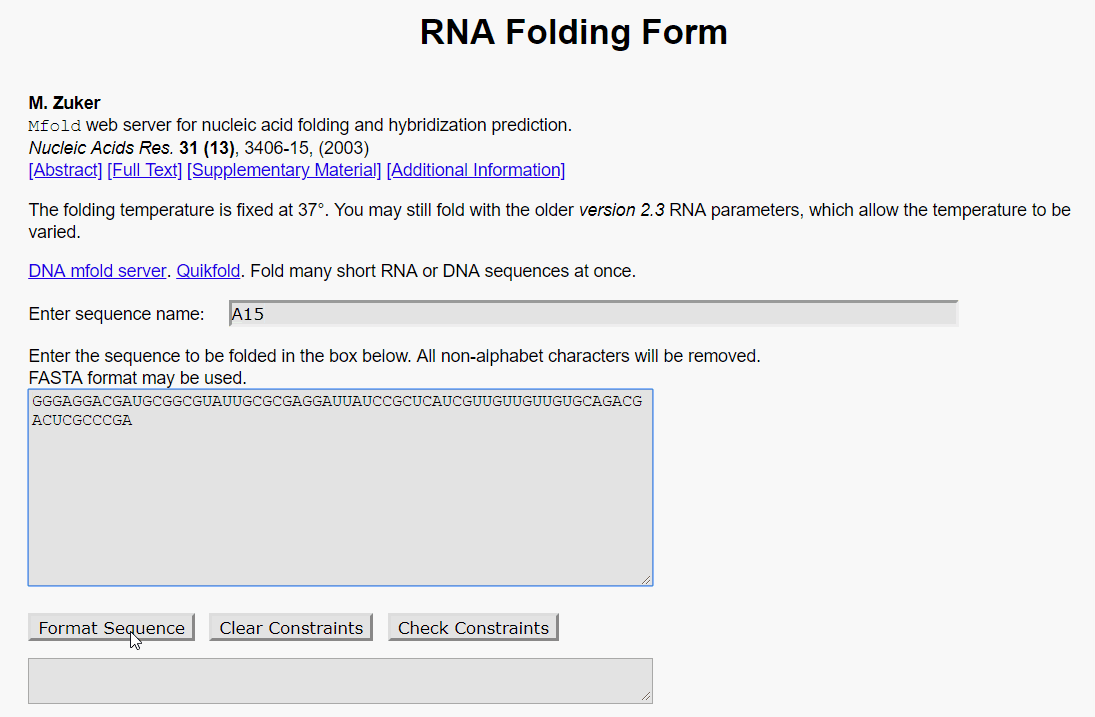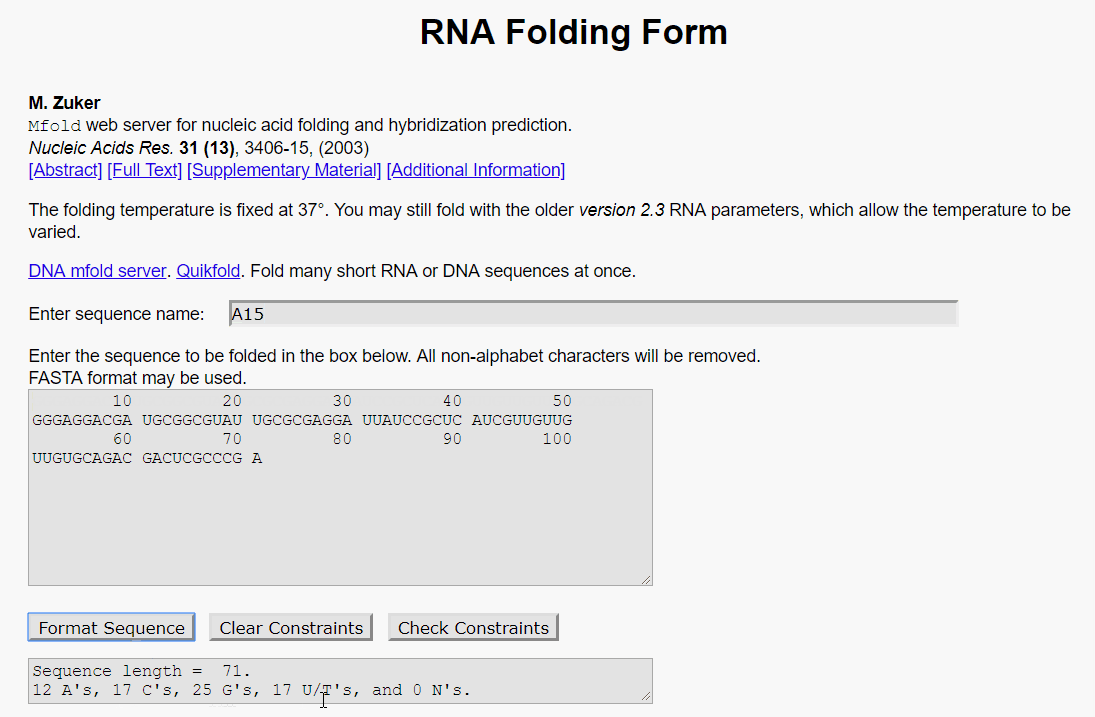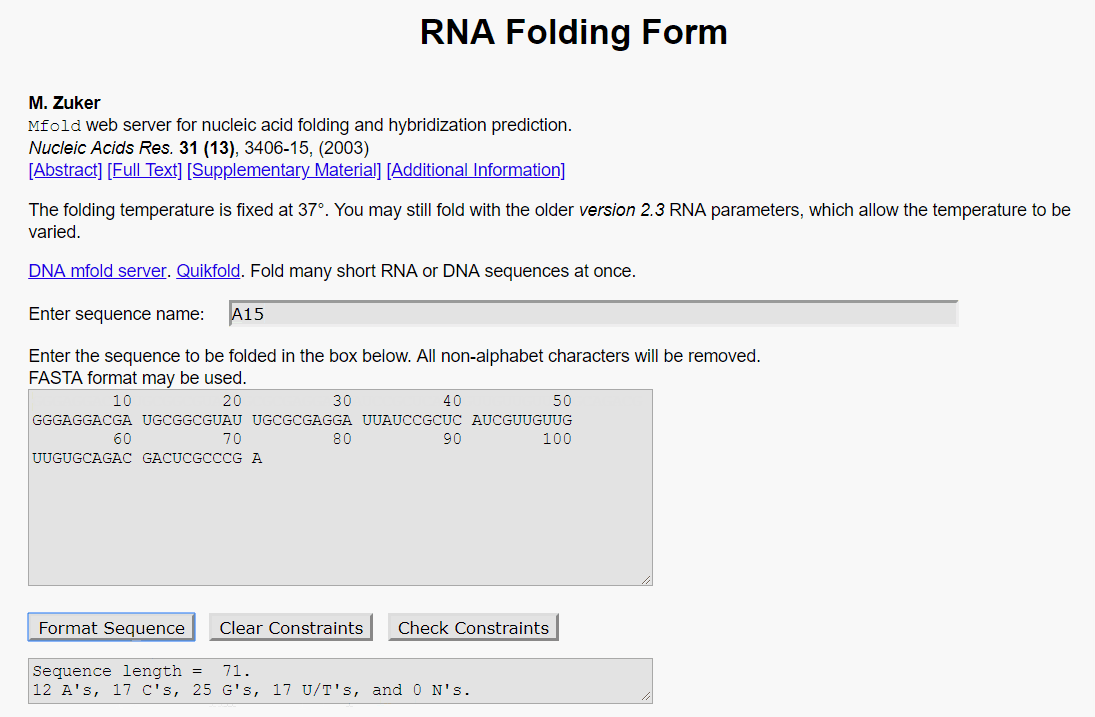
点左侧窗口的RNA Folding Form,进入RNA二级结构预测窗口。将单条RNA序列复制粘贴到序列框中,记得为序列命名,点Format Sequence按钮可清楚查看序列的长度,后面的两个按钮用来去除序列中的其他“字母”,比如“N”。
这里用的RNA序列为第一个示例文章中A15的序列(Molecular Therapy-NucleicAcids,2013)。为了方便大家练习,我把A15和A09的序列复制粘贴到这里,如下:
>Aptamer A15
GGGAGGACGAUGCGGCGUAUUGCGCGAGGAUUAUCCGCUCAUCGUUGUUGUUGUGCAGACGACUCGCCCGA
>Aptamer A09
GGGAGGACGAUGCGGCGUAUUGCGCGAGGUUAUCCGCUCAUCGUUGUUGUUGUGCAGACGACUCGCCCGA
一般情况下,生信软件的参数设置最简单的,因为大多数情况下“默认设置”是最佳的选择。对于Mfold来说其实也这样的,它会根据你提交的序列长度自动匹配相应的参数。不过,我这里还是会给大家介绍一些我觉得比较重要的参数。在此之前,我们先看下RNA的二级结构相关的一些术语,如下图。
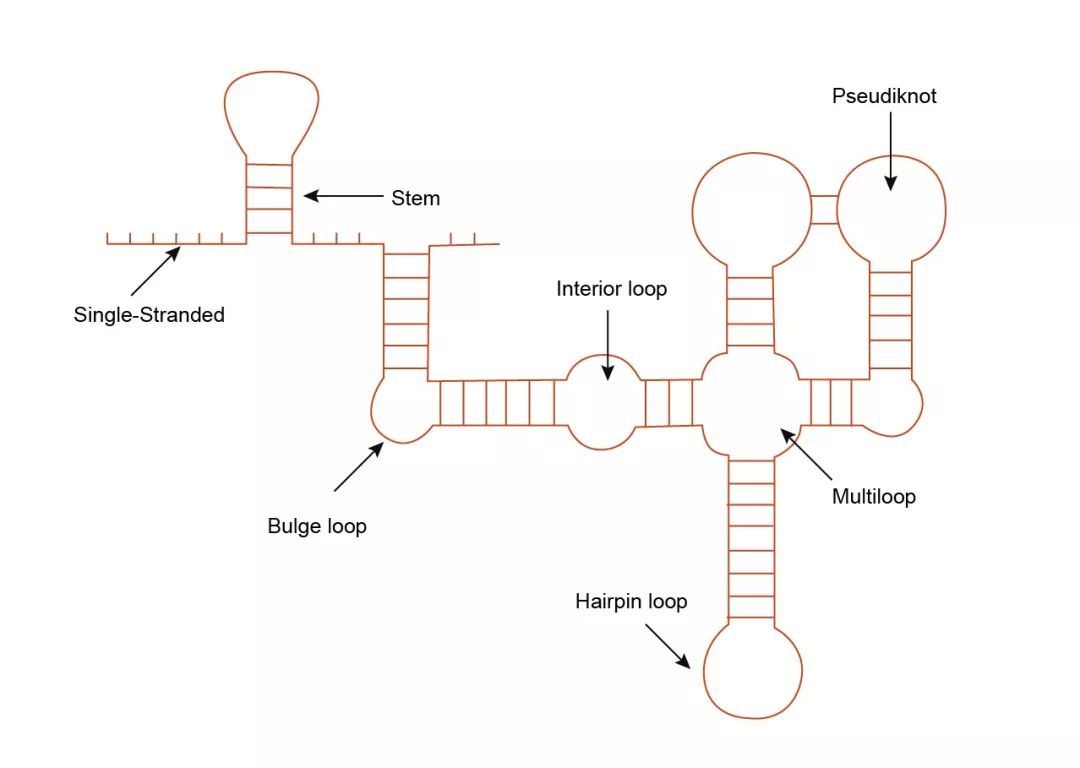
RNA的二级结构
看完上图,接下来的参数设置就容易理解多了。比如下面的控制窗口,在窗口中输入“ F 7 0 5 ”表示强制序列的第7~11个碱基形成双链,当然这样做的前提是你已经有确定证据。

下面的这些关于算法的参数也保持默认就好,大家如果感兴趣可以点击蓝字查看每个参数的详细解释。
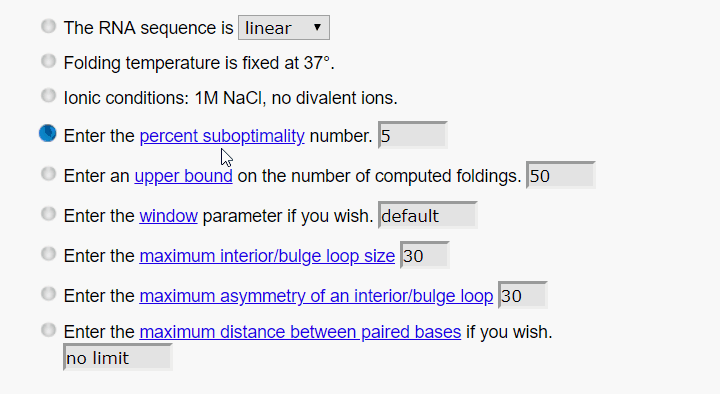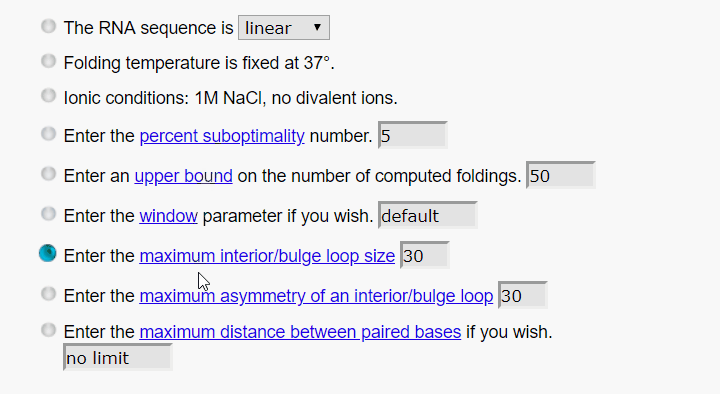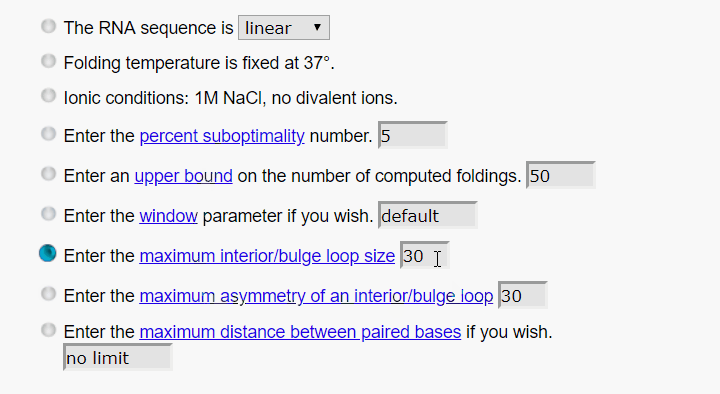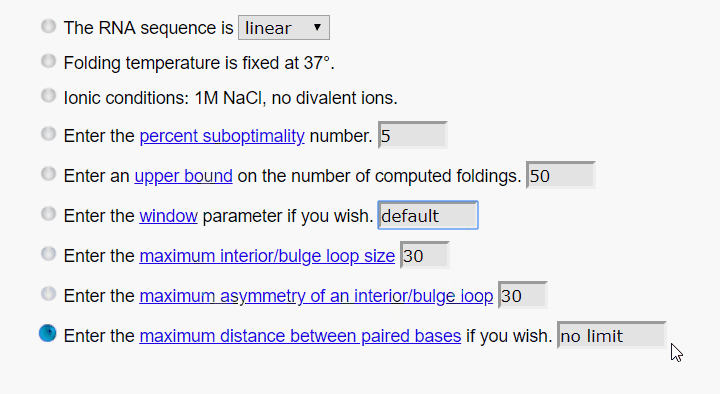
接下来是关于结构图的设置,保持默认就好,如果是批处理,可填写自己的邮箱。大多参数在结果窗口还可以接着调整,直接点Fold RNA提交任务。
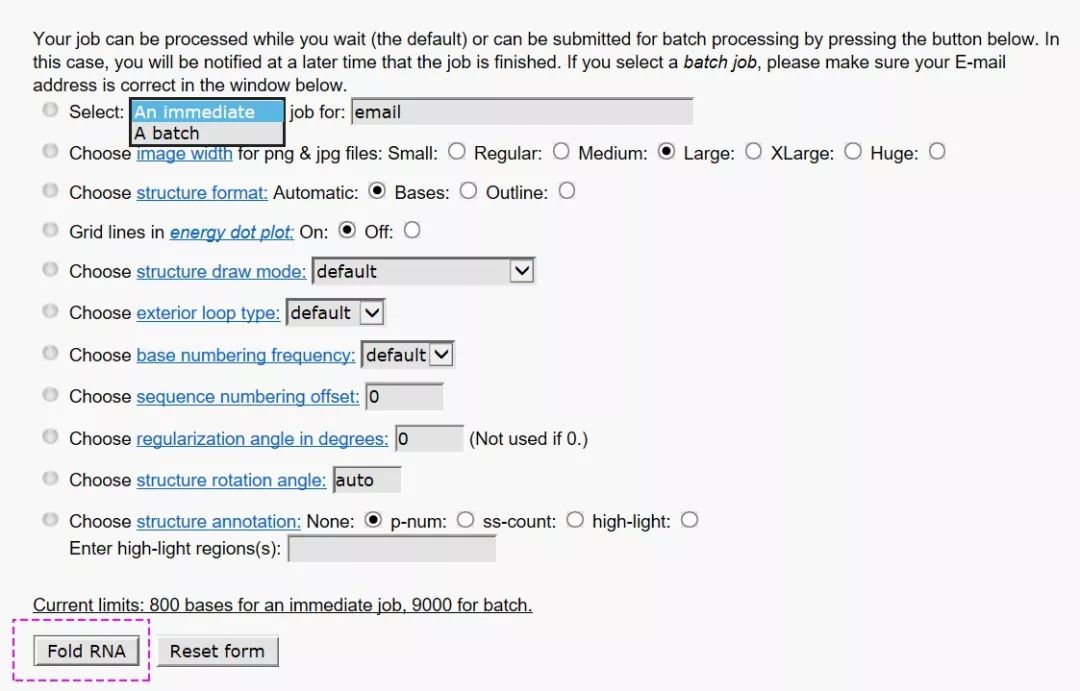
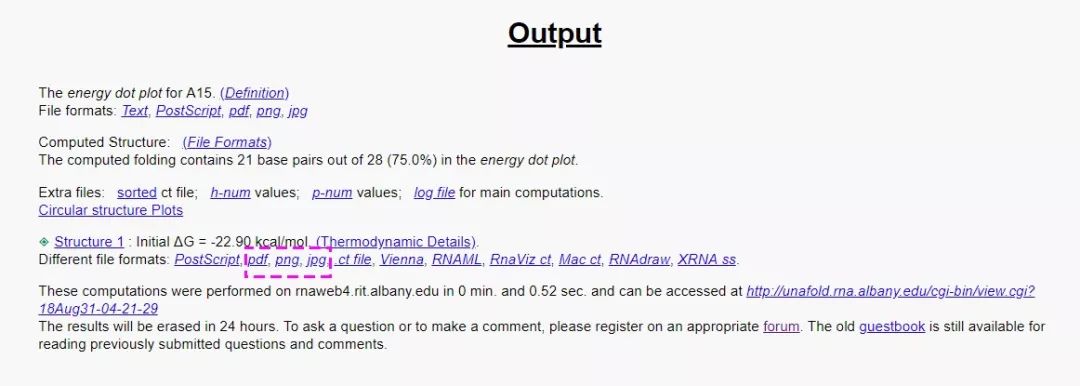
预测的结果会出现在新窗口中,如下图,关于dot plot 和 circular plot 这里不做展开,我们只需点击相应的文件格式,就可进入结构文件调整窗口,比如这里点jpg(你也可以保存为“.ct”格式,用其他RNA二级结构可视化软件编辑)。
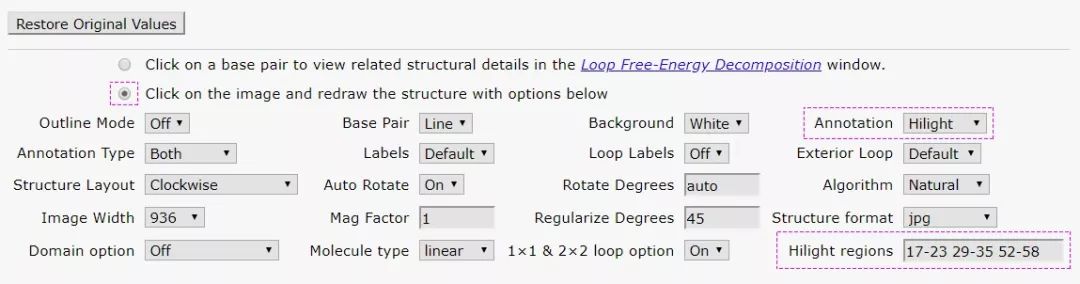
接下来可以对结构图进行自定义调整,比如这里的注释方式选Hilight,在Hilight regions添加着色的碱基范围,如下图,点一下图片,图片会自动刷新。初学者可选一个参数,点一下图片,这样就可以看到每个参数的效果,这一点非常赞。如果没反应,注意查看你的浏览器是否阻止窗口弹出。
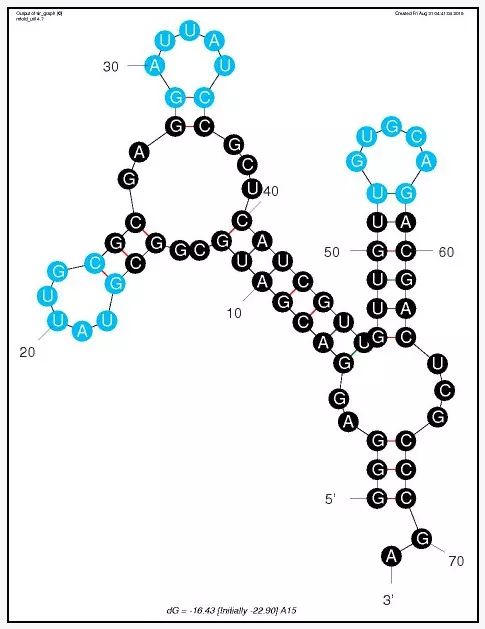
自定义的着色效果如下:
如果要下载pdf格式的图片,可将Structure format选择成PDF,点击图片就会弹出一个新窗口,接下来就可以像下文献一样把图片下载下来了。
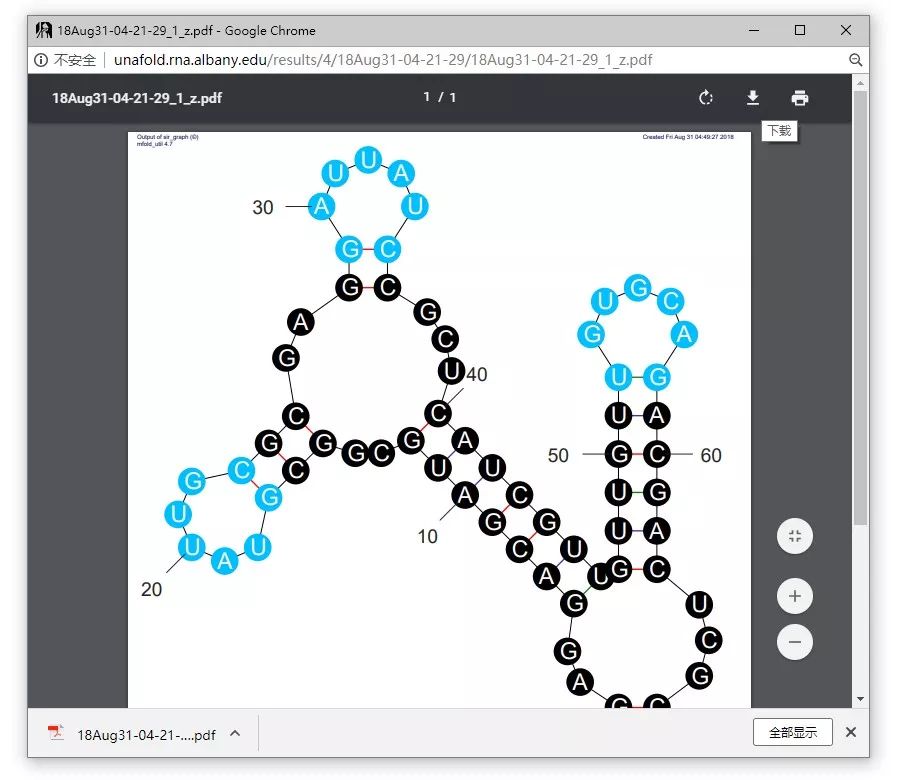
只要拿到PDF格式的图片就可以用矢量图编辑软件比如Illustrator(Ai)做更精细的调整、美化,甚至绘制出那篇NB文章中的(示例2)组合图。
今天的内容就到这里啦~
参考文献:
Zuker, M. (2003). Mfold web server fornucleic acid folding and hybridization prediction. Nucleic acids research,31(13), 3406-3415.
Cheng, C., Chen, Y. H., Lennox, K. A.,Behlke, M. A., & Davidson, B. L. (2013). In vivo SELEX for identificationof brain-penetrating aptamers. Molecular Therapy-Nucleic Acids, 2.
Hwang, W. Y., Fu, Y., Reyon, D., Maeder, M.L., Tsai, S. Q., Sander, J. D., … & Joung, J. K. (2013). Efficient invivo genome editing using RNA-guided nucleases. Nature biotechnology, 31(3),227.
老师您好,请问突变位点在外显子上,我想看一下突变位点是否会引起mRNA二级结构改变,请问我是要输入整段外显子碱基还是只输入突变位点前后20左右bp碱基即可?
请问这个画图完用什么软件上色啊?
为什么打开都显示 error
你好,请问这个网站需要翻墙才能使用吗?怎么总显示 404 NOT FOUND
你好,请问这个网站可以预测硫代修饰的DNA吗?
老师您好,请问一下用DNA foldding form 的 时候,我会模拟单链结构,但是我想要表达经过扩增后这个重复的单链结构,应该怎么操作呢?
您好
没明白什么是 “表达经过扩增后这个重复的单链结构” ?
就是有一段单链DNA序列,比如是ATGCCCAAGGGAAA能形成一个特殊的形状,在经过PCR或者其他方式扩增后,是N个这样的ATGCCCAAGGGAAA重复性长长的序列,这个软件可以模拟出这个扩增后这段长序列的N个这样的形状吗?
进老师好,请问如果是对核酸链开头和结尾部分进行了修饰,例如甲基化。该在图里怎样表示呢?
咦 这可能得画完之后手动在绘图软件加上了 具体如何体现看文献中怎么画的
您好,请问您弄清楚怎么弄了吗?
老师您好,我想问一下RNA Folding Form与RNA Folding Form V2.3有什么不一样的吗,因为它们预测出的结果不太一样。
您好,这个你需要看官方说明,网站有都有介绍的,我没有研究过
您好,这个网站可以处理两条杂交链的二级结构吗?谢谢,木有太整明白操作,谢谢。麻烦啦
您好,这个网站的另外一个工具貌似可以,我试了一下,结果界面Results下面有杂交二级结构图。你可以试试:顶部菜单DINAMelt–> Application –> Two state melting hybridizattion
您好,请问您会了吗,我也有相应的问题和需求qwq如果您会的话能麻烦教我吗
你好,请问怎样进行批量预测结构呢,我尝试每一列都加入一组序列,但最后的结果把我所有的序列都加在一起预测了,我看输入窗口也没有批量输入文件的功能。
你好 网页版是不可以的,只能处理一条序列,如果需要处理多条,只能一个个任务进行,或者下载本地版进行批量操作
好的,谢谢解答。
还有请问,本地程序是只能在LINUX环境下安装吗
Download mfold:Version 3.6 (and earlier versions) of mfold for Unix, Linux and Mac OS X operating systemsis available via a free license for academic and nonprofit use only;
网站确实出现问题了,DNA预测不了
今天已经可以访问了,刚刚试了一下,只能设置hilight的碱基(蓝色),不能自定义颜色。如果需要可以下载对应编辑软件格式的图片文件进行编辑
将碱基Highlight,能否改变碱基的颜色,窗口没有弹出,是不具备这个功能吗?
您好,mfold网站现在好像有问题,没法打开结果页面,晚一点再试试。改不了的话您可以保存为.ct格式,然后在photoshop或其他图像编辑软件里面更换颜色
你好,circRNA的二级结构也可以通过这个预测吗
当然是可以的,输入序列后可以选择是线性或环状
想请问一下,出的图有结构1、结构2等等,每个图都有差异,应该相信哪一个比较好?
看吉布斯自由能△G,越低应该越可靠
你好,Mfold这个在线网站已经不运作了,请问还有最新的网站吗?谢谢
你好,该工具并不是不运作,只是迁移了,你应该打开看到网页提示:The UNAfold webserver hosted at the RNA Institute has been discontinued as of November 1st, 2020.
Please proceed to http://www.unafold.org for information. A new webserver is being hosted at this URL. mfold工具整合到了UNAfold网站
文章已更新,附上了正确的打开方式。