随着表观遗传学研究的不断深入,研究人员发现了一种可逆性的 RNA 甲基化—m6A,同时陆续定位了哺乳动物转录组中的 m6A,鉴定了具有动态修饰的「readers」、「writers」和「erasers」蛋白,解析了 m6A 在转录后调控中的一些功能。由此,RNA 甲基化(m6A)研究在表观遗传领域开始崭露头角,成为前沿的热点!
RNA甲基化修饰过程中的关键分子
Writers:将甲基化修饰“写入”RNA,即介导RNA的甲基化修饰过程。最常见的分子是METTL3和METTL14,两者可在体外和体内催化mRNA(和其他细胞核RNA)的m6A甲基化。WTAP是这种甲基转移酶复合体中的另一个关键组分。
Readers:“读取”RNA甲基化修饰的信息,并参与下游RNA的翻译、降解等过程。比如具有YTHDF结构域的蛋白能够识别并结合mRNA中的m6A,而这种结合会减少mRNA的半衰期促使其降解
Erasers:将RNA甲基化修饰信号“擦除”,即介导RNA的去甲基化修饰过程。FTO和ALKBH5可以去除mRNA(和其他细胞核RNA)上的m6A甲基化。
想蹭热点?给大家介绍一个免费在线预测哺乳动物m6A修饰位点的网站SRAMP。
进入SRAMP网页
点击“Prediction”按钮
选择预测的模型
左边的“Full transcript mode”在对编码和非编码RNA进行预测时,建议使用此模式。 注意,在该模式中,应使用完整转录物(具有内含子)而不是成熟mRNA / cDNA序列的基因组序列。而对于没有完整基因组序列的用户来说,可以选择右侧的“Mature mRNA mode”,这种预测模式是一种备选解决方案,相对来说预测的准确性不如左边的,该模型适用于成熟mRNA(cDNA)序列,不能预测内含子中的m6A位点。
下面我们演示的是网站默认的test1(TROVE2)序列,后面的选项都是默认的,点击“Submit”就可以预测序列的m6A结合位点了。

耐心的等待

预测结果
有两个结合位点,在“Decision”列中可以看出预测的两个结合位点具有很高的可信度。SRAMP网站提供了几个阈值:99 %/95%/90%/85%,分别对应very high/high/moderate/low的自信度,当然我们一般都挑选“very high”啦。预测结果是按照“Position”位置排序的,所以还需要综合各个位点“Score”情况。

SRAMP网站还提供了RNA二级结构m6A位点结合预测功能,写材料,发文章,有个图不是更加美观?看到这个画面的时候,网站默认选项是“NO(default)”,我们选择“YES(much slower)”,然后点击“Submit”就行,计算过程需要花更多的时间,这个取决于提交序列的长度,请各位客官耐心等待。

预测结果

点击“Draw”,RNA二级结构m6A位点预测图就出来了
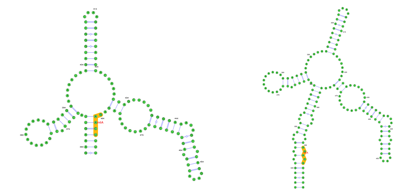
SRAMP网站运算速度比较慢,如果需要预测的序列较多,有一台不错的电脑,可以自行下载SRAMP tool(压缩包大概2G),同时安装R语言和Perl语言就可以运算了。
SRAMP算是一个不错的网站,界面相当简洁,由北京大学创建的;还有两个m6A甲基化预测的网站都是由中山大学创建的,点个?。
(1)RMBase v2.0
网址:http://rna.sysu.edu.cn/rmbase/
(2)m6Avar
网址:http://m6avar.renlab.org
老师您好,最近网站新出了deepSRAMP,预测的结果有什么不同呢?我看多了好多结果,编号也是在之前的基础上继续编的,请问这个相比之前的有什么区别呢?最新的这个deepSRAMP好像还没有引用的文章
老师好,请问我在完整转录模式中输入序列之后显示序列太长怎么办?
老师好,如果一个基因有不同的转录本怎么办呢
老师,你好我想问一下,SRAMP预测出来的序列,引物设计公司说序列太短了没法设计引物,怎么解决?
老师好,请问可以出RMBase和RMVar的预测m6a位点的详细流程吗?
老师好,点击draw后只出现了m6A修饰位点大图,而没有完整的二级结构图是怎么回事呢?
有个问题 输入的是DNA序列,要预测RNA的甲基化位点的话 是不是要输入互补的RNA序列呢?
如何查找我的目的基因的基因组序列啊
NCBI Ensembl都可以哇,我习惯用Ensembl,export里面选择genome就可以
老师好,请问这个网站所预目标基因的m6A甲基化位点,是预测该基因的m6A甲基化水平高低?例如预测出的位点多,表明m6A水平高。 还是为了后续检测该基因甲基化设计引物序列呀?
你好 这个网站是用来预测位点的,与甲基化高低无关,确如你所说,预测位点用于设计引物进行鉴定
老师您好,请问一下怎么根据预测到的甲基化位点,设计这个位点的引物呢
老师,你好。想问一下我知道这个m6A位点后,怎么设计改靶基因的引物?