这个在线站点功能比较丰富,可以选择基因编辑工具的其他用途(激活,抑制基因表达等)(图1-1)。
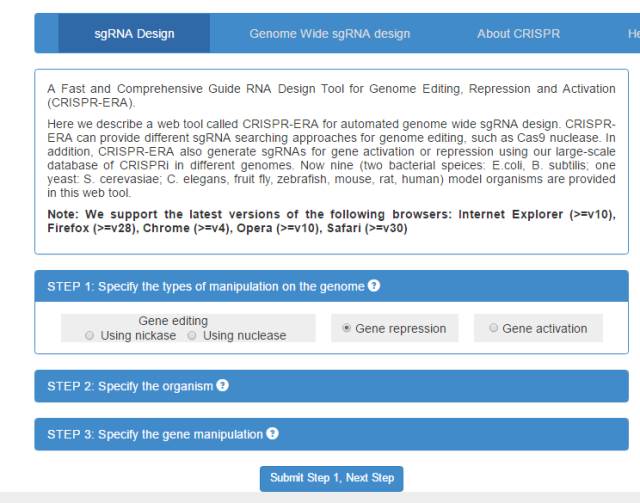
图1-1 选择编辑类型
下一步就是选择物种(图1-2)。但只有常见的9种。
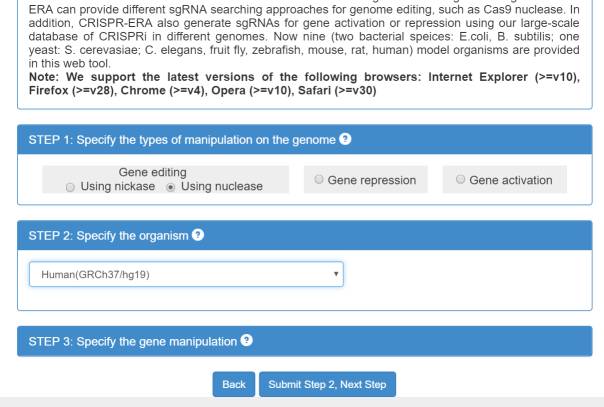
图1-2 选择物种
再下一步就是直接输入基因的名称(或序列)即可(图1-3)。
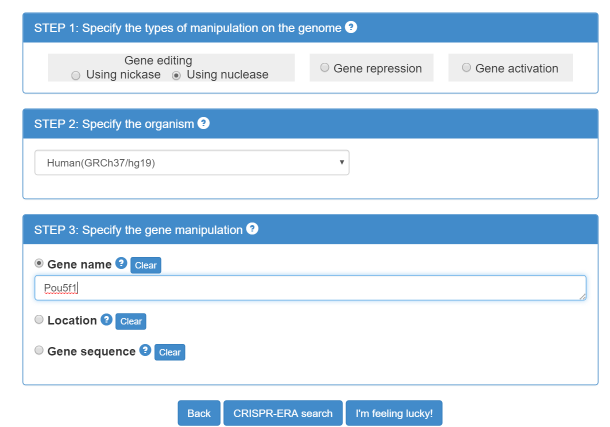
图1-3 选择基因名称。
但是出来的sgRNA位置有一些并不总在ATG的下游(图2所示,1,2,6,9,12,16,22几条sgRNA位于ATG的上游)。所以根据位置,对于做基因的敲除(KO)而言,强烈建议选择ATG下游通常100 aa以内范围内的sgRNA来用(比如图3中的3,5,7,10等较下游的);而且,一定要位于CCDS区域内(CCDS是consensus CDS,即公共的CDS区域,是针对很多个转录本[isoforms]定义,图2中路色条框即是。这个很重要!!!)。
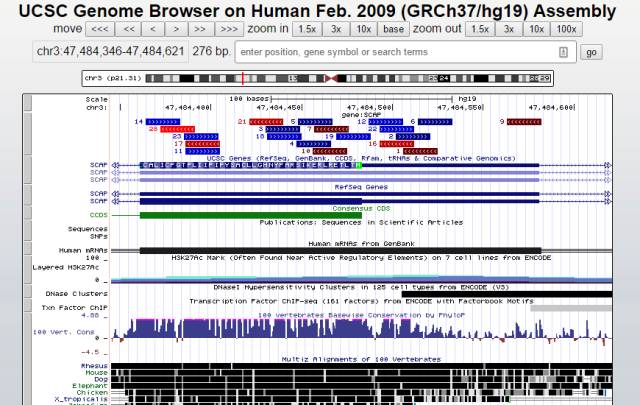
图2
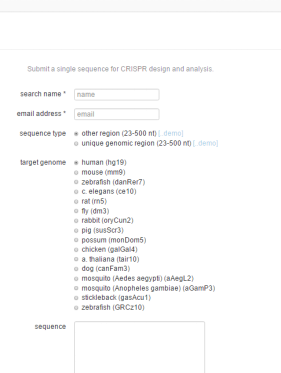
图3
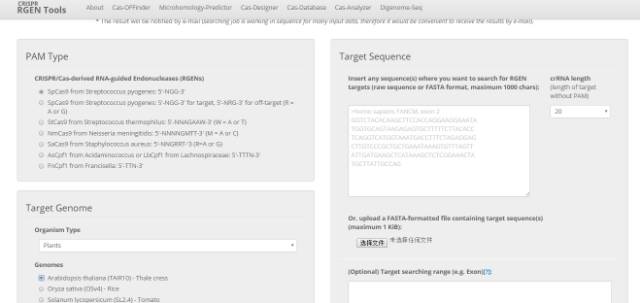
图4
 图5
图5
图6
手动版本虽然费事,但有时候却会事半功倍,节约后续鉴定单克隆的成本。我们举例来说:

图7

图8
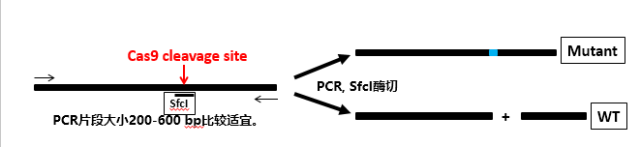
图9
☞ 注意:PCR引物设计的时候确保PCR片段内部不要出现第二个SfcI的酶切位点。这样后面酶切的时候结果是唯一的。实验流程和鉴定结果应该是这样的(图10):
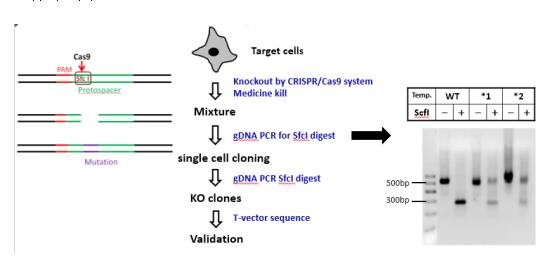
图10

图11
TA克隆结果(图12):

图12
因为以上介绍的站点没有涉及Cpf1和saCas9,特别是saCas9(非常适合AAV介导的载体基因编辑)。http://crispr.cos.uni-heidelberg.de/index.html 这个网站兼容几乎所有类型的Cas9设计。
打开页面,输入要编辑区域的基因组序列,然后在PAM类型里选择你需要的Cas9类型,比如spCas9,saCas9以及cpf1等。其他条件默认设置即可,然后点击sunmit即可。非常傻瓜式的操作(图13,14)
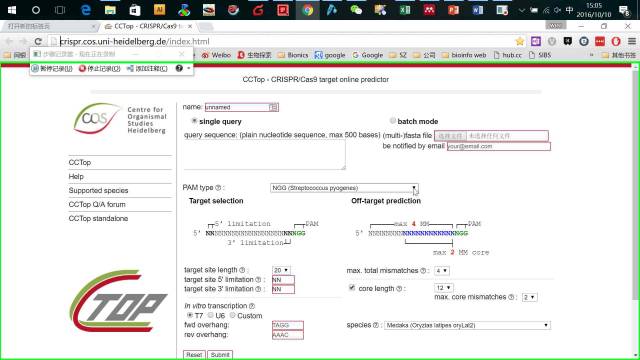
图13
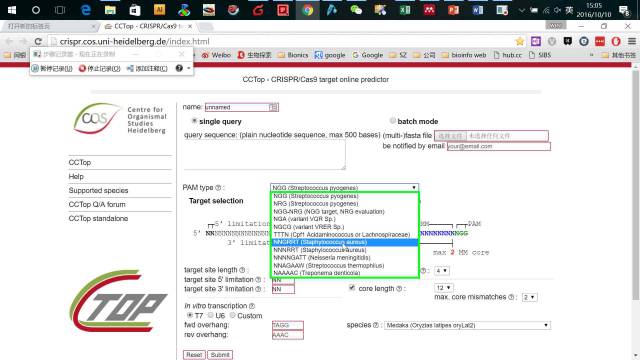
图14