今天进哥花了一个小时左右学会了一个新技能:网络韦恩图。现在我就把简要教程分享给大家,平时比较忙,整理的着实比较简洁,有问题欢迎沟通讨论。
(1)首先,按如下格式整理好所需绘制网络韦恩图的表格(就是我用VennDiagram R包绘制venn图的文件:本文所用数据下载链接Venn):
library(readxl)
CT <- read_excel("E:/Venn.xlsx",sheet="Down")
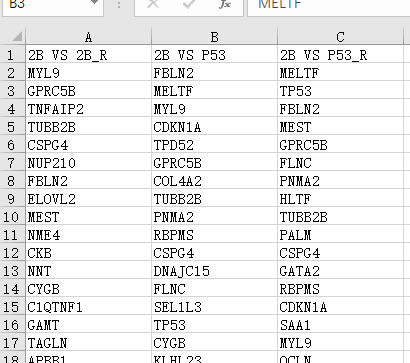
(2)使用R语言整理数据,当然不嫌麻烦也可以使用Excel整理成Cytoscape所需格式:
B2_R<-data.frame(CT$`2B VS 2B_R`)
names(B2_R)<-c("Symbol")
B2_R[,2]<-"2B_R"
P53<-data.frame(CT$`2B VS P53`)
names(P53)<-c("Symbol")
P53[,2]<-"P53"
P53_R<-data.frame(CT$`2B VS P53_R`)
names(P53_R)<-c("Symbol")
P53_R[,2]<-"P53_R"
all1<-rbind(rbind(B2_R,P53),P53_R)
write.csv(all1,"I:/down.csv",row.names = F)此时得到第一个绘图文件,Excel打开删除第一列(或者在R语言保存时不导出rowname)
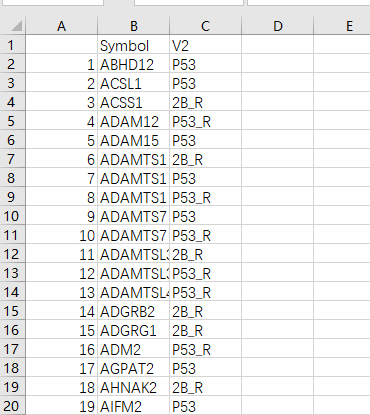
(3)继续保存为另一个格式,为后续绘图准备
all2<-merge(merge(B2_R,P53,by="Symbol",all = T),P53_R,by="Symbol",all = T)
write.csv(all2,"I:/down_Group2.csv")
Excel打开将表格中NA全部删除(替换功能),得到如下表格:
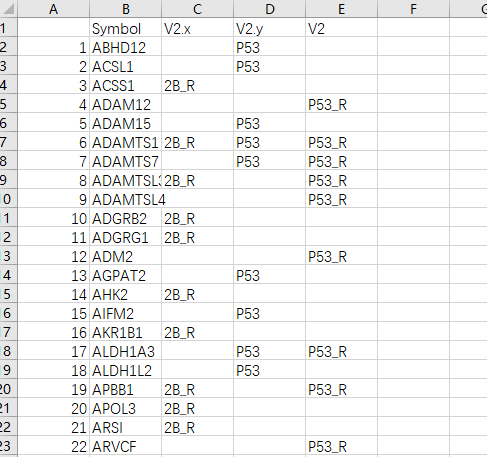
使用Excel函数连接三个单元格C2&D2&E2,全部填充:
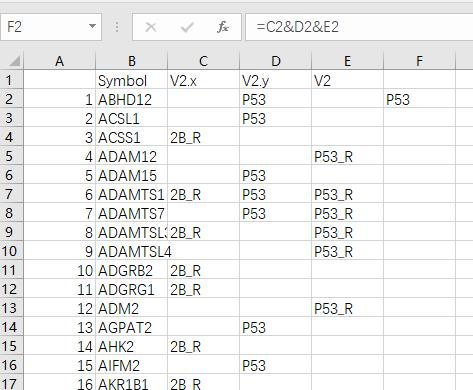
将新生成的这一列复制粘贴为数值,然后删除原来的三列,保存:
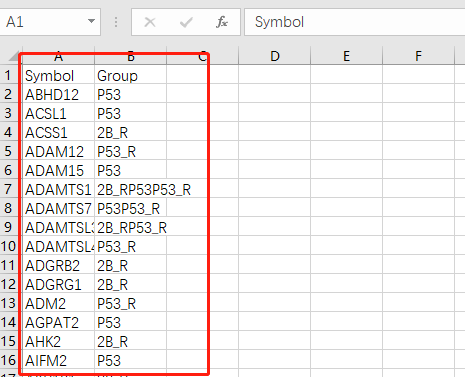
(4)然后就将上述生成的文件导入Cytoscape:
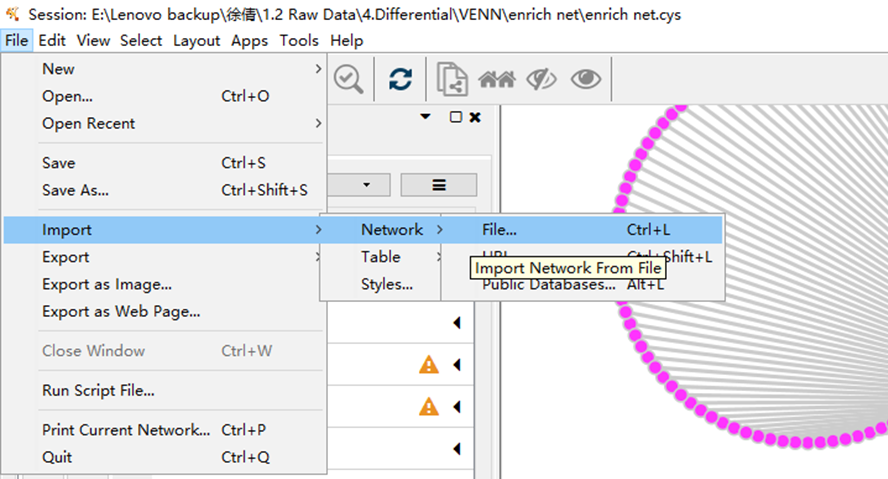
首先导入网络图文件(上述down.csv):

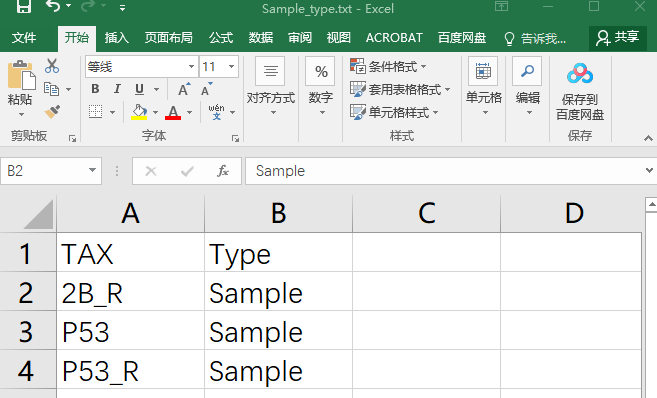
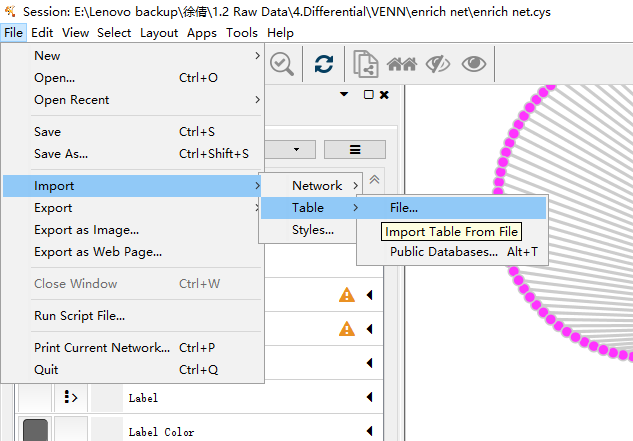
然后导入其余注释文件(上述down_group.csv以及另外一个文件Sample_type.txt):
(4)接下来,选择layout-Group attribute layout-Group:

(5)然后按照自己的要求拖拽,修改颜色(Ctrl+鼠标左键多选):
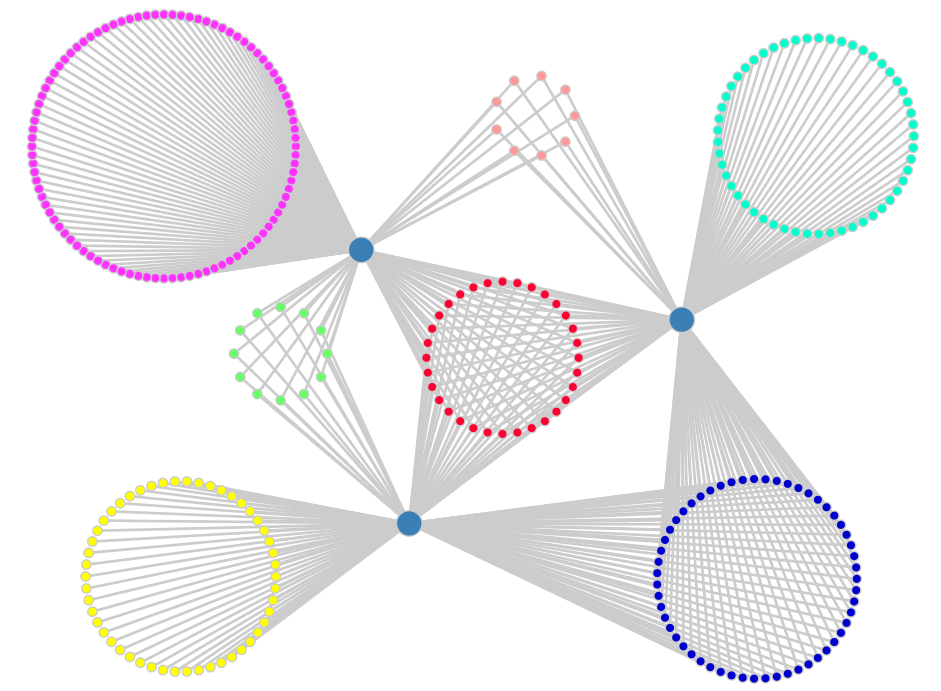
教程比较简洁,主要用于本人方法整理总结,如有问题欢迎留言讨论。
老师您好,请问三组数据(比如微生物组,代谢组,以及生化指标)如何绘制相关性网络图?
谢谢!
我完全按照你的格式整理了数据格式,请问第二个sample_type是干什么用的,为什么我导入之后在layout group那一列没法按照group来排列
您好,P53[,2]<-"P53" 和后面的P53_R[,2]<-"P53_R",没太看懂,而且用了您的代码和您的源文件无法重现如图所示的文件。
你好,这一篇写的忒乱,我已经修改了,第一个文件目的是将原本的短数据变成长数据,就是两列,第一列基因名字,第二列组别,把三组的合并到一起。P53[,2]<-"P53"就是提取出P53这一组数据之后第二列赋值P53。其他组同理,然后用rbind合并三组数据,至此第一个文件形成。第二个文件用merge合并,目的看同一个基因出现在哪几组。R语言代码可能是黏贴的时候乱掉了,目的比较简单
多谢,看您的简历也是苏大的:)
客气了,我是苏大公卫毒理学