TIMER是交互式web工具,能够全面、灵活地分析肿瘤浸润免疫细胞并可视化,采用反褶积方法从TCGA中不同癌症类型样本的基因表达谱中推断肿瘤浸润免疫细胞(B细胞、CD4+T细胞、CD8+T细胞、巨噬细胞、中性粒细胞和树突状细胞)的丰度。如今,TIMER升级到了TIMER2.0版本(http://timer.cistrome.org/)。主要包含Immune,Exploration, Estimation三个方面。
由于网站不稳定,时常打不开,在此留下网站TCGA分析好的泛癌免疫浸润数据链接:
https://pan.baidu.com/s/1m-EW31TELZrXeZyJH9XmJg?pwd=6fjq
提取码:6fjq

Part1:Immune
1.1 Gene–免疫浸润与基因表达之间的相关性
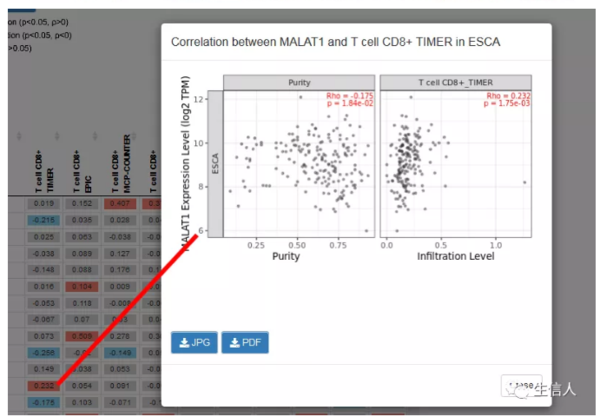
提交感兴趣的基因和免疫细胞类型后,结果图表中会将展示各种癌症类型中该基因和免疫细胞类型的spearman相关性的热图,并且点击相关感兴趣的单元格还可以展示出相关的散点图。

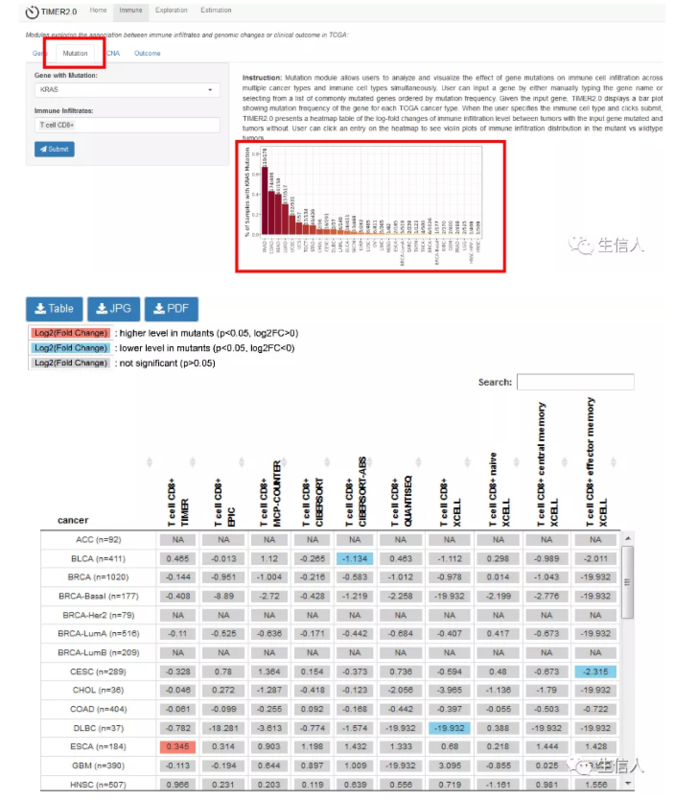
1.2 Mutation–免疫浸润与突变状态之间的相关性
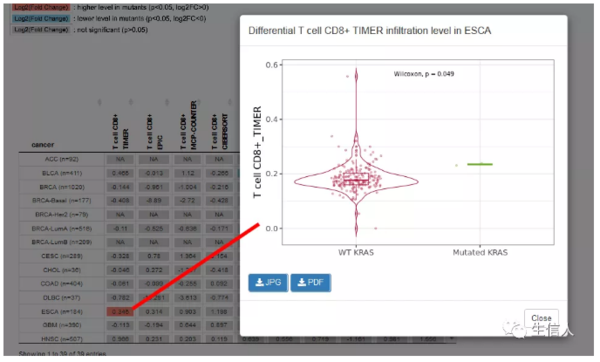
在输入感兴趣的基因后,右侧会先采用条形图展示该基因在各个癌症类型的基因突变频率。再选择免疫细胞类型后,结果图表中展示输入基因突变的癌症与没有突变的癌症之间的免疫浸润水平的对数倍变化的热图,并且点击相关感兴趣的单元格还可以展示出在突变体与野生型中免疫浸润分布的小提琴图。
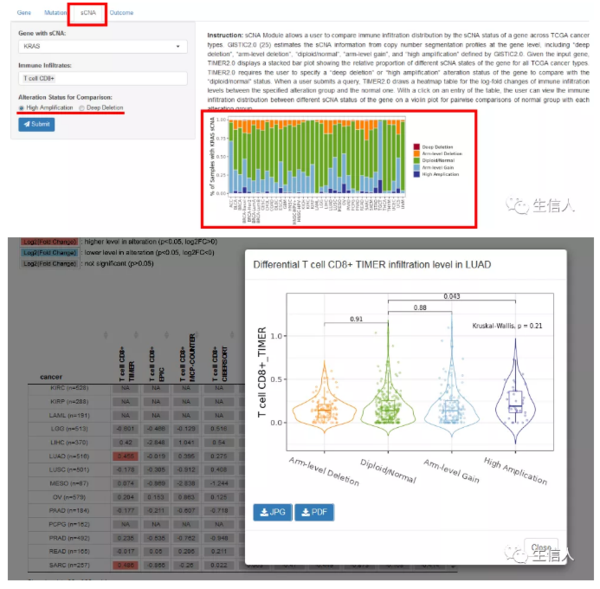
1.3 sCNA–免疫浸润与体细胞CNV之间的相关性
输入感兴趣的基因后,右侧会先采用堆积条形图展示该基因在所有癌症类型中的不同sCNA状态的相对比例。再选择免疫细胞类型和改变状态(深度缺失或高扩增)后,结果图表中展示不同sCNA状态下的免疫浸润分布的差别。
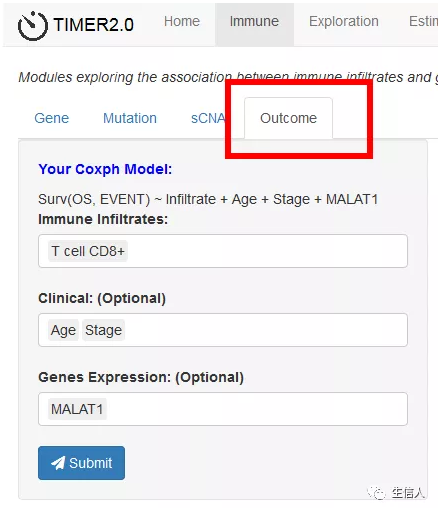
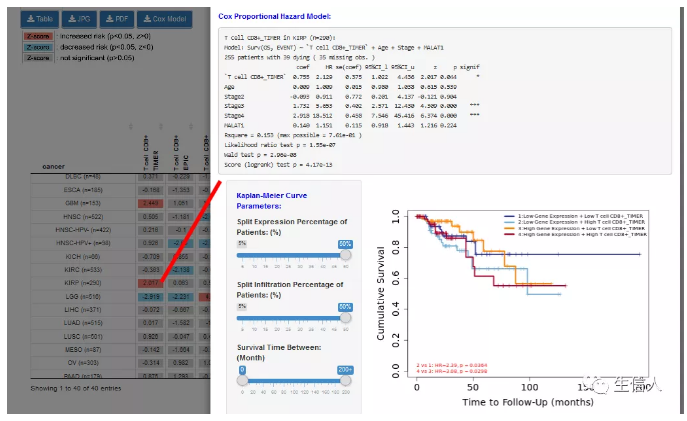
1.4 Outcome–免疫浸润与临床结果之间的相关性
输入感兴趣的免疫细胞类型、临床信息和基因,TIMER将进行cox回归分析,图表中展示每个模型的标准化浸润系数,点击每个单元格都对应一个独立的Cox模型。
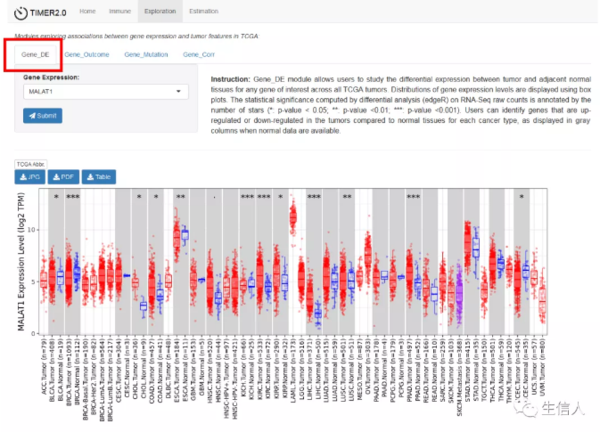
Part2:Exploration2.1 Gene_DE–癌症与正常人之间的差异基因表达
输入感兴趣的基因,识别在各个癌症类型中正常组织和癌症组织的差异表达基因。
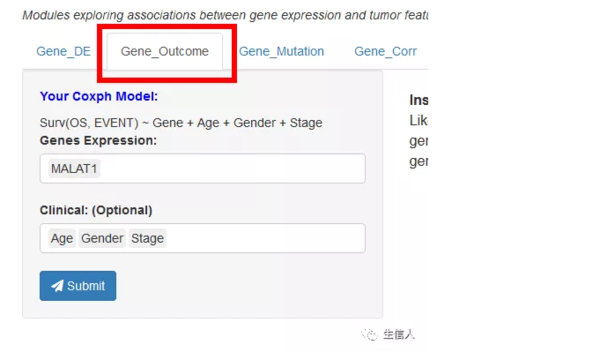
2.2 Gene_Outcome–基因表达与临床结果之间的关联
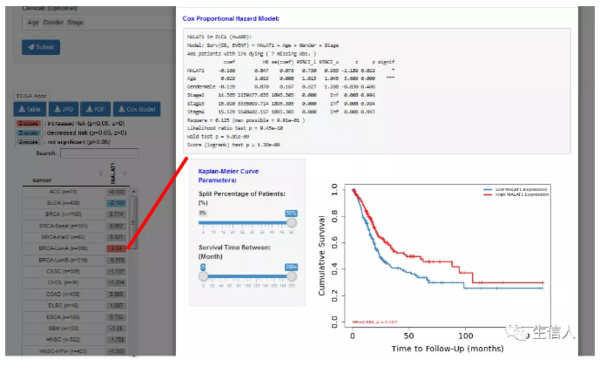
输入感兴趣的基因和临床信息,图表中展示使用Cox比例风险模型评估各肿瘤类型之间基因表达的临床相关性,点击每个单元格都对应一个独立的Cox模型和基因的KM曲线。
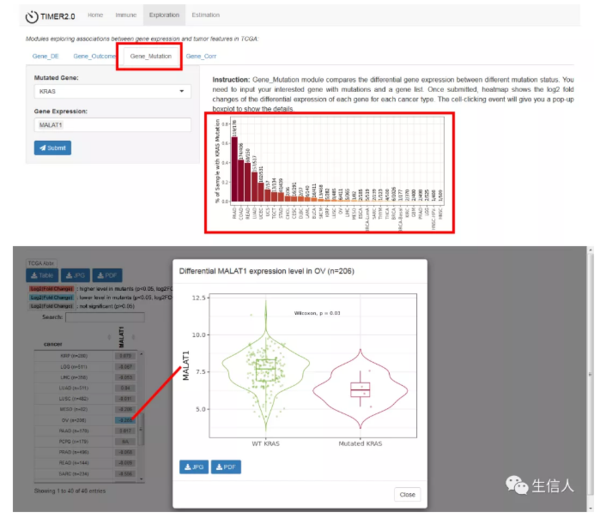
2.3 Gene_Mutation–基因突变状态之间的差异基因表达
输入感兴趣的突变基因和基因(基因集),结果图表显示每种肿瘤类型中每个基因差异表达的变化,点击每个单元格对应该基因在野生/突变型基因的差异表达水平的小提琴图。
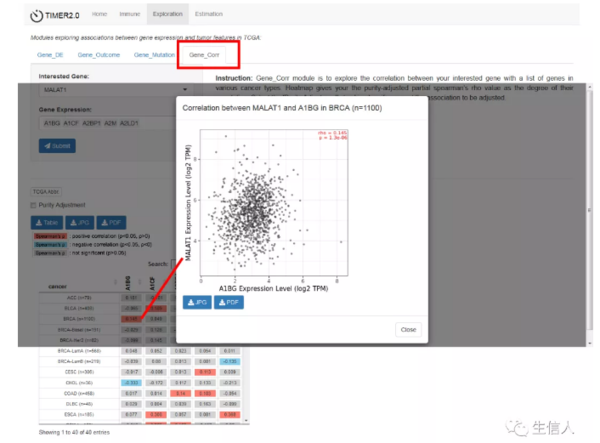
2.4 Gene_Corr–基因之间的相关性
输入感兴趣的基因和基因集,结果图表显示每种肿瘤类型中基因间的spearman相关性,点击每个单元格对应两基因相关性的散点图。
Part3:Estimation
该功能模块用于对用户提供的表达数据进行免疫浸润评估,需要输入表达谱矩阵,选择物种及癌症类型。结果图表分别显示免疫细胞在各样本中的丰度值、样本间的免疫细胞浸润水平和每个样本中免疫细胞比例。
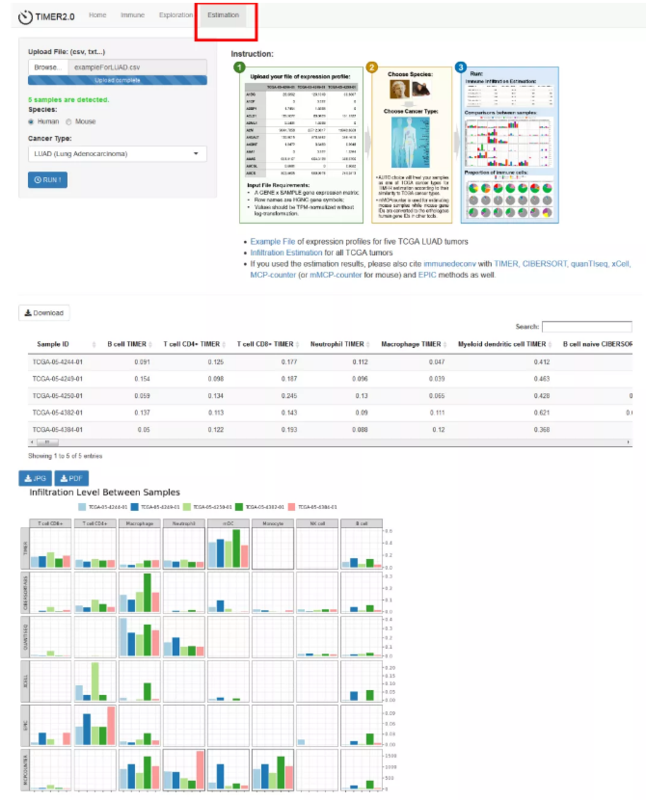
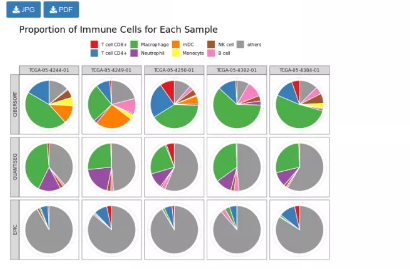
这么易操作的TIMER2.0还不赶快使用起来~
转自生信人:
进博士,请问我点两个基因的相关性分析,但是感兴趣基因那里下拉下来只有A打头的基因,是为什么呢
大佬您好,我也想要一份Timer上的TCGA数据库免疫浸润数据,方便发下吗?
请问师兄,我想要知道不同癌症患者免疫细胞的浸润情况,可以用这个网站分析吗?我之找到了与生存期相关的界面
请问如果我想用从TCGA下载自行合并的数据进行免疫浸润分析,是不是只能“protein-coding”类型的基因呢?
进哥,可以向你要Exploration2.1 Gene_DE中的GATA1的数据吗,可以发下邮箱吗,谢谢
王博士,你好,是否能将Timer上的TCGA数据库免疫浸润数据分享一份给我呢,想自己分析一下,谢谢~
你好王老师,我也想要TCGA肿瘤免疫浸润的数据,可以发一下吗,感谢~
你好王老师,请问Timer上的TCGA数据库免疫浸润数据是多少种癌症的呀,可以发我一份吗,邮箱是wangjunpupu@163.com,万分感谢~
您好,把您b站上的视频都看了,请问您会出这些视频教学嘛?比如:您这个免疫浸润数据处理得到的?还有后面视频怎么拆分的33种肿瘤什么的,因为您给的现成的,所以想学习了解一下数据处理的步骤
你好博主,想要肿瘤免疫浸润的数据,可以发邮箱吗,感谢
quchaolove@163.com
已发
请问 怎么加您微信
进哥您好,看了您B站的泛癌单基因免疫浸润分析和可视化的教学视频,但是在您这个网站上没找到对应的数据和代码,请问您能方便分享一下么,十分感谢~
您好,在这儿呦!
B站代码获取 – 王进的个人网站
https://www.jingege.wang/bilibili-code/
明白了,感谢解答!
师兄您好,紧急求教!请问根据肿瘤纯度分析两基因之间的相关性得到一个四象限图,图中有两个cor 和一个partial.cor,请问这个图该怎么理解呢,最后结果是partial.cor吗?希望看到后能回复,非常感谢!
您好,意思是计算相关性的时候根据肿瘤purity标准化了?如果需要考虑肿瘤纯度这个变量,最终要看的是partial.cor,叫偏相关性系数,是矫正肿瘤纯度的相关性
王老师,可以加您微信分享一下TCGA免疫浸润数据信息吗?最近正好在做这方面的东西苦于找不到TIMER的原始数据,如果可以就太感谢了
现在TIMER2.0是不是进不去,一直进不去,显示服务不可用 ,:服务器暂时无法为您的服务 由于维护停机时间或容量而请求 问题。请稍后重试。最近是在维护停机期间吗??
不清楚,我这儿有Timer上的TCGA数据库免疫浸润数据,需要的话发你
王博士,你好,是否能将Timer上的TCGA数据库免疫浸润数据分享一份给我呢,想自己分析一下,谢谢~
没问题 已发送到这个邮箱
非常感谢~
您好,我也想要一份Timer上的TCGA数据库免疫浸润数据,方便发下吗?
ok 没问题
TIMER2.0完全进不去
有一个可以 就是有点慢,实在不行用R语言自行分析,我有timer下载下来的数据,需要的话发你
王老师,timer数据怎么找您下载呢?这边实在登录不上去了
您好 加我微信吧 我发你
http://timer.comp-genomics.org/最近也进不去了,咋回事?
用这个吧http://timer.cistrome.org/
另一个可能服务器有问题,你也可以把上面数据下载下来自己分析 更方便
这两个现在都进不去,太难了
第一个可以 只是比较慢 可能需要使用VPN
或者实在不行你需要什么分析加我微信 我帮你分析一下
TIMER2 网站现在为什么进不去?要如何解决
可以访问,可能有点不稳定http://timer.cistrome.org/;不行的话试试这个网址http://timer.comp-genomics.org/国内访问快一点
好的,感谢您的解答。
请问师兄,那个感兴趣的基因可以是lncRNA吗 我输入我想要分析的lncRNA之后没有数据?
你好 Timer不支持非编码RNA,不过你可以下载作者计算好的免疫细胞浸润数据,与你的lncRNA进行pearson相关性分析
你好,请问一下非编码RNA相关的免疫细胞浸润数据在哪里下载,正在做LncRNA的实验设计但是证据不足,请问能指导一下吗,谢谢啦
你好,刚刚看见留言,请问现在明白了吗?不管研究编码还是非编码,免疫细胞浸润分析一样的,这个免疫浸润的评分是针对样本的,是根据各个免疫细胞特征基因计算得到的
谢谢您的回复,可能我研究的这个lncRNA比较新,好多数据库都找不到信息,timer2.0也用不了,我自己再琢磨一下。