一、什么是TIDE?
TIDE(http://tide.dfci.harvard.edu/)代表肿瘤免疫功能障碍和排斥。它是一个计算框架,用于评估肿瘤样本基因表达谱中肿瘤免疫逃逸的可能性。该工具主要有以下功能模块:
预测反应(Predict Response):为每个肿瘤样本计算的TIDE评分可以作为替代生物标记物,预测对免疫检查点阻断的反应,包括黑色素瘤和NSCLC的抗PD1和抗CTLA4。
查询基因(Query Gene):潮汐信号中得分较高的基因也显示了肿瘤免疫逃逸和癌症免疫治疗抵抗的潜在调节因子。
二、响应预测的输入数据格式是什么?
输入数据应为所有患者基因表达谱的平方矩阵。每列代表患者ID,每行代表一个基因名称,可以是符号名称(如TGFB1)或Entrez ID(如7040)。请看官网提供的案例:一些黑色素瘤抗PD1或抗CTLA4疗法的样本。
注:基因表达值应针对对照样本进行标准化,对照样本可以是与癌症类型相关的正常组织,也可以是来自不同肿瘤样本的混合样本。
PS:参考文献Signatures of T cell dysfunction and exclusion predict cancer immunotherapy response。The expression values of all genes are normalized by subtracting the mean values across all samples in a data set。
RNA-seq实验的log2(RPKM+1)值可能没有意义,除非有一个良好的参考对照可用于调整批次效应和癌症类型差异。在我们的研究中,我们使用每个研究中的所有样本平均值作为标准化对照。
预测反应(Predict Response)
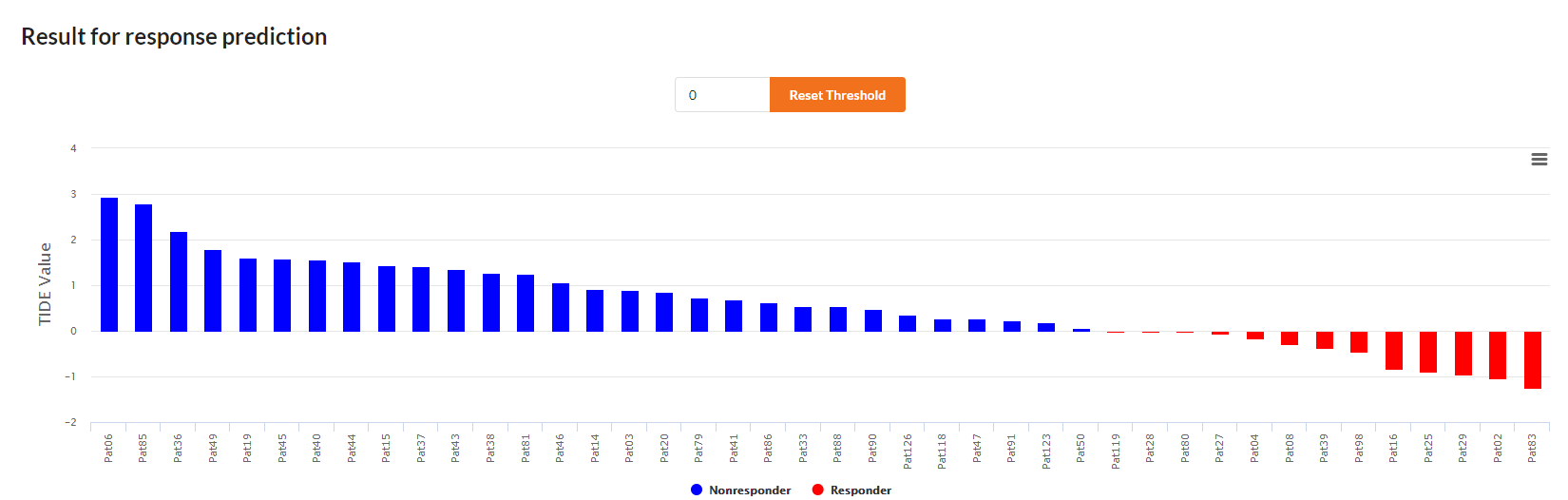
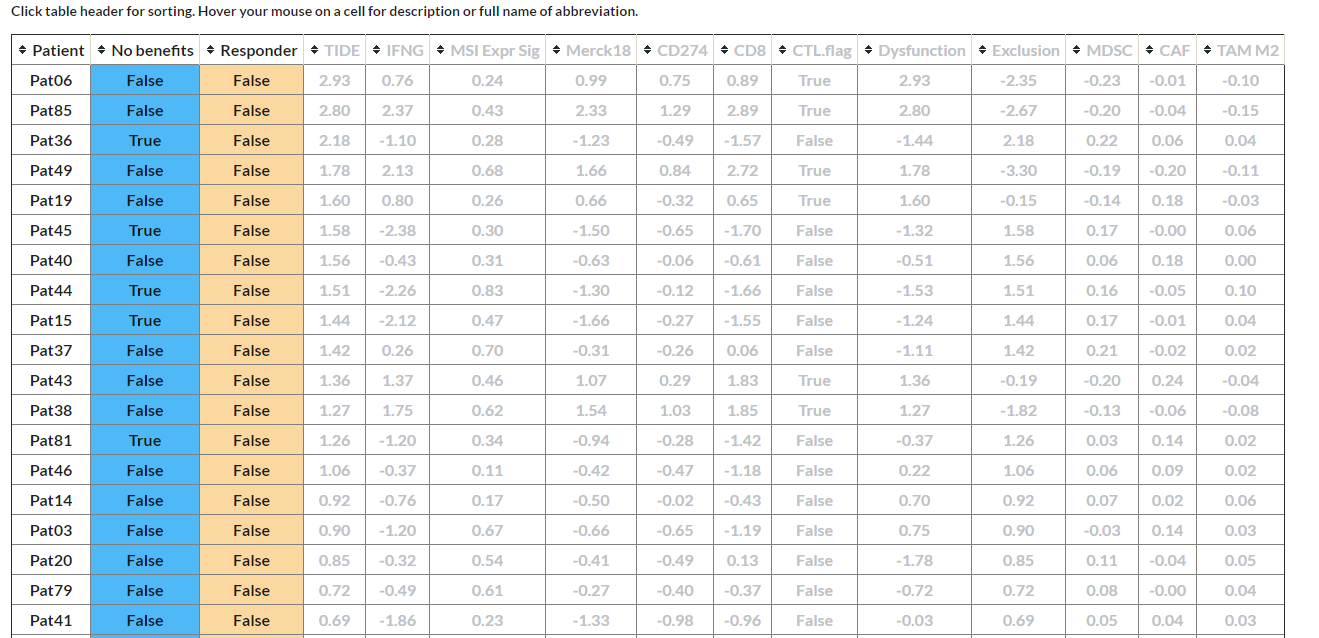
结果中报告了三种限制肿瘤中T细胞浸润的细胞类型,即肿瘤相关成纤维细胞(CAF)、髓源性抑制细胞(MDSCs)和肿瘤相关巨噬细胞(TAMs)的M2亚型。
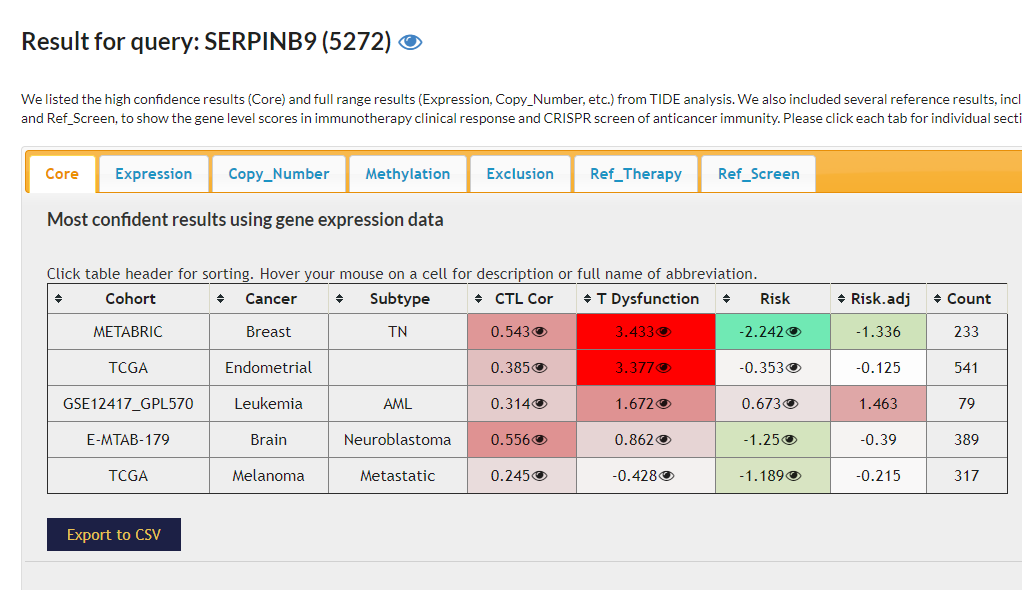
查询基因(Query Gene)
此外,网站提供了TCGA和一些GEO数据集已经分析好的结果,可供用户检索。
此外,网站还可以下载所有作者团队对TCGA、PrecG和METABRIC队列的分析结果。
如需了解更多,请参考作者文章:
- Large-scale public data reuse to model immunotherapy response and resistance. Jingxin Fu, Karen Li, Wubing Zhang, Changxin Wan, Jing Zhang§, Peng Jiang§, X. Shirley Liu§
Genome Medicine, 2020. PubMed | Read the paper | PDF
- Signatures of T-cell dysfunction and exclusion predict cancer immunotherapy response. Peng Jiang*, Shengqing Gu*, Deng Pan*, Jingxin Fu, Avinash Sahu, Xihao Hu, Ziyi Li, Nicole Traugh, Xia Bu, Bo Li, Jun Liu, Gordon J. Freeman, Myles A. Brown, Kai W. Wucherpfennig§, X. Shirley Liu§
Nature Medicine, 2018. PubMed | Read the paper | PDF | F1000 Prime Highlight
你好,想问一下,我用R保存的txt文件,write.table(Expr, file = ‘allGene_new_TIDE_cesc.txt’, sep = “\t”, quote = F, row.names = T) ,但是跑TIDE的时候一直显示“Please check the delimiter of your file. We only accept file with TAB delimiter”,这是为什么呢,实在找不到解决办法
进哥请问这个文件在哪呀,我一直找不到
为什么我提交后总是提示:
Page unavailable
Sorry, but the requested page is unavailable due to a server hiccup.
Our engineers have been notified, so check back later.
Sorry for this inconvenience.
是这个服务器停止服务了吗?
应该是的 回头再试一下看看
您好,现在还是这样的提示,该怎么办呀?
请问得到解决了吗?现在这个网站还是没办法运行
这个网页现在还是打不开,咋办。
数据提交之后,网页就报错,进不去。
如果使用GEO芯片数据进行TIDE,那么芯片数据需要取log吗?还是说按照正常limma方法判断完log,以及normalizeBetweenArrays标准化,得到的这个表达矩阵再输入。
具体我也不是很清楚,应该芯片数据没有特殊要求 可以拿你GEO下载的数据直接进行分析,没有log的建议还是log一下,不然数据偏态分布
看了视频感觉有点没懂,视频1:35说不建议使用测序实验的log2(RPKM+1),然后视频1:42建议转化成log2(TPM+1)或log2(FPKM+1)然后再减去所有样本平均值,请问数据是能够用log2之后的吗,还有用TPM值是不是也可以?
你好,用log(TPM/FPKM+1),不用RPKM,数据都进行一下log转换吧
大佬,我注册收不到验证码,请问可以借我账号用下么?非常感谢!
加我微信吧 18021308280
你好,大佬,想咨询一下我是用TCGA和gtex联合数据做的生信分析,是不是不能用TIDE进行计算啊,做肿瘤免疫逃逸还有其他的方法吗?
不明白什么意思,TIDE只需要肿瘤组织数据,用不着GTEx和TCGA中的normal样本
意思是输入数据的矩阵文件里面只需要癌症病人的信息就可以了?一句话点醒我,感谢大佬
老师是不是输入基因表达举证就可以了,还有我做其他癌肿也可以?
may not work on cancer types other than melanoma and NSCLC (e.g., glioblastoma, or renal cell carcinoma)
这是网站说明,主要是针对黑色素瘤和非小细胞肺癌,其它类型只能稍作参考
对于输入数据,基因表达矩阵即可
你好,王老师,我也注册不了,能用用你的邮箱吗,非常感谢
可以的 如果还需要加我微信吧
进哥,您好!想问一下我从Xena上下载的fpkm数据是经过log化的 我可以拿它按照TIDE网站的方法直接做标准化,然后上传TIDE网站吗?
你好 按照网站说明 是不建议使用log之后的数据的,除非有一个很好的对照,建议使用时还是将其变回fpkm,在进行标准化 计算
不过TCGA数据网站都有提供计算好的 你可以直接下载计算好的结果
进哥,FPKM,不就是标准化的数据吗?还需要进一步标准化吗
你好,因为TIDE算法的要求 需要进一步数据标准化 详见网站说明
进哥请问那个文件在哪呀,我找不到
进哥您好,请问可以借账号用下么?我也注册不了。
您好 没问题 加我微信发你
王老师您好,请问可以借您的账号用吗?注册不了
你好 加我微信 发你
大佬好,方便加您个联系方式吗?
哈哈,荣幸 网页导航栏有我的简历 和 联系 都有微信和邮箱
大神 请问除了在线提交,还有其他方法可以做嘛?在线提交显示服务器错误……
sadly,作者并没有开放代码,可能他们在做网站工具和数据库整合,不太稳定,过两天看看
python也可以,可以试试,我网页搞了n久老是报错,用了python,挺快的
棒,需要的可以试试,谢谢分享
请问您用python怎么操作的啊?
我来回复一下
tide网站有提供:GitHub – liulab-dfci/TIDEpy: TIDE (Tumor Immune Dysfunction and Exclusion), a gene expression biomarker to predict the clinical response to immune checkpoint blockade in Python.
您好,我也是遇到了收不到验证码的情况,可以借用您的账号吗?
已发送至邮箱
十分感谢
呜呜呜,同样收不到验证码,可以借用一下您的账号吗
已发送至邮箱,请查收
十分感谢!
大神你好,我想分析TCGA一个癌症病种队列的数据,如你文中所说,我上传了log2(FPKM+1)数据,还有基因的表达差异倍数,结果都分析不出来,我又上传了counts数据,可以分析出来,但是结果tide score基本全是负值,并且有免疫应答的结果,请问这种分析TCGA数据的,到底上传什么数据是正确的?谢谢
您好,网站首页下面有介绍如何标准化,不建议log转换,你可以先看看,不懂的话加微信讨论
您好,请问您负值这里是如何解释的?
您好,我也是遇到了各种邮箱都收不到验证码的情况,可以借用您的账号吗?
已发至您邮箱,查收
非常感谢您!
进哥,我也遇到了注册账号收不到验证码的问题,连gmail都不行,可以借账号用一下吗,非常感谢!
已发您邮箱 查收
多谢多谢!
您好 想请问一下TIDE 网站现在为什么连登录界面都打不开呢 ? 一直是 404 not found,,之前还能用的
这个实验室好像在整合他们的在线工具,tide服务器可能还没有对接好,过几天试试吧
哈哈明白啦 谢谢!
今天可以进去了 你试试
多谢进哥提醒!!下午用IE浏览器登进去了,但是现在好像又在维护
我尝试了各种邮箱都收不到验证码,也想借用一下账号,非常感谢!
已发送至您邮箱
我的邮箱也收不到验证码,请问可以借我一下账号吗,非常感谢!
已经邮箱发您了,有需要再讨论
试了各种邮箱和浏览器,就是收不到验证码,大神,求一个账号,非常谢谢!
您好,已发您邮箱,有需要再讨论
你的微信号码是什么呀,可以借我账号下载吗,感谢进哥哥!
您好 微信号就是手机号,我的简历里面有 18021308280
请问可以借我账号用下么?我也进不去。
可以的 加我微信吧
进哥,也可以帮我帮我下载TIDE上的一些数据吗?拜托拜托
我不知道你要下载TIDE上的什么数据,如果需要 我可以借你我的账号
请问怎么登陆上的呢,一直无法注册,邮箱收不到验证码
确实,已经有几个同学联系我问同样的问题,可能是他们服务器邮件系统出现问题,也可能维护者故意限制用户数量。你加我微信吧 我借你我的账号
进哥,我也遇到了注册账号收不到验证码的问题,可以借账号用一下吗,非常感谢!
当然可以,没问题