CCLE数据库
CCLE(Cancer Cell Line Encyclopedia),它是Board Institute维护的一个癌症细胞系的数据库,目前有目前有1,400多个cell line。移步官网查看数据库说明:https://sites.broadinstitute.org/ccle

CCLE中细胞系表达谱的GEO数据库GSE36133
CCLE数据库数据下载链接

数据处理
样品信息处理
通过R语言来处理数据,需要一定R语言基础;
因为下载下来的数据包含了多种癌症,首先是处理细胞系信息,选择自己需要的细胞系,进行后续操作。
rm(list = ls())
sample<-read.csv(file="sample_info.csv")
colnames(sample)
# [1] "DepMap_ID" "cell_line_name"
# [3] "stripped_cell_line_name" "CCLE_Name"
# [5] "alias" "COSMICID"
# [7] "sex" "source"
# [9] "Achilles_n_replicates" "cell_line_NNMD"
# [11] "culture_type" "culture_medium"
# [13] "cas9_activity" "RRID"
# [15] "WTSI_Master_Cell_ID" "sample_collection_site"
# [17] "primary_or_metastasis" "primary_disease"
# [19] "Subtype" "age"
# [21] "Sanger_Model_ID" "depmap_public_comments"
# [23] "lineage" "lineage_subtype"
# [25] "lineage_sub_subtype" "lineage_molecular_subtype"
可以看到,样品信息包含了以上26中信息,可以根据需要,选择信息进行后续处理,比如说,我选择样品ID,细胞系名称,原发灶或转移灶,原发疾病类型,亚型等信息。
sample_info<-sample[,c(1,3,17,18,19)]
###choose CRC cell lines
unique(sample_info$primary_disease)
# [1] "Ovarian Cancer" "Leukemia"
# [3] "Colon/Colorectal Cancer" "Skin Cancer"
# [5] "Lung Cancer" "Bladder Cancer"
# [7] "Kidney Cancer" "Breast Cancer"
# [9] "Pancreatic Cancer" "Myeloma"
# [11] "Brain Cancer" "Sarcoma"
# [13] "Lymphoma" "Bone Cancer"
# [15] "Fibroblast" "Gastric Cancer"
# [17] "Engineered" "Thyroid Cancer"
# [19] "Neuroblastoma" "Prostate Cancer"
# [21] "Rhabdoid" "Gallbladder Cancer"
# [23] "Endometrial/Uterine Cancer" "Head and Neck Cancer"
# [25] "Bile Duct Cancer" "Esophageal Cancer"
# [27] "Liver Cancer" "Cervical Cancer"
# [29] "Unknown" "Eye Cancer"
# [31] "Adrenal Cancer" "Liposarcoma"
# [33] "Embryonal Cancer" "Teratoma"
# [35] "Non-Cancerous"
可以看到有35种不同的癌症类型,我们选择特定的一种即可,比如我选择肝癌。
which(sample_info$primary_disease=="Liver Cancer")
cell_lines<-sample_info[which(sample_info$primary_disease=="Liver Cancer"),]
save(cell_lines,sample,file="Data1_sample_information.Rdata")
这样我们就选择了我们想研究的癌症类型及需要的细胞系名称及相关信息,先保存下来。
基因表达信息
先读取我们下载的表达信息
exp<-read.csv(file="CCLE_expression.csv")
rownames(exp)<-exp[,1]
exp[1:3,1:3]
exp<-exp[,-1]
# TSPAN6..7105. TNMD..64102. DPM1..8813.
# ACH-001113 4.990501 0.0000000 7.273702
# ACH-001289 5.209843 0.5459684 7.070604
# ACH-001339 3.779260 0.0000000 7.346425
##choose samples from expression matrix
a<-cell_lines$DepMap_ID
##%in%判断exp中的元素是否都在我们选择的细胞系中
b<-c(rownames(exp) %in% a)
length(b)
exp_liver<-exp[b,]
##判断细胞系信息中细胞名称的元素是否在肝癌细胞系exp中
c<-c(cell_lines$DepMap_ID %in% rownames(exp_liver))
cell_lines<-cell_lines[c,]
现在我们就得到了我们需要的表达矩阵,但是并没有对应细胞名,我们要把ID和对应名称匹配
rownames(cell_lines)<-cell_lines[,1]
merge<-cbind(cell_lines,exp_liver)
save(merge,file="input_sample_and_exp.Rdata")
rownames(merge)<-merge$stripped_cell_line_name
matrix<-merge[,-c(1:5)]
matrix<-t(matrix)
d<-rownames(matrix)
class(d)
d<-as.matrix(d)
matrix<-cbind(d,matrix)
write.csv(matrix,file="CRC_exp.csv")
matrix<- read.csv(file="CRC_exp.csv")
去除NA值,整理表达矩阵
sum(is.na(matrix))
newdata<-na.omit(matrix)
duplicated(newdata$X.1)
h<-newdata[duplicated(newdata$X.1),]
mydata<-newdata[!duplicated(newdata$X.1),]
rownames(mydata)<-mydata$X.1
mydata<-mydata[,-c(1:3)]
原文链接:https://blog.csdn.net/Eric_blog/article/details/119089696
您好,不好意思,我不知道您下载的是哪两个文件,请问可以指导一下吗?
为啥我下载不了这两个文件呀 进哥哥.求帮忙
你好进哥哥,请问CCLE的数据可以看到某个细胞系的全部转录表达情况吗?生信小白求助
老师你好,请问现在如何下载肿瘤细胞系的表达谱数据,我看教程里读取的csv文件在官网已经找不到了
有些基因找不到是因为什么呀,比如FAM231A( GeneCards Symbol: LOC124903857 3
FAM231A/C-Like Protein LOC102723383 NCBI Gene: 124903857; Ensembl: ENSG00000268674)
您好,因为你查询的这个基因是一个novel gene, 相关注释信息还不全
进哥哥你好,请问这里面有小鼠的细胞系吗?比如乳腺癌小鼠细胞系4T1和EMT6,我在CCLE中没有找到。不知道小鼠相关的数据在哪里能找到呢?
没有诶 不好意思
请问你找到了类似的数据库吗,我也需要查找小鼠相关的数据
想知道+1
想知道+1
老师 我下载数据后,超过几是高表达?小于几是低表达呢?
高低表达是相对的,你可以根据median、mean、Q1\Q4区分高低
老师您好,请问CCLE数据库中怎么下载可变剪接的数据呀,谢谢老师
您好,这个不清楚诶 网站提供了转录本数据CCLE_RNAseq_transcripts.csv,不知道您可不可以用?
王老师您好,我想请教一下,可以用这个数据分析同一癌种不同细胞系之间的差异表达吗?
是的 可以的 可以分析不同细胞中基因表达差异
王老师您好,我想问一下因为CCLE里没有normal 的样本数据,那我可以用TCGA里的normal数据和CCLE里是数据做差异分析吗?
不好意思 忘记回复了
这个当然不可以了 一个是组织一个是细胞,当然不能一起分析
好的,谢谢老师
进哥,请问CCLE数据库里的细胞是肿瘤细胞还是正常细胞也有啊
进哥哥,ccle有rna可变剪接的数据,下载路径一样吗
因为CCLE有过大更新,现在的组学数据更全面了以往的下载方法只能分析基因表达,有点大材小用了。
老师您好,我想对比CCLE中肠癌细胞和正常细胞中,有哪些基因表达有差异,类似于TCGA数据库进行的正常组织肿瘤组织差异分析。请问您觉得正常细胞选取那些细胞系比较好呢?谢谢您~~
您好,CCLE中的细胞样本都是cancer cell,其中colon cancer 70种,没有正常细胞哇,并不合适做差异分析
哇,谢谢老师回复。那细胞的差异分析怎么办呢?
CCLE不合适,可以检索GEO数据库,应该会相关细胞高通量数据
你好,我想请教一下,这个库CCLE的每个细胞系的表达和拷贝数水平一个蛋白或者一个基因对应只一个数据,如果去做不同细胞系比对的话,有可比性吗或者有参考价值吗?
你好,刚刚看到你的消息,我的理解是,你要统计比较肯定不合适,只能大概看一下一个基因在不同细胞中的表达情况,可以结合多组学一起看 用于细胞系筛选,后续可以实验验证一下。用得最多的还是看一种类型癌症中的分析,每个细胞就作为一个样本
王老师您好,我merge那一步出了问题,我是选择的leukemia,提示错误
> merge<-cbind(cell_lines,exp_leukemia)
Error in data.frame(…, check.names = FALSE) :
参数值意味着不同的行数: 137, 104
您好,不好意思,是我的疏忽贴错了一句代码,上面一句代码错了,文中已经更正:
##%in%判断exp中的元素是否都在我们选择的细胞系中
b<-c(rownames(exp) %in% a) length(b) exp_liver<-exp[b,] ##判断细胞系信息中细胞名称的元素是否在肝癌细胞系exp中 c<-c(cell_lines$DepMap_ID %in% rownames(exp_liver)) cell_lines<-cell_lines[c,]
不过建议您理解每一句代码的含义,这样方便去debug没一个错误
谢谢您提出问题,文中已修改更新了
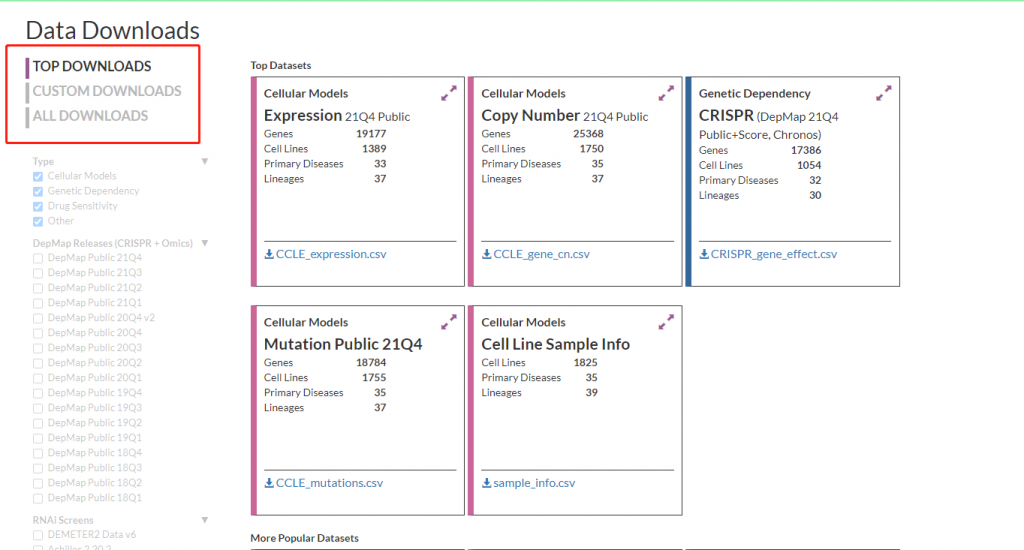
王老师您好,请问现在CCLE是不是升级啦?我找不到以前的界面了,新的界面里数据集必须要通过R语言下载吗?
你好 ,是的呢,网站改版了,下载链接文章中有https://depmap.org/portal/download/,我又添加了下载界面的截图,你看看,有问题在讨论
你好,不好意思,我是初学者,我不太看懂那些数据是什么意思,这些数据是RNA-seq的原始数据吗?同时,请问下载下来是什么格式
这些数据有很多类型的 RNA-seq的就是不同样本各个基因表达的数据,其它的比如甲基化拷贝数也一样 。count数据算是原始数据,一般我们下载的是标准化之后的数据 一般TPM 或者 fpkm,文件格式就是csv或者txt直接用Excel就可以打开 一般分析使用R语言
请问,我想下载tpm数据格式在哪里呀,没有找到呀,然后您分享的代码下载的又是哪个呢