GTEx全称如下:Genotype-Tissue Expression
该项目对来自人体多个组合和器官的样本,同时进行了转录组测序和基因分型分析,构建了一个组织特异性的基因表达和调控的数据库。网址如下
包含的组织类型和样本个数如下图所示
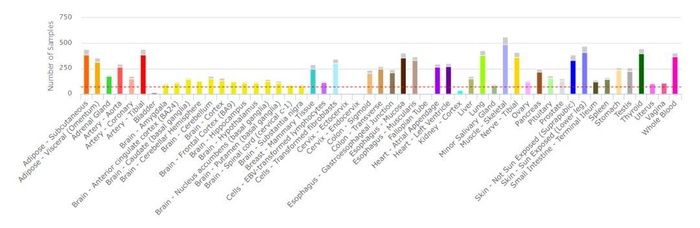
对于所有的样本,主要进行了以下三种分析
1. RNA seq
通过illumina Truseq试剂盒构建polyA+文库,采用Hiseq 2000/2500进行测序,对于下机数据,采用STAR进行比对,参照选择的是gencode V19版本的gtf文件,进行了以下3个level的定量
- gene-level,采用RNAseQC软件,对基因的raw count和TPM两种方式进行定量
- exon-level, 对exon的raw count进行定量
- transcript-level,采用RSEM进行转录本水平的定量
2. genotype
通过WGS对样本进行分型, 采用的是GATK germline variants calling的流程,步骤如下
- bwa-mem alignment
- picard markduplicate
- BQSR
- indel realign
- haplotypeCaller
3. eQTL
通过FastQTL软件进行cis-eQTL分析,将基因型和基因表达量进行关联。
通过官网可以查看基因表达量和eQTL分析的结果,以TP53为例,每个基因给出了以下3个层级的表达量
- Isoform Expression
- Exon Expression
- Junction Expression
分别对应转录本,外显子,剪切序列的表达量,对于不同组织中的表达量,以热图的形式进行展示,示意如下
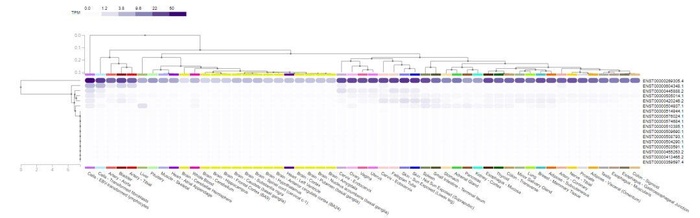
对于基因结构,也进行了可视化,示意如下
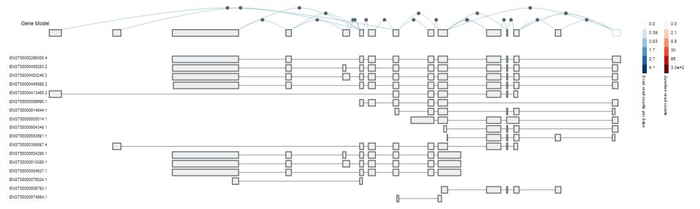
eQTL的结果示意如下
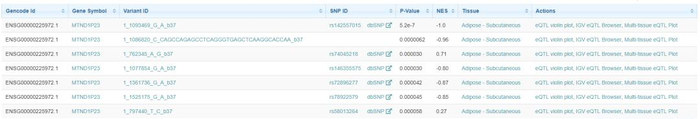
提供了以下两种可视化方式,第一种是在单个组织内的小提琴图,eQTL violin plot, 示意如下
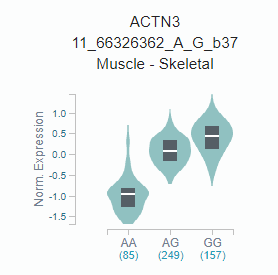
第二种用于多个组织间的比较,Multi-tissue eQTL plot, 示意如下
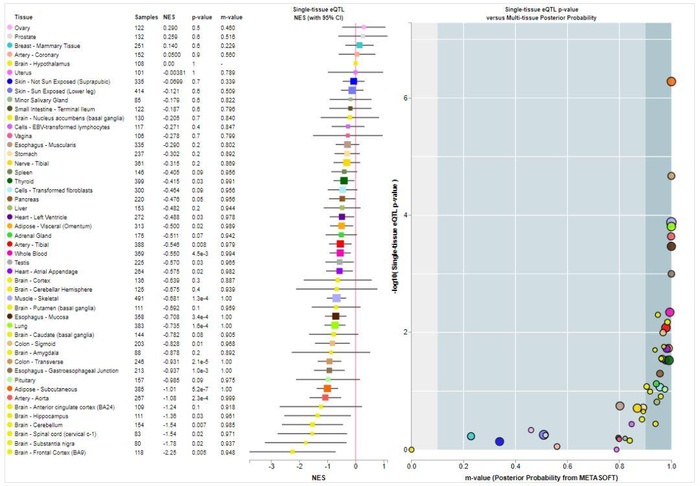
所有的分析结果可以通过官网进行下载,GTEx数据库不仅仅是一个正常组织的基因表达量数据库,其eQTL分析的策略更值得我们借鉴。
您好,请问GTEx数据库中,可以查看单个基因表达量的上调或者是下调吗