microRNA(miRNA)是生理和疾病中调控基因表达的关键因子,通过抑制mRNA翻译或促进降解介导靶基因的翻译抑制。miRNA翻译抑制的功能在过去的很多年被广泛研究,但miRNA自身的转录调控机制却缺乏有效的研究,其难点是miRNA启动子难以捕获。
miRNA启动子难以捕获???启动子不就是紧挨着转录起始位点(TSS)嘛,为什么说启动子难以捕获呢?小编见证了太多的科研者获取miRNA启动子时,直接延伸上游2k,这样对吗?
lncRNA、mRNA等分子获取启动子时,直接取TSS上游序列即为启动子,但此操作不适用miRNA,这要从miRNA的形成机制说起:前体pri-miRNA在基因组上转录,在细胞核进行第一次剪切,形成第二个前体pre-miRNA(长约70~120nt),pre转移至胞浆进行第二次剪切,形成成熟体Mature,也就是功能性的miRNA(长约20nt)。目前数据库只收录了pre以及Mature,而没有pri,但实际pri 5’端的第一个碱基才是TSS,由于pri未知,这就让miRNA启动子的获取变得困难,而直接取pre上游序列作为启动子肯定是不对的。
一、RACE实验获得准确的
pri-miRNA前体序列
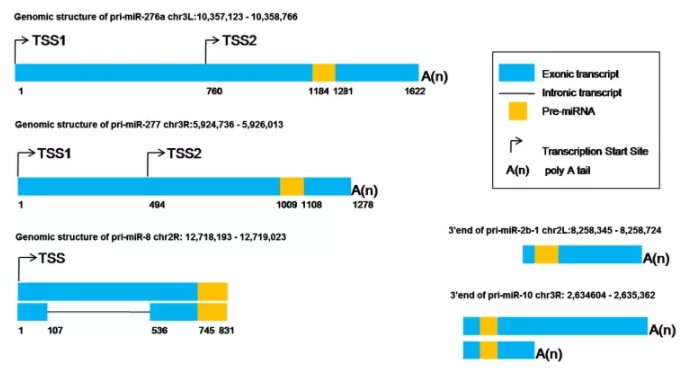
要想准确获得miRNA启动子,必须确定前体pri的序列,至少是5’端序列。因此,RACE实验是不二的选择。通过已知的pre设计引物,进行cDNA末端克隆,获得5’末端序列,此为TSS位点,TSS上游即为启动子序列。Science的一篇文献,通过RACE技术,成功扩增了多个果蝇pri(mir-276a,mir-277和mir-8)的5’端和3’端,并且还鉴定出pri-miR-276a和pri-miR-277有两个TSS3:
二、基于miRNA位置、算法的分析
虽然RACE是最严谨的做法,但不是每个科研工作者都有条件操作,那么可以根据miRNA位置,并结合一些预测类型的数据库进行分析、判断、验证。miRNA在基因座上存在三种位置情况:基因间(intergenic)、内含子内(intronic)、宿主基因内(host gene),miRNA位置信息可在NCBI-GENE(点击进入)主页的“Genomic context”及“Genomic regions, transcripts, and products”图中获得。
目前预测miRNA启动子的方法不外乎基于启动子保守性、组蛋白H3K4me3和DHS标记、TATA盒预测等方法。小编推荐三个网站分析(当然也是用):
1. 懂生信的可以借助PROmiRNA(基于CpG含量,保守性和TATA盒亲和力的算法)分析,基于DeepCAGE数据,预测了7244个miRNA TSS,并标注了与miRNA的距离;
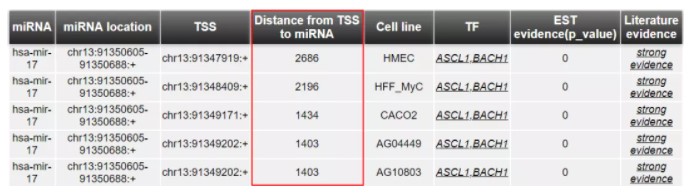
2. mirtrans 基于组蛋白修饰、启动子预测等算法预测miRNA启动子。使用网站时,直接输入miRNA的名称,预测TSS位置,如查询has-mir-17,网站给出了不同细胞系的起始位点不一致,并预测了转录因子,十分实用(对于TSS位点大于10k的,请谨慎参考):
3. 基于大部分pri前体在10k以内,则取pre上游5~10kb进行启动子分析,找出可能存在的启动子位置,再进行验证(点此)。小编输入人源MIR-183上游5k,分析出三个预测的启动子,其中距离pre 1400bp=(5000-3600)的位置启动子可能性最高。

而后,结合图三进行启动子截断,构建不同长度的启动子区段,通过双荧光素酶验证启动子具体座落位置即可。
请问现在 mirtrans 网站打不开是什么原因?
sorry,链接网址错了:http://123.207.219.84/mirtrans/
请问大佬,pre-miRNA序列在哪里可以查询。目前数据库好像都只能查询到人的miRNA启动子序列,没有小鼠的,不同细胞TSS也不一样,要怎么在小鼠上面进行筛选验证启动子结合位点呢
师兄,你好,预测到miRNA启动子后,如何通过双荧光素酶报告验证呢,找到启动子,目的一般是那些方面呢,;师兄你说的那篇Science文章有Doi嘛?
The full-length transcripts and promoter analysis of intergenic microRNAs in Drosophila melanogaster – PubMed
https://pubmed.ncbi.nlm.nih.gov/21333734/