sample<-read.table("E:/Study/cigarettes study/1.4 Analysed Data/GEO/106b.txt",header = TRUE)
方法一:
library(metafor)
metawsd=rma.uni(n1i=n1,n2i = n2,m1i = mean1,m2i=mean2,sd1i=sd1,sd2i = sd2,data=sample,measure = "MD",method="FE",slab=study)
forest(metawsd)
方法二:
library(meta)
metawsd=metacont(n1,mean1,sd1,n2,mean2,sd2,data=sample,sm="SMD",comb.fixed = FALSE,comb.random = TRUE,studlab = study)
forest(metawsd,digits.se =2,lab.e="NSCLC",lab.c = "Normal")
metabias(metawsd,k.min = 7)
funnel(metawsd,lwd = 2)
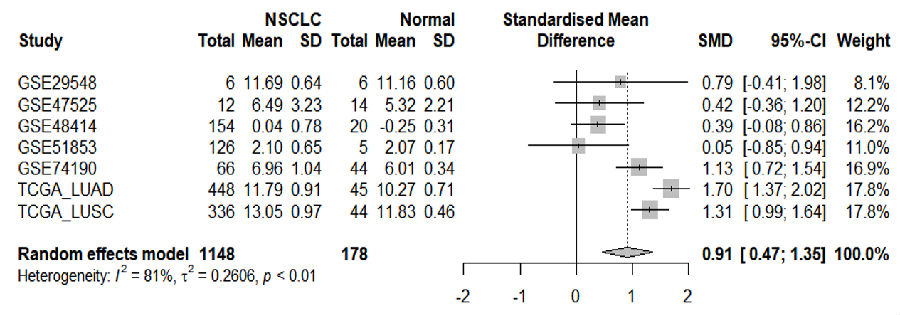
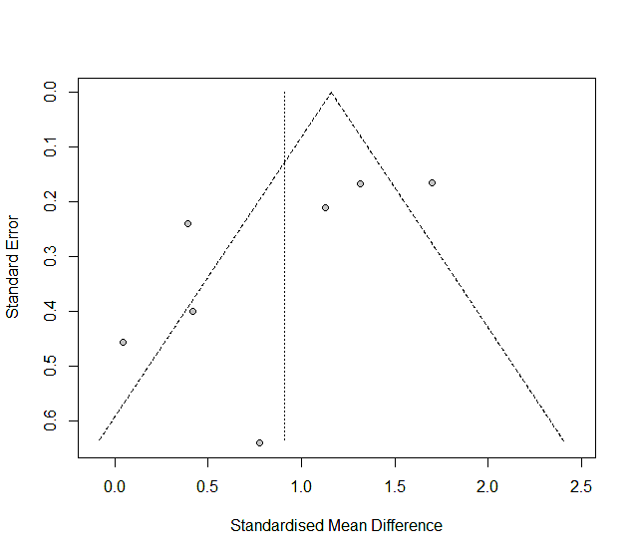
老师您好,请问106b.txt文件里面基因表达的均值和标准误怎么获得啊
您好,如示例数据文件,其实就是不同数据集中分析的对照和实验组样本的样本量n、平均值mean和标准差sd。可以直接用Excel计算得到
这种方法可能得到的异质性I2比较大,因为不同数据集表达量可能差别很大,可以把它们归一化处理之后分析
感谢!!!