TCGA(The Cancer Genome Atlas)GDC(Genomic Data Commons)data portal 2.0(https://portal.gdc.cancer.gov)是2024年全新推出的癌症基因组数据共享平台,此版本界面采用了最先进的Web技术,为用户提供了更加直观、互动性强的数据浏览体验。新版界面的设计注重用户友好性,以直观的信息布局和流畅的操作流程,大幅提升了数据检索的效率。
PS:如果大家不习惯2.0版本的界面,贴心的TCGA团队仍然保留了原来的GDC1.0版本:https://portal.gdc.cancer.gov/v1
PS:新版功能比较多,详细介绍请查看Release Notes – GDC Docs。
此处,咱们以常规数据下载流程为目的简要介绍:
- GDC Data Portal 2.0的标题包含常用链接和功能。

左上角是 GDC Data Portal 徽标,它链接到 GDC Data Portal 的主页。徽标下方是按以下顺序排列的链接:
• 分析中心:用于访问 GDC 数据门户中所有工具的中心枢纽;
• 项目:允许浏览 GDC 数据门户中的所有项目;
• 队列生成器:队列生成器工具由各种临床和生物样本过滤器组成,用于构建用于分析的自定义队列;
• 存储库:允许浏览与队列关联的文件。
2. 对于分析中心(Analysis center),网站提供了很多方便的分析功能,大家可以自行摸索。尤其对于单个基因或者少数基因数据的获取可以基于gene expression clustering获得,不用下载完整的数据。
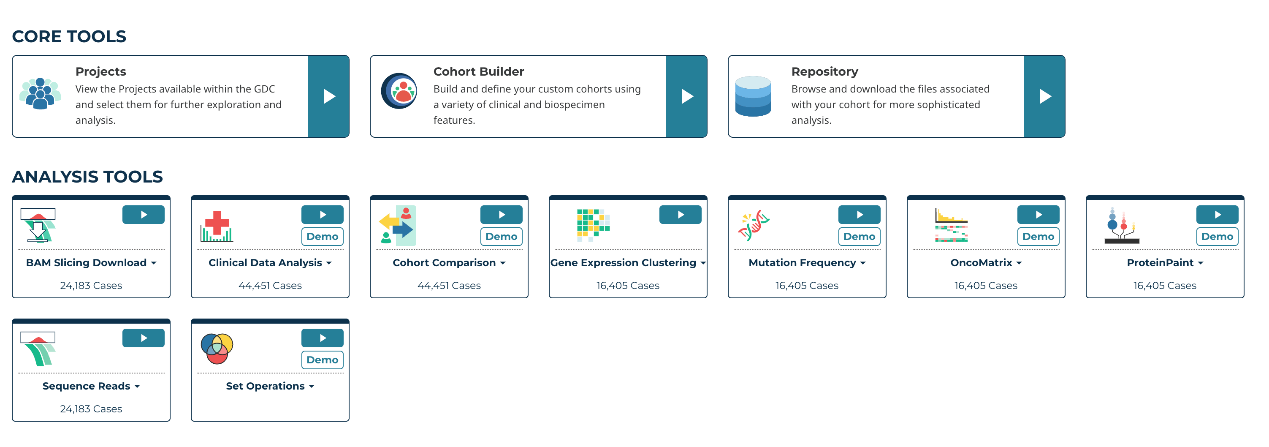
3. 使用数据分析和数据存储库之前需要通过队列生成器(cohort builder)创建自己感兴趣的cohort,比如TCGA-LUAD,选好之后自定义命名:
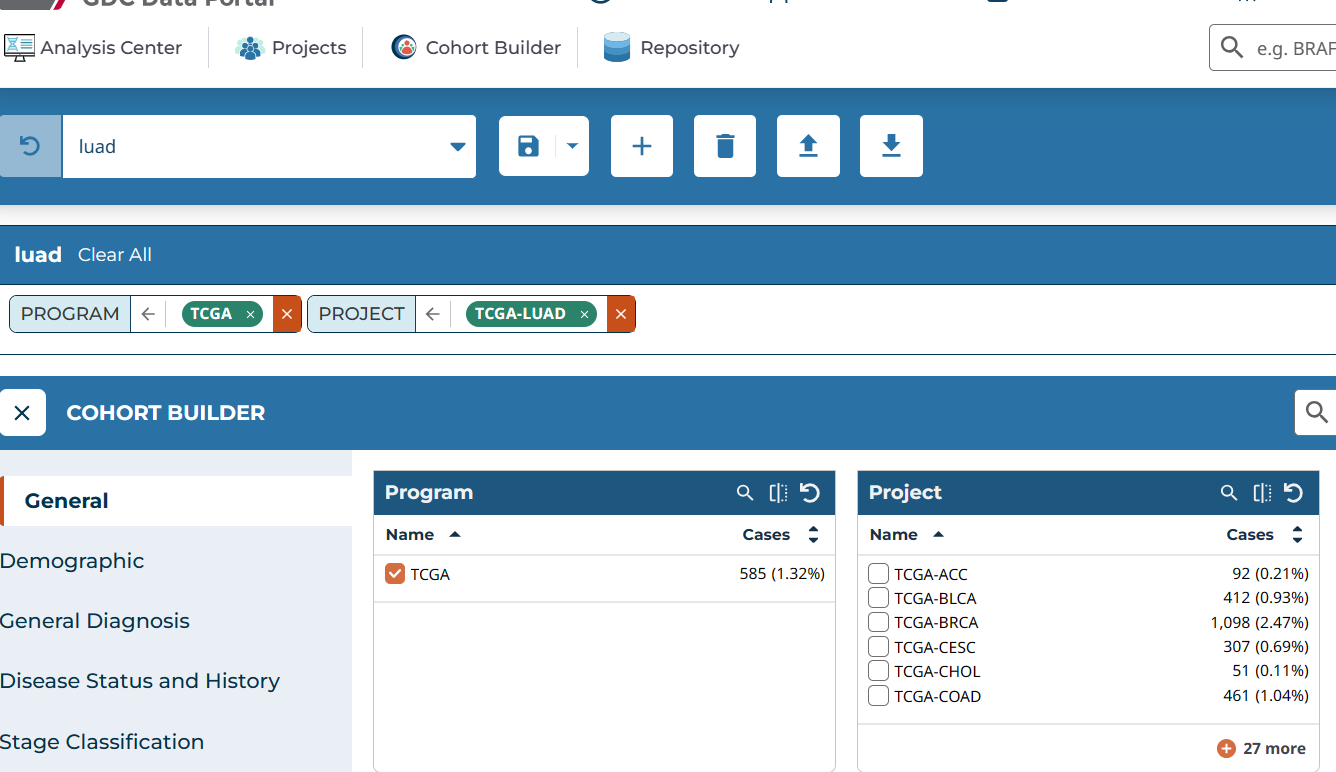
4. 转到存储库(Repository),选择自己的cohort,选择自己需要的数据类型
4.1 RNA测序数据,filter中勾选策略如下:
Experimental Strategy:RNA-Seq
Data Type:Gene Expression Quantification
Workflow Type:STAR – Counts
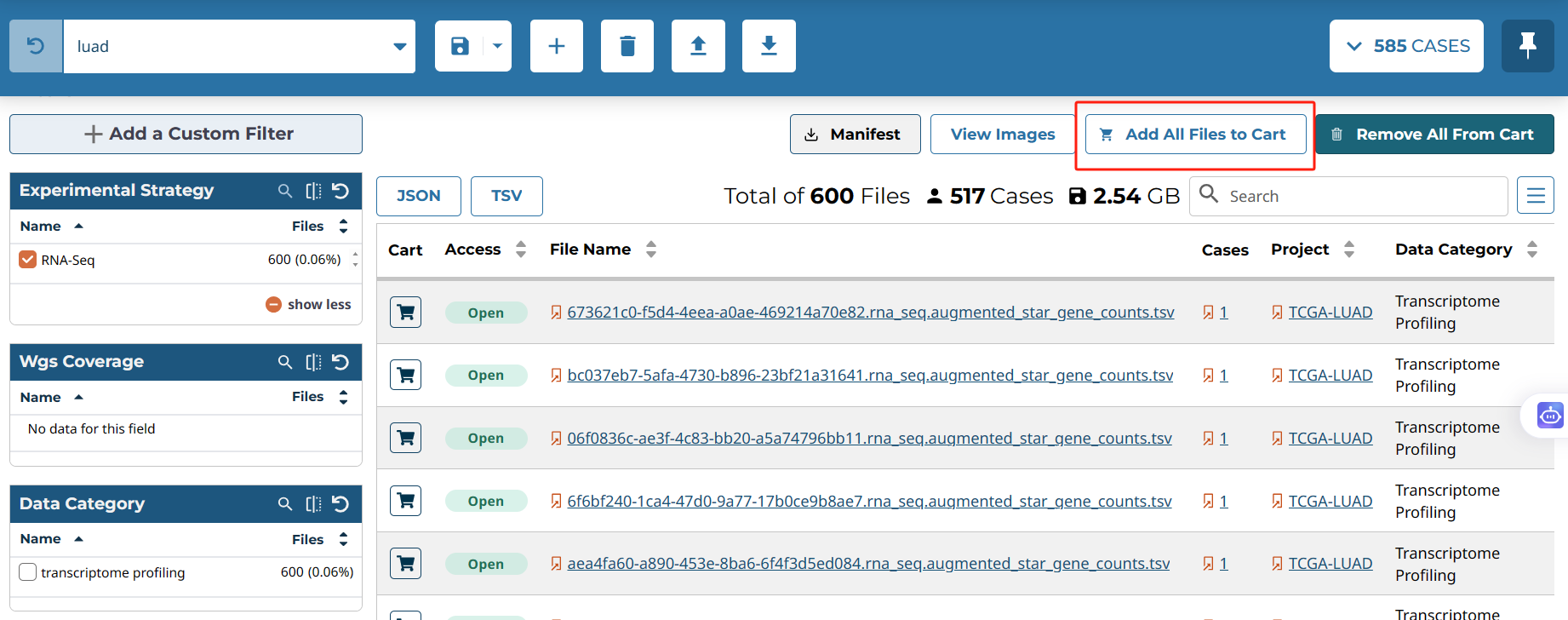
前俩选择好就可以了,显示如下,同1.0版本网站,add all files to cart:
4.2 对于miRNA数据
Experimental Strategy:miRNA-seq
Data Type:Isoform Expression Quantification(for isoform,包含5p/3p的数据);miRNA Expression Quantification(相当于stem-loop的定量数据,不区分5p/3p)
Workflow Type:BCGSC miRNA Profiling
同上,add all files to cart。
4.3 对于突变数据
Experimental Strategy:WSX
Data Type:Masked Somatic Mutation
Workflow Type:Aliquot Ensemble Somatic Variant Merging and Masking
同上,add all files to cart。
5. 下载数据
点击右上角购物车(Cart)进入购物车:

同GDC1.0,咱们需要下载两个文件用于整合数据:Download Cart(Cart)和Download Associated Data(Metadata)。同时还可以下载clinic data,TSV格式即可,excel可以打开访问。
对于数据整合,我网站上的代码依然适用,毕竟文件还是这俩:
新版TCGA表达mRNA/miRNA和临床数据下载及R语言整合代码 – 王进的个人网站
新版TCGA突变maf数据下载整合及瀑布图绘制和TMB计算 – 王进的个人网站
B站分享的整合及分析可视化APP也依然适用(列表中点击超链接致B站查看视频介绍):
B站代码获取 – 王进的个人网站
非常感谢您的分享教程,我想指正其中的一个笔误。在“4.3 对于突变数据”部分,您提到了”WSX“数据,应当是“WXS”数据
老师您好,我想为一下,我看有些文献直接下载了比如BRCA2突变的胃癌患者和非BRCA2突变患者的数据(包括转录组和临床数据),这种数据是怎么下载的啊,我没有找到如何选择某个基因突变的患者数据
大佬您好!为什么Download Cart(Cart)显示错误,没有下载下来?