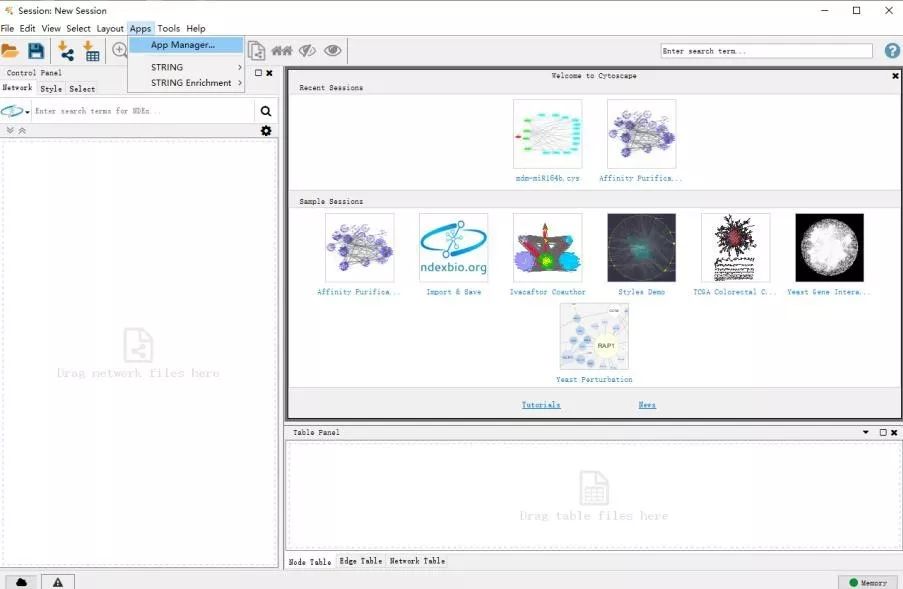
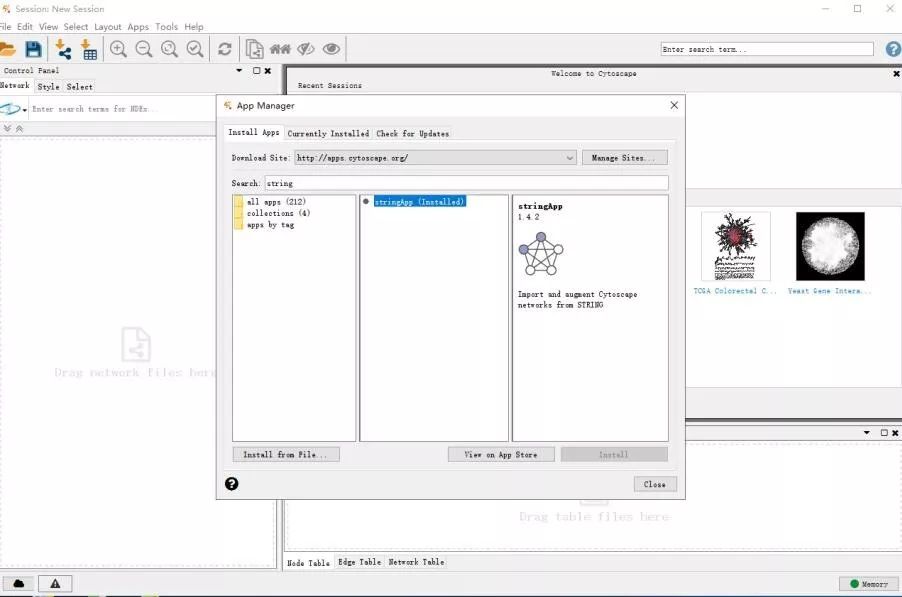
1. 打开cytoscape,点击Apps,选择App Manage,选择STRING APP进行安装。
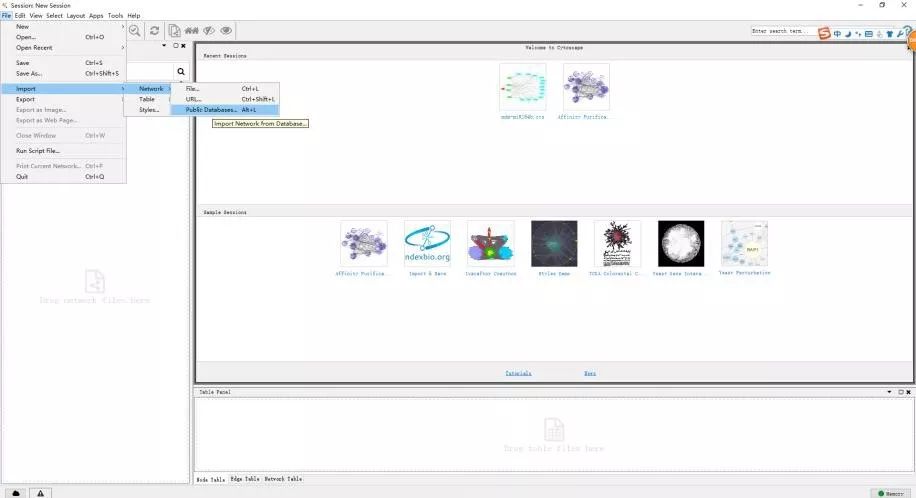
2. 安装完成后,开始导入蛋白互作网络,选择从公共数据库导入网络。打 开File-Import-Network–Public_Database。
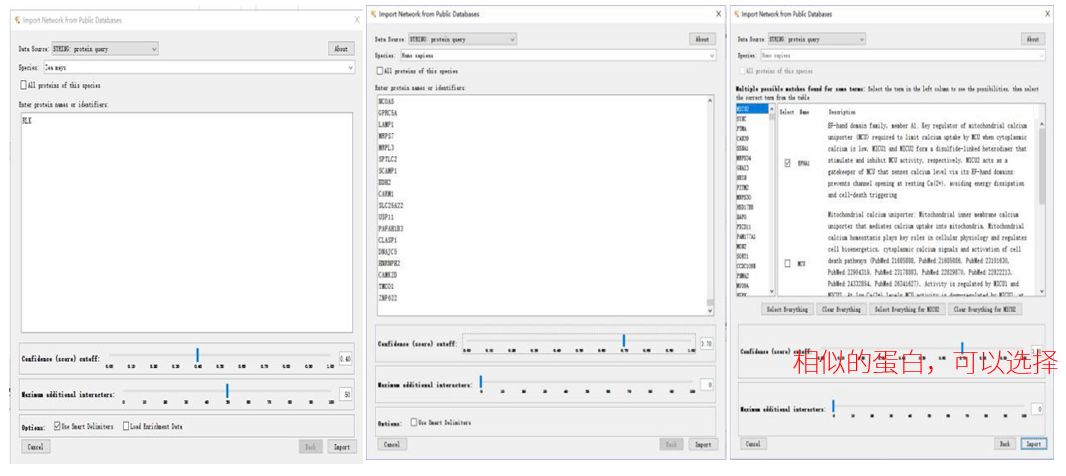
3. 选择Data Sourse为STRING:protein query,然后输入需要富集的蛋白。输入一个蛋白,则会导入与这个蛋白相关的相互作用;如果输入多个蛋白,则会导入多个蛋白间的相互作用。输入蛋白时,可能会和STRING数据库匹配到多个相似的蛋白,可以自行进行选择。Confidence Score代表蛋白间相互作用强弱,所设置的cut–off值即为所能接受的最低值。(low=0.15; medium=0.4;high=0.7;highest=0.9)。Maximum是为互作蛋白设置的最大值。
4. 蛋白导入后,会有一个初步的网络图。

5. 关于图片的样式设置,可以调整style部分。
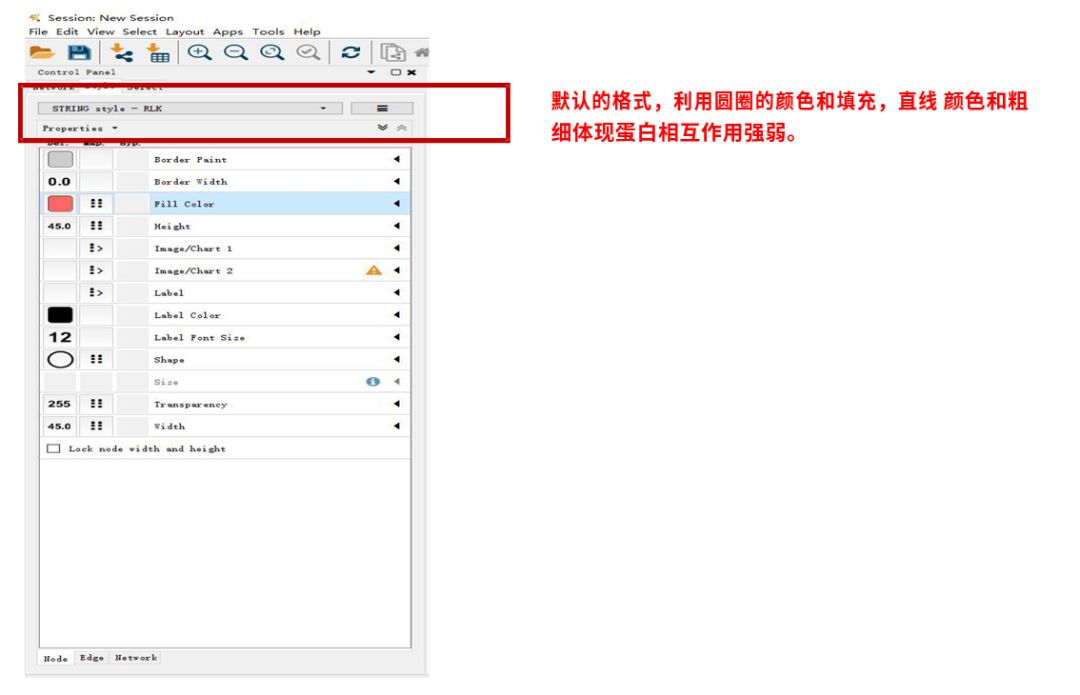
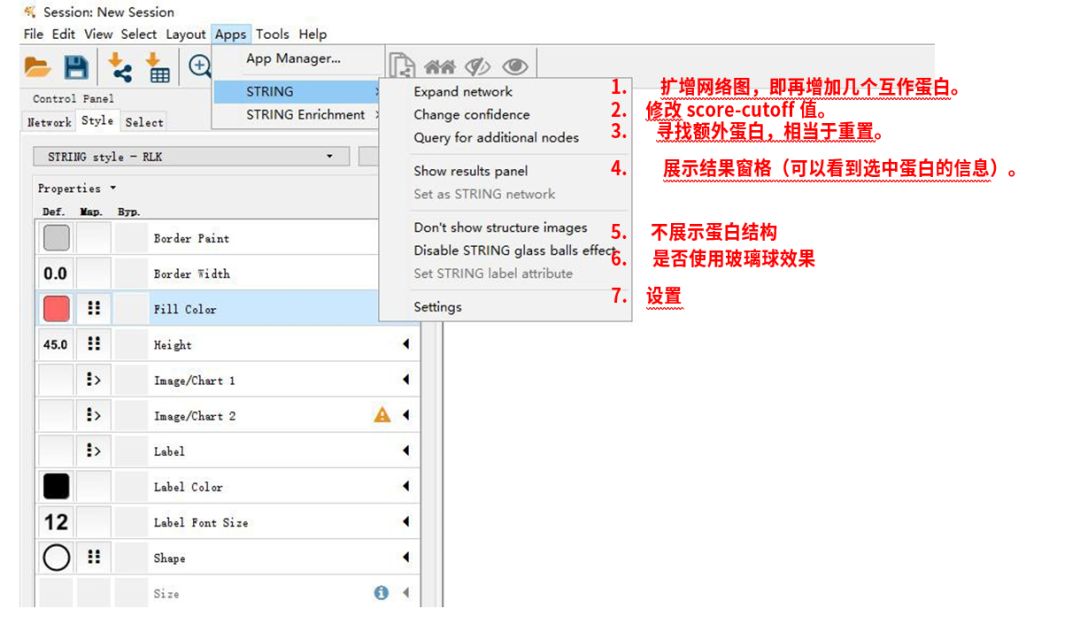
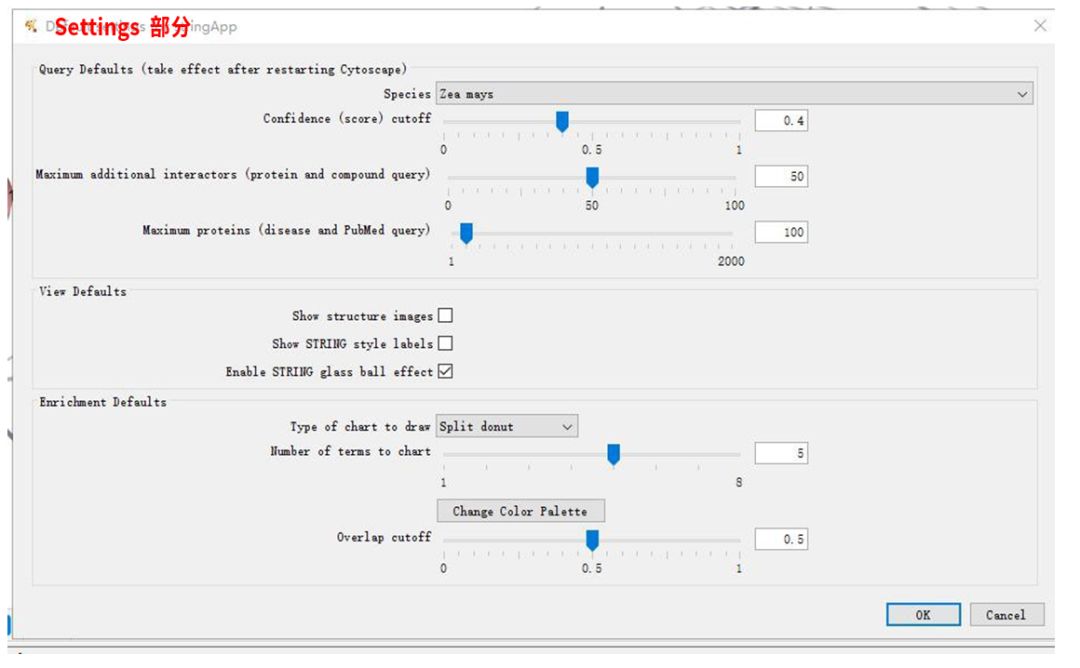
6. 关于网络图中内容的更改,在Apps–STRING下修改。
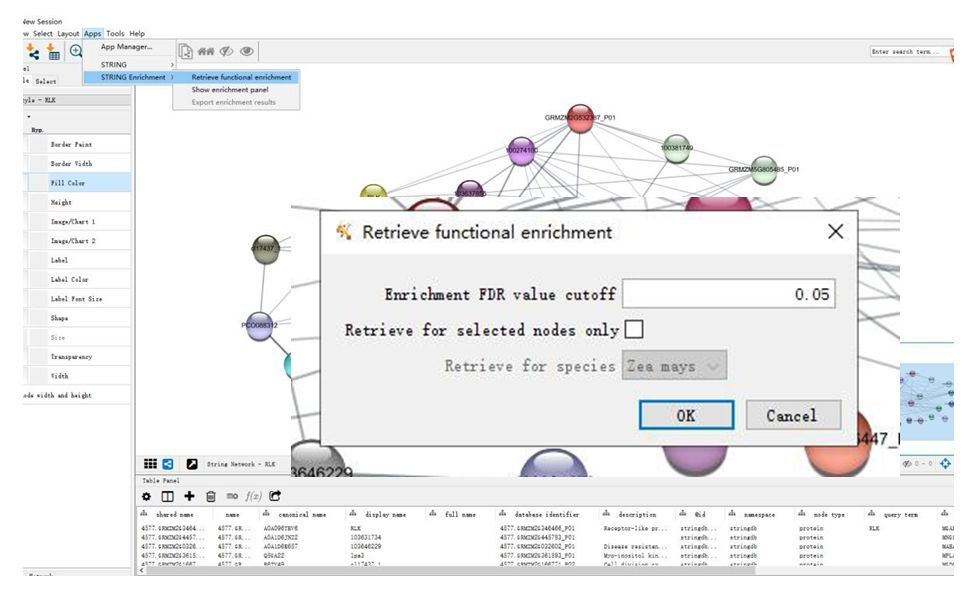
7. 富集分析——使用STRING Enrichment进行富集分析。选择STRING Enrichment下第一项。 默认Pvalue为0.05。
8. 富集结果在STRING Enrichment中展示。包括多种类别,通路,相关描述及富集结果。
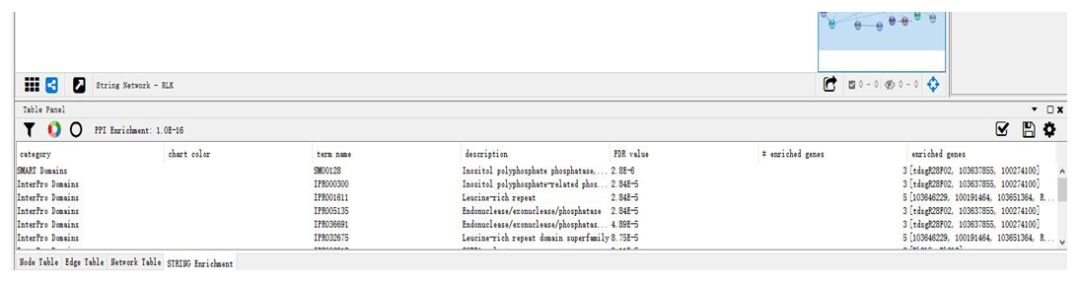
Table Panel上的黑色筛选键,可以用来筛选不同的功能富集类别。以KEGG为例,可以得到该网络图中富集到KEGG上的结果。选中通路成员,可以在网络图中体现。


为了展示结果,现将所有蛋白统一成粉色,选中一条通路,图中黄色的部分就是该通路的成员。
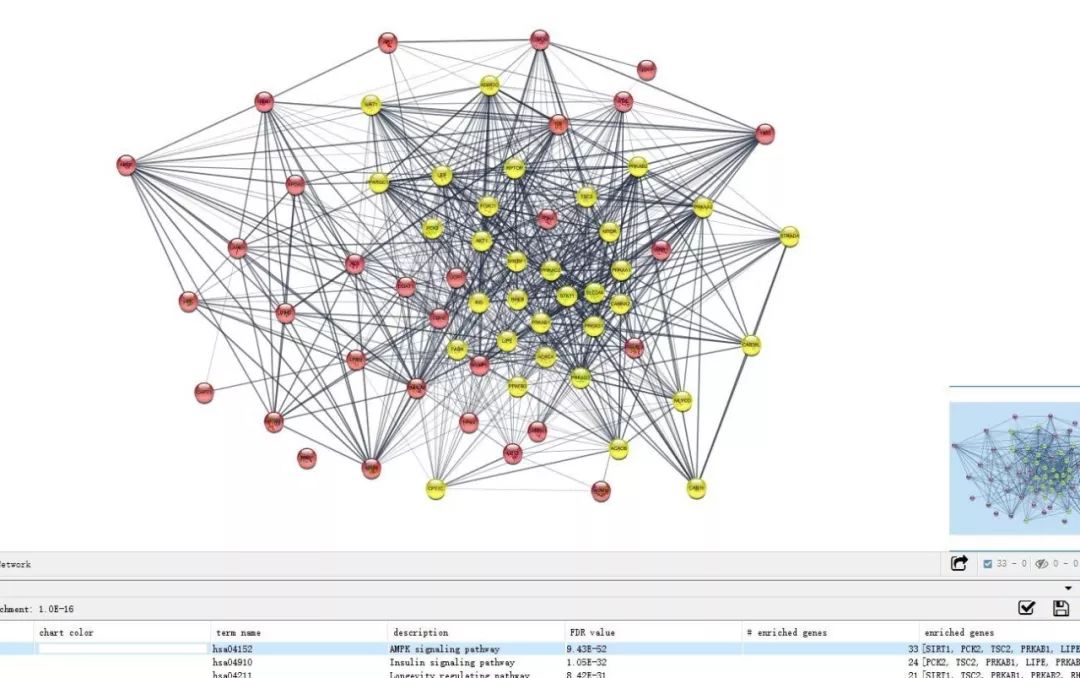
如果这条通路正是你所关注的,那么你可以将这些成员及其边构建成一个新网络,进行进一步查看。

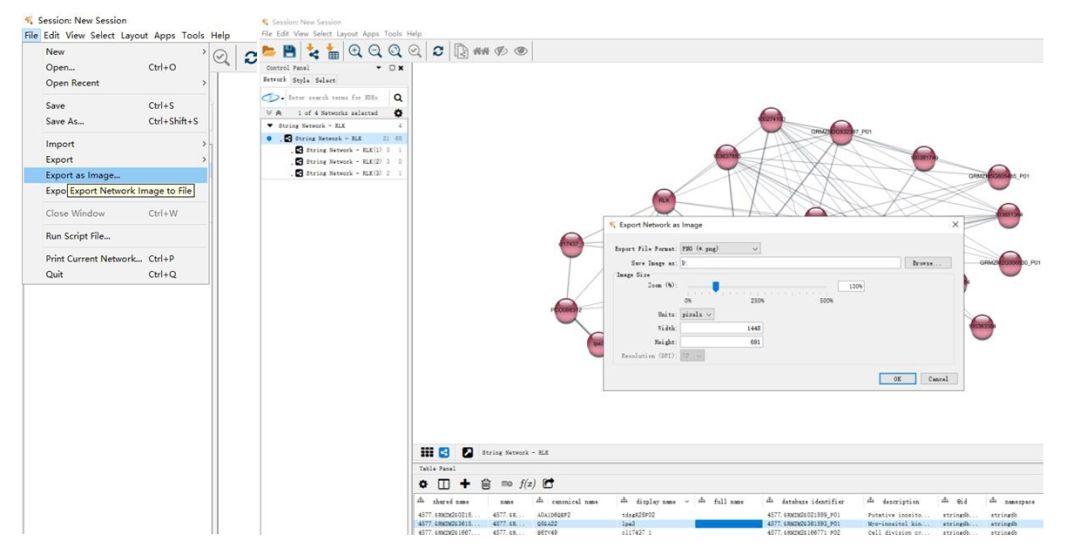
9. 没有连通到网络中的蛋白,可以删掉。然后就可以导出图片。
你好,请问我从app manager 里面 install 之后,install还是可以点击,currently installed 里面显示 stringApp 是Failed to Start 状态,这是怎么回事呢,怎么解决呢
Data Sourse为STRING:protein query,这一项不能选择是什么情况??只有universal interaction database client
你好,我的Cytoscape也安装不了插件,求string app插件的安装包
您好,我使用cytoscape内插string,粘贴蛋白后提示Your query returned no results
进哥哥您好,我以前用过stringApp,当时可以正常使用。现在隔了一年,重新打开cytoscape用stringApp的时候,import之后总会显示Your query returned no results。请问您知道是怎么回事吗?我重新安装后也没用。
您好,我用的cytoscape的3.10.0,但是我的插件都下载不了,能分享一下stringapp等常用软件的安装包吗?
https://console.box.lenovo.com/l/RJJUrH 提取码:psvk
这里面有几个 有其它需要告诉我,我补充 3.10.1好像不怎么方便
你好,请问这个插件的安装包还在吗
我有个问题就是,每次用stringapp绘制的数据直接做cytohubba的时候,显示出来的都是ensp的名字,不知道王老师有没有解决这个问题的办法