有这样的使用场景么?
1.已经确定研究的基因,但是想探索他潜在的功能,可以通过跟这个基因表达最相关的基因来反推他的功能,这种方法在英语中称为guilt of association,协同犯罪。
2.我们的注释方法依赖于TCGA大样本,既然他可以注释基因,那么任何跟肿瘤相关的基因都可以被注释,包括长链非编码RNA。
GSEA需要的gene set是现成的没有问题,但是genelist没有,这里我们可以把所有基因跟单个基因的相关性系数当做LogFC,有正有负,就解决了geneList的问题。这个想法不是我的,是我的一个学员的,不过他要解决的是microRNA把基因的问题。
下面来实战一下:
1.首先加载数据
这个数据是我下载了TPM数据,然后提取出乳腺癌的数据得来的。
load(file = 'BRCA_mRNA_exprSet.Rdata')
exprSet <- mRNA_exprSet
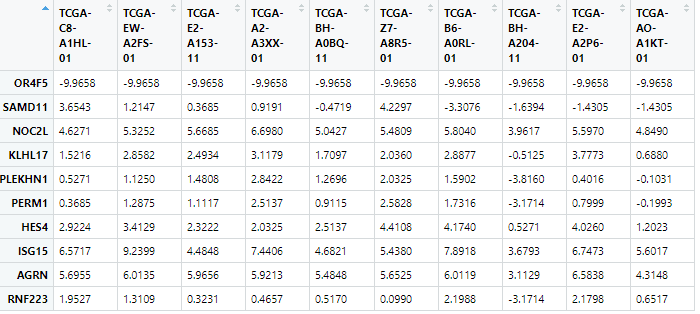
test <- exprSet[1:10,1:10]
2.写一个函数批量计算相关性
这个函数只要输入一个基因,他就会批量计算这个基因跟其他编码基因的相关
性,返回相关性系数和p值。
###对于有缺失值的基因,有效样本小于4会报错
batch_cor <- function(gene){
y <- as.numeric(exprSet[gene,])
rownames <- rownames(exprSet)
do.call(rbind,future_lapply(rownames, function(x){
dd <- cor.test(as.numeric(exprSet[x,]),y,type='spearman')
data.frame(gene=gene,mRNAs=x,cor=dd$estimate,p.value=dd$p.value )
}))
}
###这是修改的代码 加一个判断 样本量<10的就不要了吧
batch_cor <- function(gene){
rownames <- rownames(exprSet)
do.call(rbind,future_lapply(rownames, function(x){
xy <- exprSet[c(gene,x),]
xy <- t(xy) %>% na.omit() %>% as.data.frame()
if (nrow(xy)>10){
dd <- cor.test(as.numeric(xy[,1]),as.numeric(xy[,2]),type='spearman')
data.frame(gene=gene,mRNAs=x,cor=dd$estimate,p.value=dd$p.value )
}
}))
}
3.并行化运行函数
以PCDC1这个基因为例
library(future.apply)
plan(multiprocess)
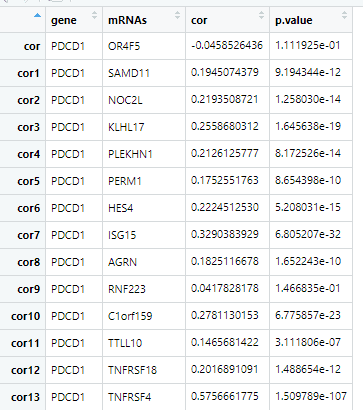
system.time(dd <- batch_cor('PDCD1'))
这是返回的结果
4.制作genelist
gene <- dd$mRNAs## 转换
library(clusterProfiler)
gene = bitr(gene, fromType='SYMBOL', toType='ENTREZID', OrgDb='org.Hs.eg.db')## 去重
gene <- dplyr::distinct(gene,SYMBOL,.keep_all=TRUE)
gene_df <- data.frame(logFC=dd$cor,
SYMBOL = dd$mRNAs)
gene_df <- merge(gene_df,gene,by='SYMBOL')
## geneList 三部曲
## 1.获取基因logFC
geneList <- gene_df$logFC
## 2.命名
names(geneList) = gene_df$ENTREZID
## 3.排序很重要
geneList = sort(geneList, decreasing = TRUE)
5.运行GSEA分析
从GESA(https://www.gsea-msigdb.org/gsea/downloads.jsp)的官网上,下载一个gmt文件
library(clusterProfiler)
## 读入hallmarks gene set,从哪来?
hallmarks <- read.gmt('h.all.v6.2.entrez.gmt')
# 需要网络
y <- GSEA(geneList,TERM2GENE =hallmarks)
作图看整体分布
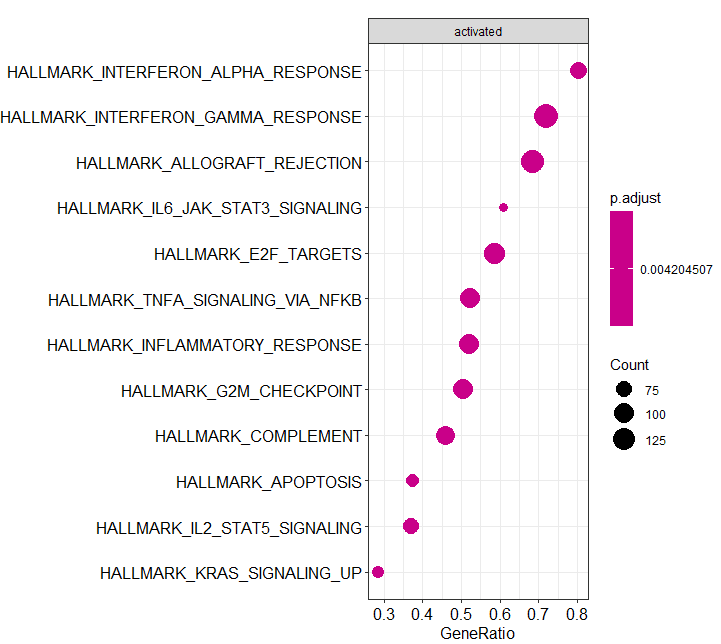
### 看整体分布library(ggplot2)
dotplot(y,showCategory=12,split='.sign')+facet_grid(~.sign)
本次结果中全是激活的
6.特定通路作图
yd <- data.frame(y)
library(enrichplot)
gseaplot2(y,'HALLMARK_INTERFERON_ALPHA_RESPONSE',color = 'red',pvalue_table = T)
PCDC1跟阿拉法干扰素正相关,这个事情没什么好说的吧。
如需要批量进行几个基因的分析,可以包进一个函数会更方便。
好了,我们又掌握了一个特别强悍,实用的技能。我是进哥哥,有问题随时留言讨论。
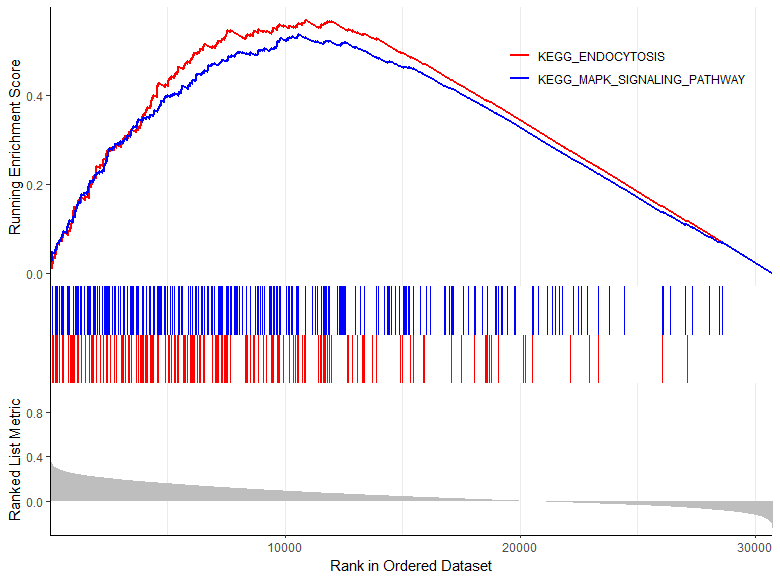
##对于多个通路绘制在一起:::
pathway=c("KEGG_ENDOCYTOSIS","KEGG_MAPK_SIGNALING_PATHWAY")
gseaplot2(y,pathway,color = c('red',"blue"),pvalue_table = F)

基于相关性分析进行GSEA是否有文献参考呢 我查了没查到 不知道是不是在方法里写清楚就行?
写清楚方法吧,这个常规做法,按照相关性系数作为权重进行GSEA
不太能够理解,以示例图为例子的话,这种方法是说明相关度高的基因在该通路中起贡献作用,相关度低的基因起抑制作用,从而推测该基因与贡献相关,这种方法也可以应用于go富集分析么,
这个方法并不能说明activate还是inhibit作用,只能表示对该通路的贡献。无非就是得到一个按照特定权重排序的基因集,这里是相关性系数,差异分析是logFC
其他分析也一样,GO、Cluster等等
好的好的,感谢解惑
基于相关性分析进行GSEA是否有文献参考呢 我查了没查到 不知道是不是在方法里写清楚就行?
您好,请问TPM数据是只需要肿瘤患者的数据还是要肿瘤+正常患者的数据呢?
因为基于相关性分析嘛 其实加上正常组织未尝不可,你方法中写清楚就好
好的!谢谢!
您好,请问为什么运行到system.time(dd <- batch_cor('PDCD1'))这一步就变得特别慢,甚至一个小时都不出结果?
你的数据很大?还是电脑配置不够?并行运行需要根据电脑配置修改一些参数 ,不然很卡很慢
或者直接用for循环进行 不并行处理
数据倒也不大以前一直可以运行的,最后用for循环解决啦!感谢!
error in if(abs(max.ES)>abs(min.ES)){:missing value where TRUE/FALSE needed,请问老师,这个是什么意思呢,最后的出图出不来。望老师帮帮我。
这样看不出来诶。。。确定你所要画的通路在富集结果里面吗 并且拼写正确?可以加我微信具体讨论
我刚刚也留言碰到同样的问题 找了GitHub也有人有相同疑问 我在另一个帖子下面留言了 o(╥﹏╥)o
您好,本人也困于此问题,请问您于王博交流后,此问题如何处理的呢。烦请告知,多有打扰,谢谢~
加我微信吧 得一步步运行看看什么问题
请问这个问题解决了吗,我也遇到了同样的问题
不清楚指的那个问题?都可以解决 如果还没解决 可以加我微信 我看看
您好,可以提供一下PTM吗?想试试
您好 发给你数据太大,这个不一定需要TPM数据,只要是任意标准化的表达矩阵都可以,你可以从XENA上下载任意肿瘤的表达矩阵进行分析,你先试试 有问题加我微信讨论
在进哥的指导下,运行成功,再次感谢进哥!
您好!非常感谢您提供便捷的学习资料。想问下您:我在运行“plan(multiprocess);system.time(dd <- batch_cor('PDCD1'))”时,显示“ Error: ‘node$session_info$process$pid == pid’ is not TRUE ” ,这个问题应该怎么解决。谢谢
你好,这个得看你的数据,要不你加我微信详细讨论,我的简历最下面有微信
请问要单基因的gsea图包含多条通路怎么做呢,谢谢!
哈哈,晚一点出个教程
对于clusterprofiler包的结果,直接绘制时添加多个通路即可,文中最后已补充 请参考
你好,王老师,多个通路的颜色没办法自己绘制,是不是要加个颜色的代码。