在R语言中运行Cibersort共需要三个文件,分别是(1)官方提供的22种细胞基因集“LM22.txt”;(2)自己的表达矩阵;(3)Cibersort代码。
(1)LM22.txt获取方法:
在Cibersort论文中(https://www.nature.com/articles/nmeth.3337#MOESM207)下载Supplementry table 1

LM22.txt获取方法
删除Sheet 1 中表头,只保留矩阵部分。
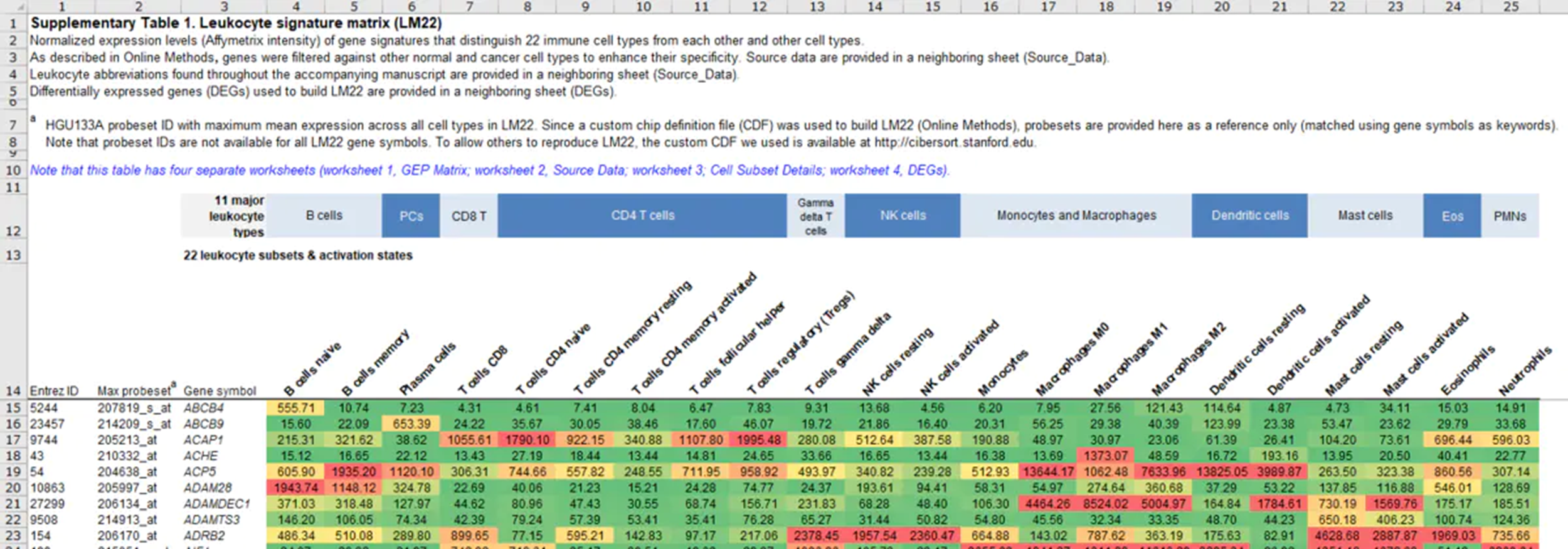
删除Sheet 1 中表头
得到如下矩阵,另存为制表符分割的txt(“LM22.txt”)
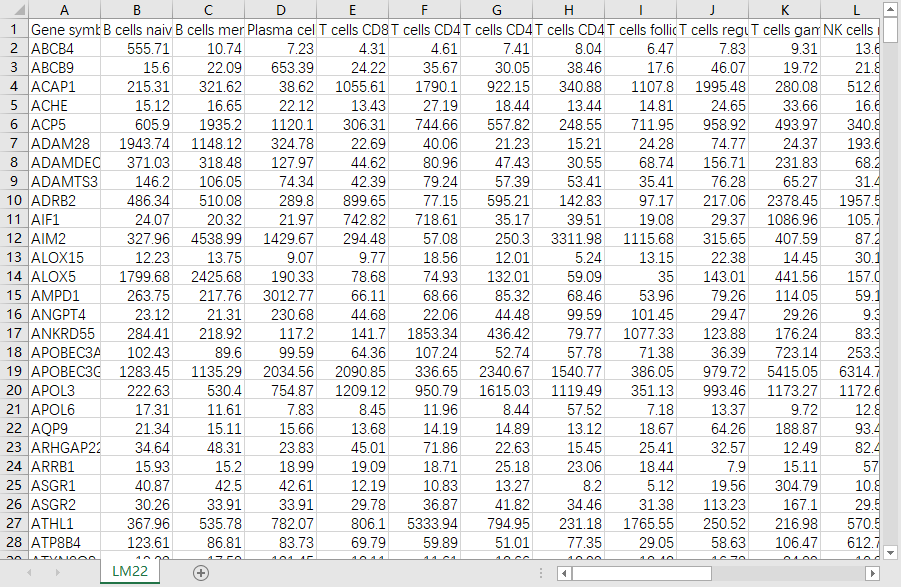
(2)自己的表达矩阵
第一列是基因名,第一行是样品名,不能有重复基因名,第一列列名不能空白。矩阵中不能存在空白或NA值,不要对表达量取Log2.
如果表达矩阵中基因不能完全覆盖LM22.txt中的基因,Cibersort同样可以正常运行,但不能少于LM22.txt中所需基因的一半。
表达矩阵保存为制表符分割的txt文本(“DATA.txt”)
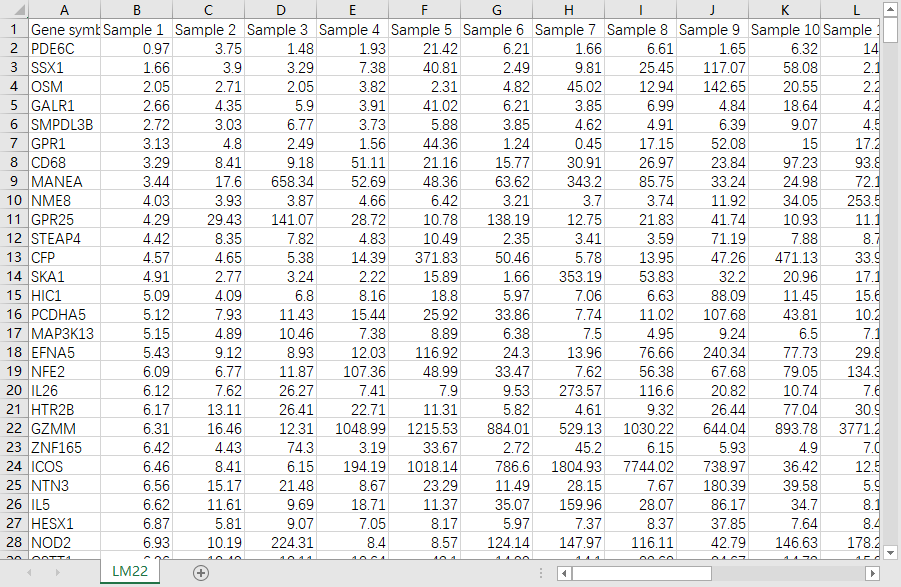
表达矩阵示例
(3)Cibersort代码
在R中新建R Script,复制以下网址中代码,保存为“Cibersort.R”(进哥已提供,下载后修改后缀为.R)
https://rdrr.io/github/singha53/amritr/src/R/supportFunc_cibersort.R
(4)以上三个文件需保存在同一文件夹,运行Cibersort的代码如下:
setwd("三个文件的文件夹")
source('Cibersort.R')
result1 <- CIBERSORT('LM22.txt','DATA.txt', perm = 1000, QN = T) #perm置换次数=1000,QN分位数归一化=TRUE在同一文件夹下可以得到运算结果(”CIBERSORT-Results.txt”)
注意Cibersort结果的默认文件名为CIBERSORT-Results.txt,在同一文件夹下进行第二次运算会覆盖第一次得到的文件,建议在每一次运算之后对文件重命名。
CIBERSORTx是CIBERSORT的进阶版,是Alizadeh和Newman实验室开发的网页分析工具,目前还没有可下载的R代码。它用于估算基因表达谱,并使用基因表达数据估算混合细胞群中免疫细胞的类型和丰度,在肿瘤数据处理中使用广泛。
网站:https://cibersortx.stanford.edu
看下tutorial

ok,来整理我的mixture。

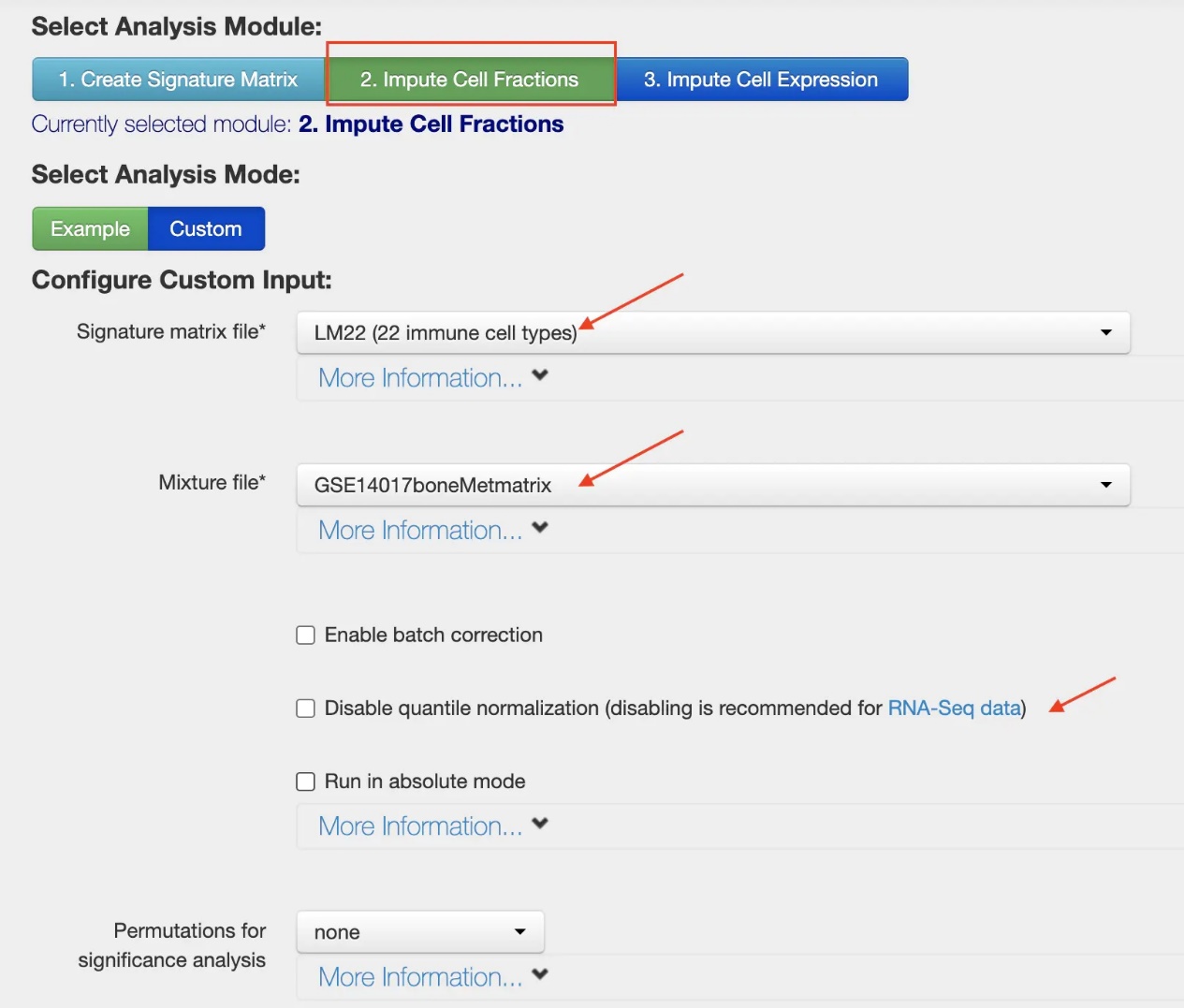
我的数据是microarray,所以最后那个箭头不勾选。
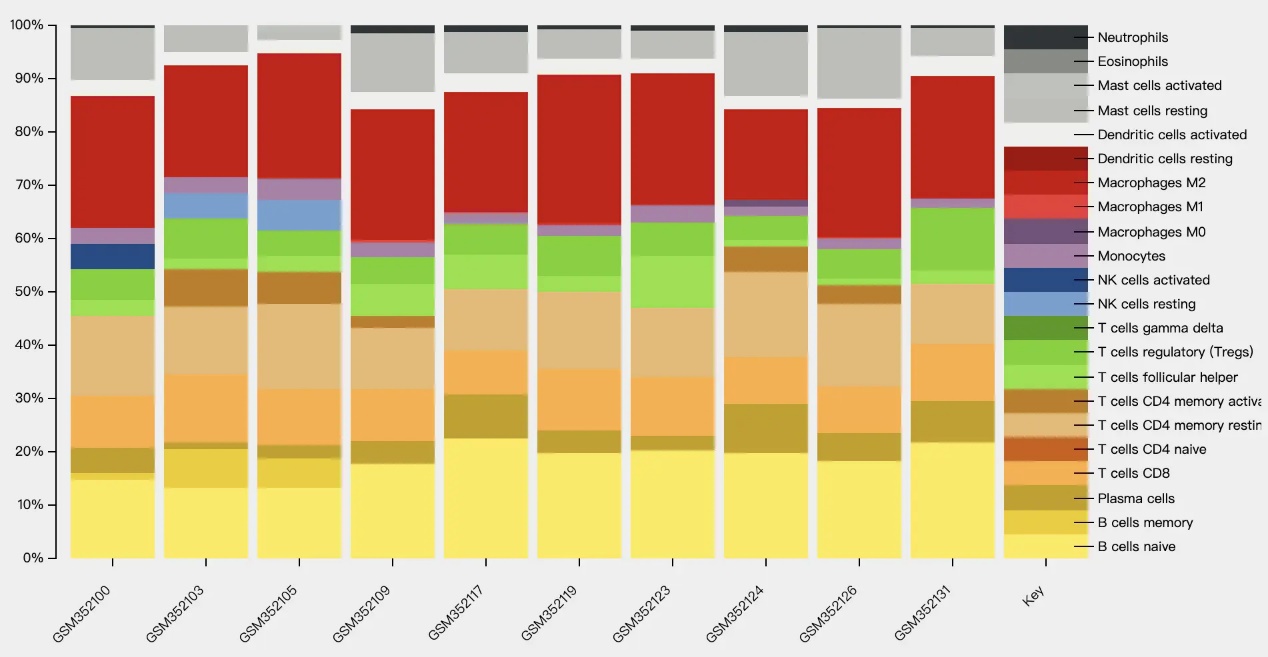
数据可下载txt/csv/..,图片是截图的。。没找到下载的地方。
Error in mat$cv : $ operator is invalid for atomic vectors
Calls: CIBERSORTxGEP -> runCIBERSORTxGEP
In addition: Warning message:
In mclapply(1:no_cores, res, mc.cores = no_cores, mc.set.seed = FALSE, :
all scheduled cores encountered errors in user code
Execution haltedan按照正常流程进行网页版分析,有两个相同流程的文件一个能跑另一个就会出现问题。可以帮忙解答一下吗
跑完setwd(“三个文件的文件夹”)
source(‘Cibersort.R’)
result1 <- CIBERSORT('LM22.txt','DATA.txt', perm = 1000, QN = T)
出现收捲时出错: 无法打开链结
Error: no more error handlers available (recursive errors?); invoking 'abort' restart
请问您解决了吗,我也碰到一样的问题,谢谢
X <- read.delim("/data2/wangyichao/gastric/data/TCGA/exp.txt", header = T, sep = "\t",
row.names = 1, check.names = F)
X <- data.matrix(X)
results <- cibersort(sig_matrix = "sig.txt",mixture_file = X,perm = 1000,QN = F)
进哥,我的能正常跑,但他一直提示WARNING: reaching max number of iterations
这个次数比设置的perm还多,也能跑出来结果,这个可以用嘛。
王博你好,我想请教一下,你有没有试过high resolution mode, 且要求超过1000个基因?因为我想得到所有基因的high resolution mode的结果,但是又限制基因数,没办法用来做下游的cell-cell communication 分析,例如CellphoneBD 和CellChat
进哥您好!在cibersortx网站上,利用单细胞测序数据集构建Signature Matrix File中,怎么做出和示例一样的single cell reference sample file呢?我做的矩阵每个样本的基因都只有一个表达值,是不是我理解有问题呢?QAQ
解决了吗?我最近比较忙 刚刚看见留言,没解决加微信讨论吧
Error in strsplit(colnames(scmatrix), “.”, fixed = T) :
non-character argument
Calls: CIBERSORTxFractions -> makeRefandClassFiles -> sapply -> strsplit
Execution halted
王博您好,我想创建一个自己的参考基因集~但是遇到了这样的报错。
Error in strsplit(colnames(scmatrix), “.”, fixed = T) :
non-character argument
提示的是你colnames(scmatrix)不是字符型变量,你的样本名称是数字?如果是这样,改成含有字母的编号
其它问题加微信讨论
王老师,您好!我遇到与楼上一样的问题,使用的是CIBERSORTx的网页版,报错如下:
Error: $ operator is invalid for atomic vectors
In addition: Warning messages:
1: In max(Y) : no non-missing arguments to max; returning -Inf
2: In mclapply(1:svn_itor, res, mc.cores = svn_itor) :
all scheduled cores encountered errors in user code
Execution halted
按照您上面的回复检查过列名无重复,请教老师如何解决。谢谢!
加我微信吧 我看看你的数据 导航栏我的简历有手机号和微信
您好,我是在CIBERSORTx网站上运行的,数据库格式按照例子来的,应该没有问题,运行就会出现以下错误,不知道怎么回事,期待您的指导。
Error in sample.int(x, size, replace, prob) : invalid ‘size’ argument
Calls: CIBERSORTxFractions -> sort -> doPerm -> sample -> sample.int
Execution halted
加微信吧,我试试 网站我的简历有手机号和微信
Error: $ operator is invalid for atomic vectors
In addition: Warning messages:
1: In CIBERSORTxFractions(mixture = arginitvals[1], sigmatrix = arginitvals[2], :
2 duplicated gene symbol(s) found in mixture file!
2: In mclapply(1:svn_itor, res, mc.cores = svn_itor) :
all scheduled cores encountered errors in user code
Execution halted
你好,我是在cibersortx网站运行的,检查了数据的格式都是正确的但是一直会报这个错误,换成别人的数据(在他的电脑上能跑)也不行,请问您知道这是为什么吗?
你好 请先检查文件列名是否有重复,也就是样本名称不能有重复 如果不是这个问题 可以加我微信发数据看看
并不是列名的问题。请问我该怎么加您的微信呢
顶上导航栏 我的简历 有电话和微信
您好,
1)请问出现报错:原因是?
Error in CIBERSORT(“LM22.txt”, “av_rena.txt”, perm = 1000, QN = F) :
None identical gene between eset and reference had been found.
Check your eset using: intersect(rownames(eset), rownames(reference))
2)samplename可以是分好组的细胞组别吗?
根据提示,你的问题应该是基因名称不对,确定是否为人的official gene symbol
老师,我也是照着您的方法做的,但是最后运行的时候老是报错
results=CIBERSORT(“dd.txt”,”cc.txt”,perm = 50, QN = F)
Error in read.table(mixture_file, header = T, sep = “\t”, row.names = 1, :
不允许有重复的’row.names’
3.
stop(“duplicate ‘row.names’ are not allowed”)
2.
read.table(mixture_file, header = T, sep = “\t”, row.names = 1,
check.names = F) at cibersort.R#126
1.
CIBERSORT(“dd.txt”, “cc.txt”, perm = 50, QN = F)
如报错提示 你的mixture_file里面行名基因名称有重复 先去重 参考这篇去重解决GEO数据中的多个探针对应一个基因
你回复的系统邮件,我贴过来:
老师,这个是我的代码
source(‘Cibersort2.R’)
results=CIBERSORT(“LM22.txt”,”dat.exp.txt”,perm = 50, QN = F)
但结果报错提示:
Error in read.table(mixture_file, header = T, sep = “\t”, row.names = 1, :
不允许有重复的’row.names’
3.stop(“duplicate ‘row.names’ are not allowed”)
2.read.table(mixture_file, header = T, sep = “\t”, row.names = 1,
check.names = F) at Cibersort2.R#126
1.CIBERSORT(“LM22.txt”, “dat.exp.txt”, perm = 50, QN = F)
老师,mixture_file这个文件是Cibersort2.R中的一个文件,意思是Cibersort2.R这个里面的代码有问题吗,这个是我去Cibersort官网粘贴的。
回复:
mixture_file文件是你自己的表达矩阵文件 你加我微信吧
> setwd(“C:\\Users\\PC\\Desktop\\cibersort”)
> source(‘Cibersort.R’)
> result1 <- CIBERSORT('LM22.txt','DATA.txt', perm = 1000, QN = T)
Error in CIBERSORT("LM22.txt", "DATA.txt", perm = 1000, QN = T) :
没有可中断/下一步骤的循环, 跳到最高层
王博您好,我完全按照您的步骤进行运行,确定没有重复,没有空白,基因也占三分之二,但是就是不行,这种还有其他可能吗。期待得到您的指导!
不好意思,这两天忙,忘记回复您了,你可以发我数据 我运行看看,可以加微信讨论
请问您解决了吗,我也碰到一样的问题,谢谢
您好,请问您最后怎么解决的呢,我遇到的也是同样的问题
source(“Gene25.CIBERSORT.R”)
> results=CIBERSORT(“ref.txt”, “uniq.symbol.txt”, perm=1000, QN=TRUE)
Error in CIBERSORT(“ref.txt”, “uniq.symbol.txt”, perm = 1000, QN = TRUE) :
没有可中断/下一步骤的循环, 跳到最高层