比值比(Odds ratio, OR):又名机会比,优势比,交叉乘积比( Cross-product Ratio),相对比值( Relative Odds ),两个比值的比。暴露因素与疾病的关系 在进行暴露因素和疾病关系的研究时,暴露和疾病的关系可以总结为下列四格表:
| 暴露 | 无暴露 | |
| 病例 | a | b |
| 对照 | c | d |
比值比OR=ad/bc。
①在病例对照研究中,比值比指病例组中暴露与非暴露人数的比值(a/b)和对照组中暴露与非暴露人数的比值 c/d) 的比,得出OR=ad/bc,所以又叫交叉乘积比,该值用作相对危险度的估计值。
②在队列研究中,指的是暴露组中患病与非患病者的比值(a/c)和非暴露组中患病与非患病者的比值(b/d)的比。也用作相对危险度的估计值。在队列研究中,可以计算相对危险度,所以一般不计算比值比,但是有的时候根据需要也应用OR作为联系的强度的指标,例如在应用logistic回归模型对队列研究的资料进行多因素分析时,即应用OR值。
本文将介绍OR的批量计算以及结果可视化,以下代码经供参考,并不是最好的实现方式,欢迎大家留言讨论,一起学习。
aa<- read.csv('I:/COR-NGS-ALL.csv')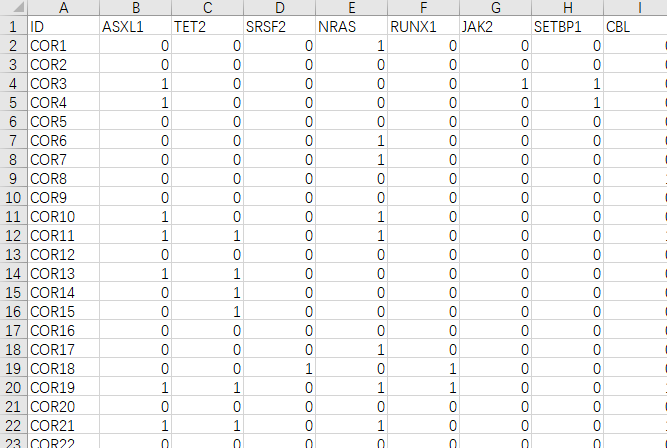
glm1<- glm(formula = ASXL1 ~ TET2,
data=aa,
family = binomial)
glm2<- summary(glm1);glm2
#计算OR并保留两位数
OR<-round(exp(coef(glm1)),2)
将上述计算包装成函数进行批量分析:
Uni_glm_model<-
function(x,y,aa){
#拟合结局和变量
FML<-as.formula(paste0(x,"~",y))
#glm()逻辑回归
glm1<-glm(FML,data=aa,family = binomial)
#提取所有回归结果放入glm2中
glm2<-summary(glm1)
#1-计算OR值保留两位小数
OR<-round(exp(coef(glm1)),3)
#2-提取SE
SE<-glm2$coefficients[,3]
#3-计算CI保留两位小数并合并
CI5<-round(exp(coef(glm1)-1.96*SE),3)
CI95<-round(exp(coef(glm1)+1.96*SE),3)
CI<-paste0(CI5,'-',CI95)
#4-提取P值
P<-round(glm2$coefficients[,4],3)
#5-将变量名、OR、CI、P合并为一个表,删去第一行
Uni_glm_model <- data.frame('Characteristics'=x,
'OR' = OR,
'CI' = CI,
'P' = P)[-1,]
#返回循环函数继续上述操作
return(Uni_glm_model)
}
for循环批量计算基因两两OR值:
#这里我选择了本数据的第2-14列的变量
#把它们放入variable.names中
variable.names<- colnames(aa)[c(2:14)];variable.names
#变量带入循环函数
results_or<-data.frame(matrix(NA,13,13))
colnames(results_or)<-variable.names
rownames(results_or)<-variable.names
results_p<-results_or
for (i in (1:(length(variable.names)-1))) {
for (j in (i+1):(length(variable.names))) {
results_or[i,j]<-Uni_glm_model(variable.names[i],variable.names[j],aa)$OR
results_p[i,j]<-Uni_glm_model(variable.names[i],variable.names[j],aa)$P
}
}
results_or<-results_or[-13,-1]
results_p<-results_p[-13,-1]
results_or$x<-rownames(results_or)
results_p$x<-rownames(results_p)
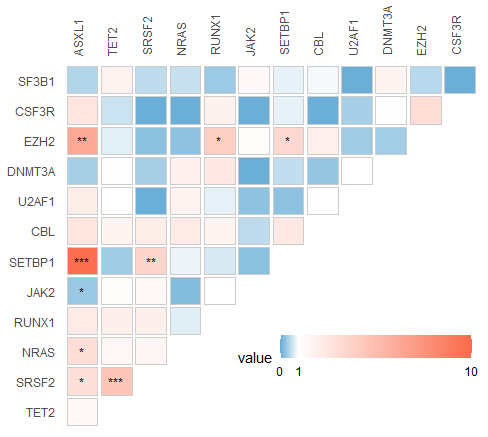
ggplot2绘制热图,其它函数都可以:
library(dplyr)
library(ggplot2)
library(grDevices)
results_or %>%
reshape2::melt(id.vars="x",variable.name="y") %>%
na.omit() -> dftmp
results_p %>%
reshape2::melt(id.vars="x",variable.name="y") %>%
na.omit() -> dftmp_p
dftmp$p<-dftmp_p$value
dftmp$plab<-ifelse( dftmp_p$value<0.001,"***",ifelse(dftmp_p$value<0.01,"**",ifelse(dftmp_p$value<0.05,"*","")))
dftmp$x<-factor(dftmp$x,
levels = variable.names)
dftmp$y<-factor(dftmp$y,
levels = variable.names)
cols<-c(colorRampPalette(c("#6BAED6","#FFFFFF"))(10),
colorRampPalette(c("#FFFFFF","#FB6A4A"),bias=2)(90))
ggplot(data=dftmp,aes(x,y,fill=value))+
geom_tile(colour="grey80",
#linewidth=2,
width=.9,
height=.9)+
theme_minimal()+
theme(panel.grid = element_blank(),axis.text.x = element_text(angle=90, hjust=-0.02))+
scale_x_discrete(position = "top")+
scale_y_discrete(position = "left")+
labs(x=NULL,y=NULL)+scale_fill_gradientn(colours= cols, limits=c(0, 10),
breaks=c(0,1,10),
na.value=rgb(246, 246, 246, max=255),
labels=c("0", "1", "10"),
guide=guide_colourbar(ticks=T,barwidth=10))+
theme(legend.position = c(0.7, 0.2),legend.direction = "horizontal")+
geom_text(aes(label = plab),col='black',cex=3)
您好,您的循环是基因间互为结局和变量的循环,如果我固定结局为生存状态,变量是不同免疫细胞的话,这个循环应该如何改进呀?我尝试着改了一下,没成功?
您好 要不你加我微信发我数据和需求 我来看看