我们经常使用Y叔的clusterprofiler包进行GSEA分析,之后使用gseaplot或enrichplot::gseaplot2来可视化结果。这两个函数中可以调整的参数较少,因此我们希望把图导出为ggplot对象,然后用ggplot2包重新对其进行调整。
enrichplot::gseaplot2最后产生的对象是使用plot_grid按列组合形成的图,我们需要组合前的图形对象,因此需要对该函数小做调整。
my_gseaplot2<-function (x, geneSetID, title = "", color = "green", base_size = 11,
rel_heights = c(1.5, 0.5, 1), subplots = 1:3, pvalue_table = FALSE,
ES_geom = "line")
{
-----------------------------------------------------------------------------------------
中间代码
-----------------------------------------------------------------------------------------
#plot_grid(plotlist = plotlist, ncol = 1, align = "v", rel_heights = rel_heights)
#修改为
return(plotlist)
}
#在这最后,需要将自定义函数的环境修改为原函数的环境,不然会报错
environment(my_gseaplot2)=environment(gseaplot2)
当然,除了自定义函数,我们也可以通过调整subplots参数,逐一输出ggplot对象来达到目的。
expr_DEG_limma_GSEA_go%>%gseaplot2(.,expr_DEG_limma_GSEA_go@result$ID,subplots = 1) ->up_plot
expr_DEG_limma_GSEA_go%>%gseaplot2(.,expr_DEG_limma_GSEA_go@result$ID,subplots = 2) ->mid_plot
expr_DEG_limma_GSEA_go%>%gseaplot2(.,expr_DEG_limma_GSEA_go@result$ID,subplots = 3) ->bottom_plot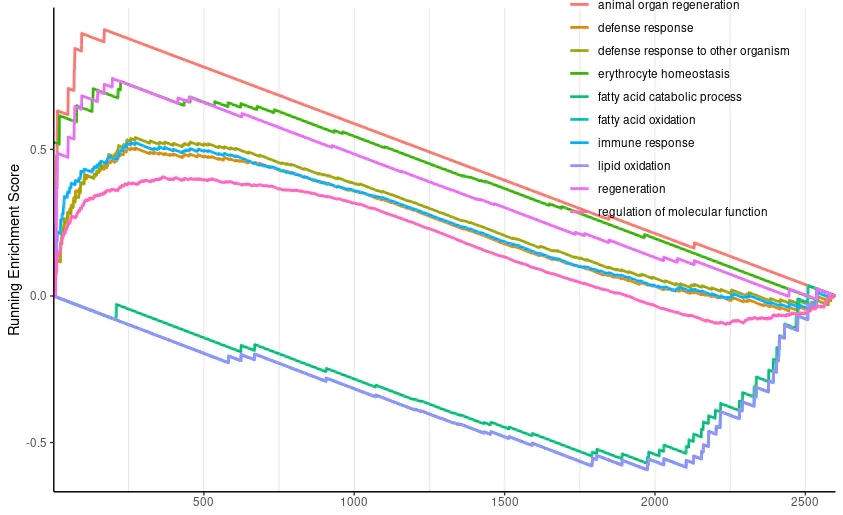
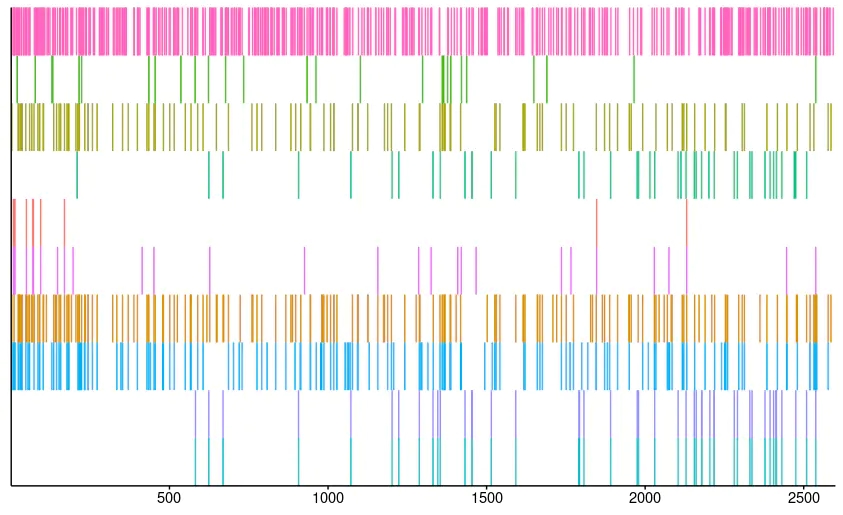
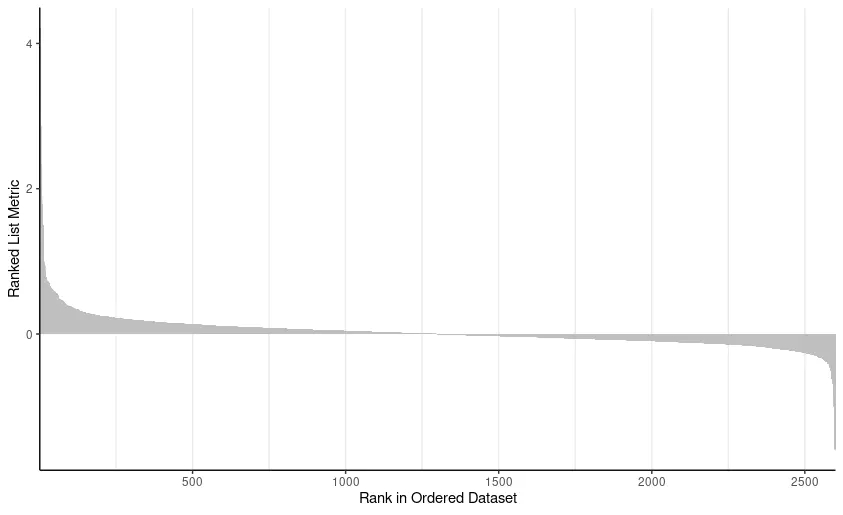
都是ggplot图形,分别修改后再拼图即可
当时我觉得y叔可以优化的地方是增加NES的显示,而不仅仅是P值。
请问GSEA出现missing value TRUE/FALSE needed 怎么解决呢
不清楚具体哪一步,方便的话加微信看看
博主您好,用clusterprofiler这个包做GSEA分析,当我想自定义通路名画图时有时候会弹出“missing value where TRUE/FALSE needed”这个错误(但是有时候重新做GSEA analysis,重新选通路时又不会出错),我查了一下发现GitHub上也有人出现这个问题,但是没有一个很好的解释,请问您遇到过吗?这是什么原因呢?
GitHub:https://github.com/YuLab-SMU/clusterProfiler/issues/513
https://github.com/YuLab-SMU/clusterProfiler/issues/307
我的R代码:
#读入基因集文件
gmt=read.gmt(gmtFile)
#富集分析
kk=GSEA(logFC, TERM2GENE=gmt, pvalueCutoff = 1)
kkTab=as.data.frame(kk)
kkTab=kkTab[kkTab$pvalue<0.05,]
#kkTab=kkTab[kkTab$p.adjust<0.05,]
#write.table(kkTab,file="GSEA.result-GO.txt",sep="\t",quote=F,row.names = F)
write.csv(kkTab,file="GSEA.result-GO.csv",quote=F,row.names = F)
#输出自己想要的通路图片
my=read.csv("myGO.csv", header=T, check.names=F)
my=as.matrix(my)
rownames(my)=my[,1]
mys=my[,2:ncol(my)]
showmy=row.names(mys)
myplot=gseaplot2(kk, showmy, base_size=8, title="") #这一步有时候会出错 有时候却正常
pdf(file="GSEA-GO-myself.pdf", width=10, height=8)
print(myplot)
dev.off()
PS.有时候我把myGO.csv的通路结果换成其他的然后重新保存,却又正常了,不知道什么情况?
若能解答非常感激!
这个我倒是没有遇到过 要不你加我微信 我远程看看 讨论一下