很多时候,在用中位数/均数、四分位数等特殊位置为截点时,生存分析往往没有差异。这个时候常常需要找到一个最佳截点使得生存分析有差异。常用的选取生存分析的最佳截断有很多种方法,如X-tile软件、R的CatPredi包、cutoff包。今天给大家介绍的是survminer 包的surv_cutpoint()函数。
载入示例数据
# 0. Load some data
library(survival)
library(survminer)
data(myeloma)
head(myeloma)
> head(myeloma)
molecular_group chr1q21_status treatment event time CCND1
GSM50986 Cyclin D-1 3 copies TT2 0 69.24 9908.4
GSM50988 Cyclin D-2 2 copies TT2 0 66.43 16698.8
GSM50989 MMSET 2 copies TT2 0 66.50 294.5
GSM50990 MMSET 3 copies TT2 1 42.67 241.9
GSM50991 MAF <NA> TT2 0 65.00 472.6
GSM50992 Hyperdiploid 2 copies TT2 0 65.20 664.1
CRIM1 DEPDC1 IRF4 TP53 WHSC1
GSM50986 420.9 523.5 16156.5 10.0 261.9
GSM50988 52.0 21.1 16946.2 1056.9 363.8
GSM50989 617.9 192.9 8903.9 1762.8 10042.9
GSM50990 11.9 184.7 11894.7 946.8 4931.0
GSM50991 38.8 212.0 7563.1 361.4 165.0
GSM50992 16.9 341.6 16023.4 2096.3 569.2
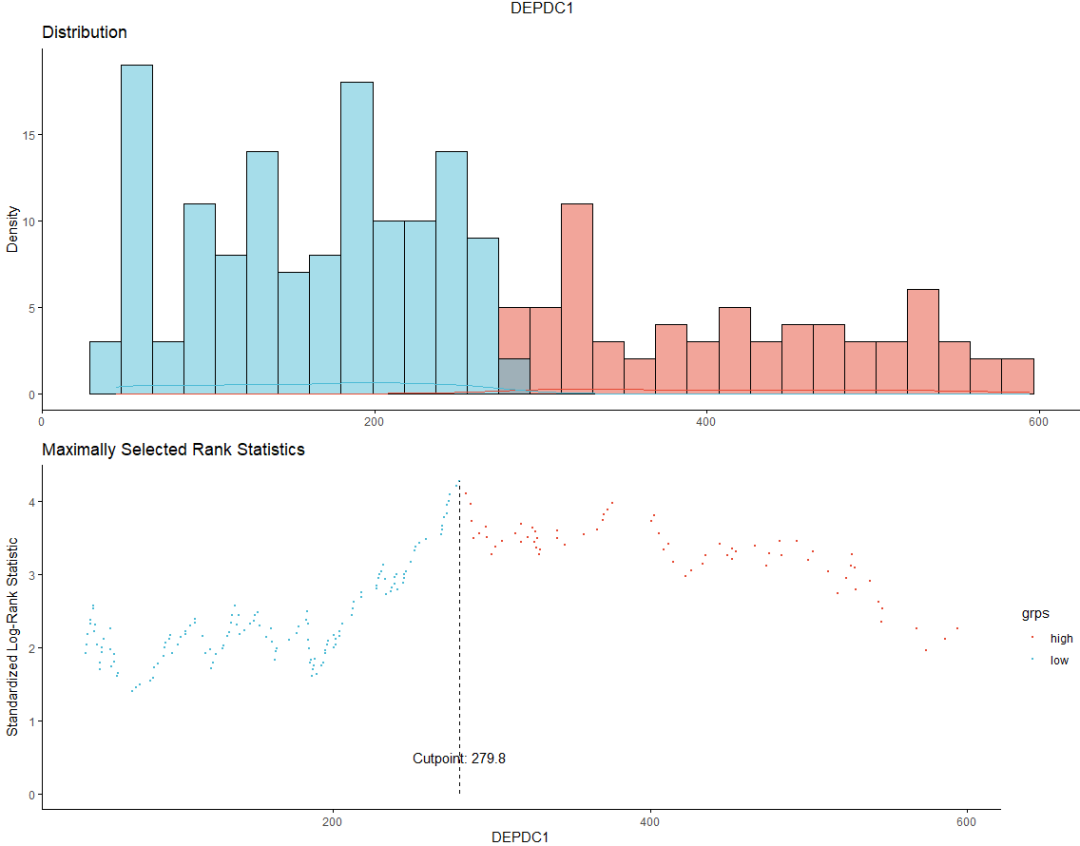
计算最佳截点
# 1. Determine the optimal cutpoint of variables
res.cut <- surv_cutpoint(myeloma, #数据集
time = "time", #生存状态
event = "event", #生存时间
variables = c("DEPDC1", "WHSC1", "CRIM1") #需要计算的数据列名
)
summary(res.cut) #查看数据最佳截断点及统计量
> summary(res.cut)
cutpoint statistic
DEPDC1 279.8 4.275452
WHSC1 3205.6 3.361330
CRIM1 82.3 1.968317
展示数据分布
# 2. Plot cutpoint for DEPDC1
# 以DEPDC1为例
plot(res.cut, "DEPDC1", palette = "npg")
根据截点分类数据
# 3. Categorize variables
res.cat <- surv_categorize(res.cut)
head(res.cat)
> head(res.cat)
time event DEPDC1 WHSC1 CRIM1
GSM50986 69.24 0 high low high
GSM50988 66.43 0 low low low
GSM50989 66.50 0 low high high
GSM50990 42.67 1 low high low
GSM50991 65.00 0 low low low
GSM50992 65.20 0 high low low
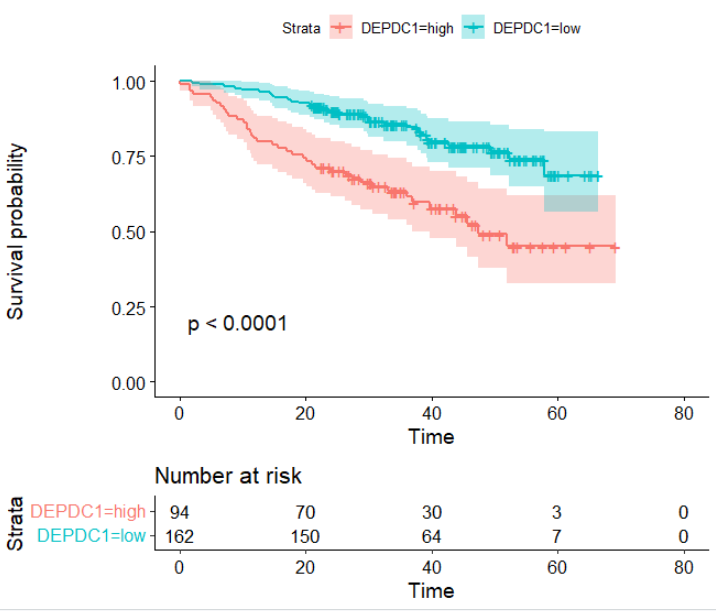
绘制生存曲线
fit <- survfit(Surv(time, event) ~DEPDC1, data = res.cat)#拟合生存分析
#绘制生存曲线并显示P值
ggsurvplot(fit,
data = res.cat,
risk.table = TRUE,
pval = T)
参考
surv_cutpoint: Determine the Optimal Cutpoint for Continuous Variables in survminer: Drawing Survival Curves using ‘ggplot2’ (rdrr.io)
进哥哥,你好,我这两天在复现一篇estimate 评分与临床预后模型的关系,其中就是分析基质评分,免疫评分等分别对os 和dfs 的影响关系。就很像这篇文章里面的生存图片,就是把DEPDC1high和low 换成IMmune scores high和low ,就一摸一样了。所以我在想是不是也可以这样去做评分后的生存预后分析,但是,文献中又提到os ,immune score高分组,km分析,p>0.52。
困扰我两天两夜了,都不知道怎么进行这一步,进哥哥能不能拯救一下我
你好,没太明白你的意思 要不加微信 或者电话讨论 18021308280
你好,进哥哥
我在用自己的数据跑上面的代码的时候,在 ggsurvplot里加入了一行conf.int = TRUE来显示95%置信区间,但是我的结果只有低表达组显示了95%置信区间,高表达组没有,请问你有没有遇到过这样的情况呢?同样的代码用测试数据显示正常,可能是数据的问题,但是我实在搞不懂是出了什么问题。。。
老师您好,因为还是R语言入门级别的菜鸟,冒昧问下能否请求得到示例数据进行复现学习?主要是手中没有具体的数据。
你好 当然可以 其实这些包大多数都有测试数据的,用data(package = .packages(TRUE))查看测试数据,用于练习
好的,明白了。谢谢
计算最佳截点部分中,最佳截断点后面的统计量是指什么?
surv_cutpoint计算的统计量不是logrank的P值对应的卡方值,而是基于maxstat包的maxstat.text函数计算出的Maximally selected rank statistics ,可以查看原包的参考文献