一、CPTAC数据库功能介绍
和TCGA数据库一样,CPTAC数据库也是NIH的一个项目。其中主要提供了不同癌症类型的蛋白质组学数据,此外还包含基因组测序、miRNA测序和DNA甲基化数据等,是不是很棒,一下子可以做多个水平的多组学联合分析。其中基因组数据包含总计1300+不同类型肿瘤病人的WGS、WES和RNA-seq数据,可通过GDC Data Portal访问,也就是咱们下载TCGA数据库的界面。
蛋白组数据可通过PDC 访问(https://pdc.cancer.gov/pdc/browse),CPTAC数据库用到的蛋白质定量技术主要是基于质谱的检测技术,包括ITRAQ和TMT。
收集的信息如下:
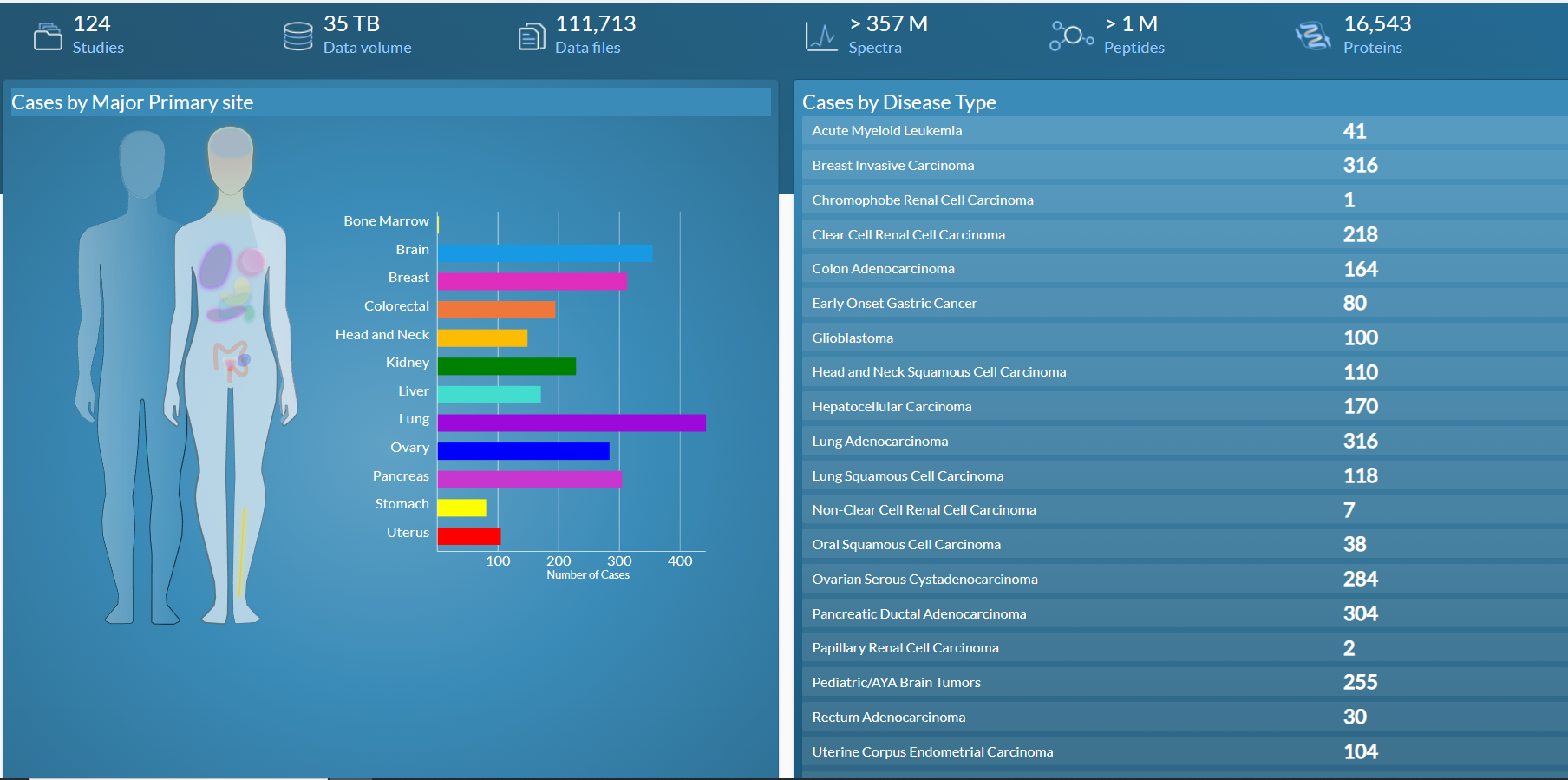
此外,CPTAC数据库还提供了生物样品的元数据和临床数据,例如病人的性别、年龄、癌症类型和临床治疗记录等。分析工具和生物信息学资源包括了一系列数据处理和分析工具等。CPTAC数据库的综合性质使其成为肿瘤分子分析的重要数据来源。
二、CPTAC数据库使用方法
1. 数据库访问
用可以通过网站(https://pdc.cancer.gov/pdc/browse)访问该数据库,下载过TCGA数据库的都应该很熟悉,基本差不多。
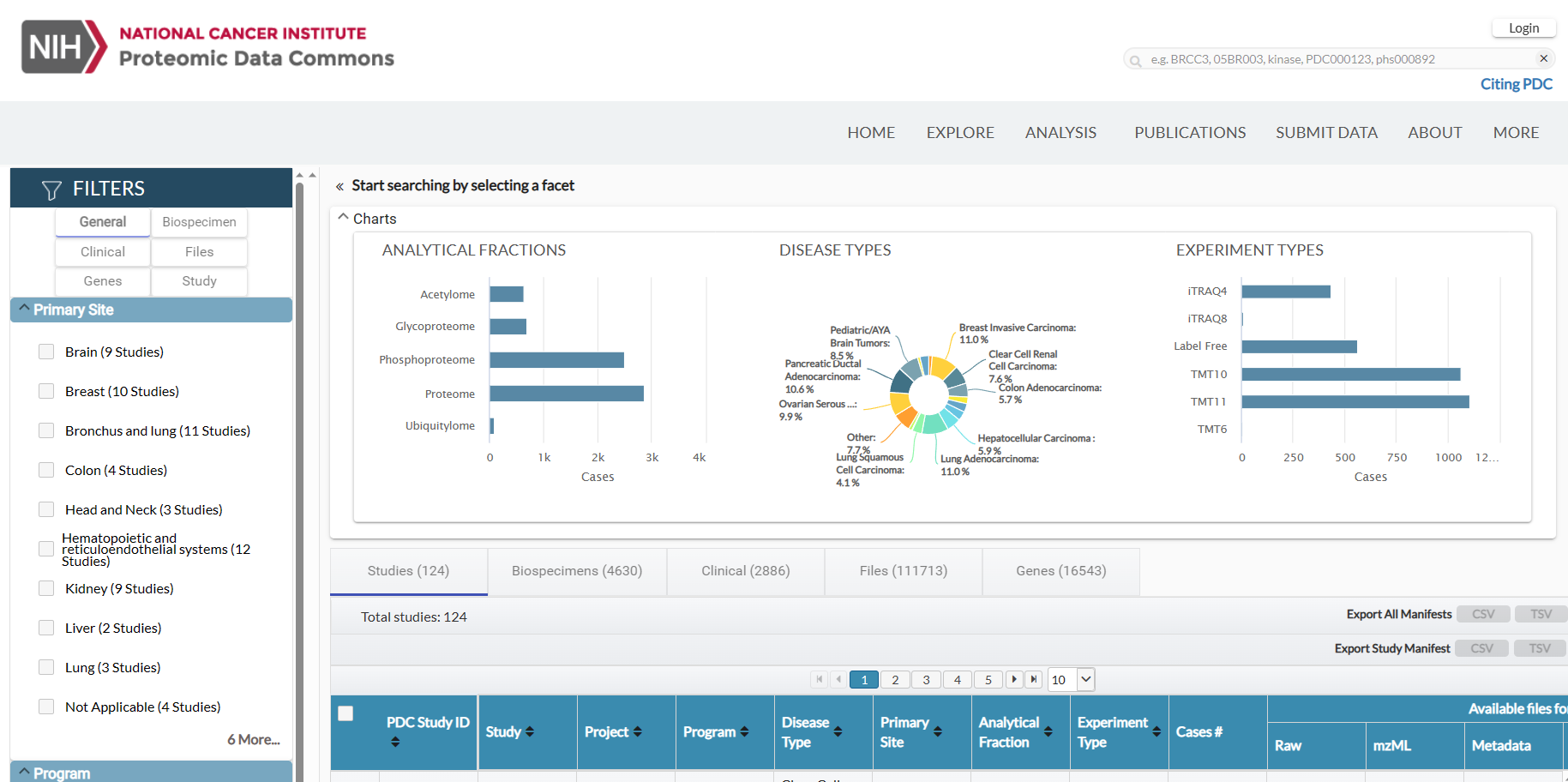
包含完善的统计信息和数据集信息,点击PDC Study ID查看详细信息:
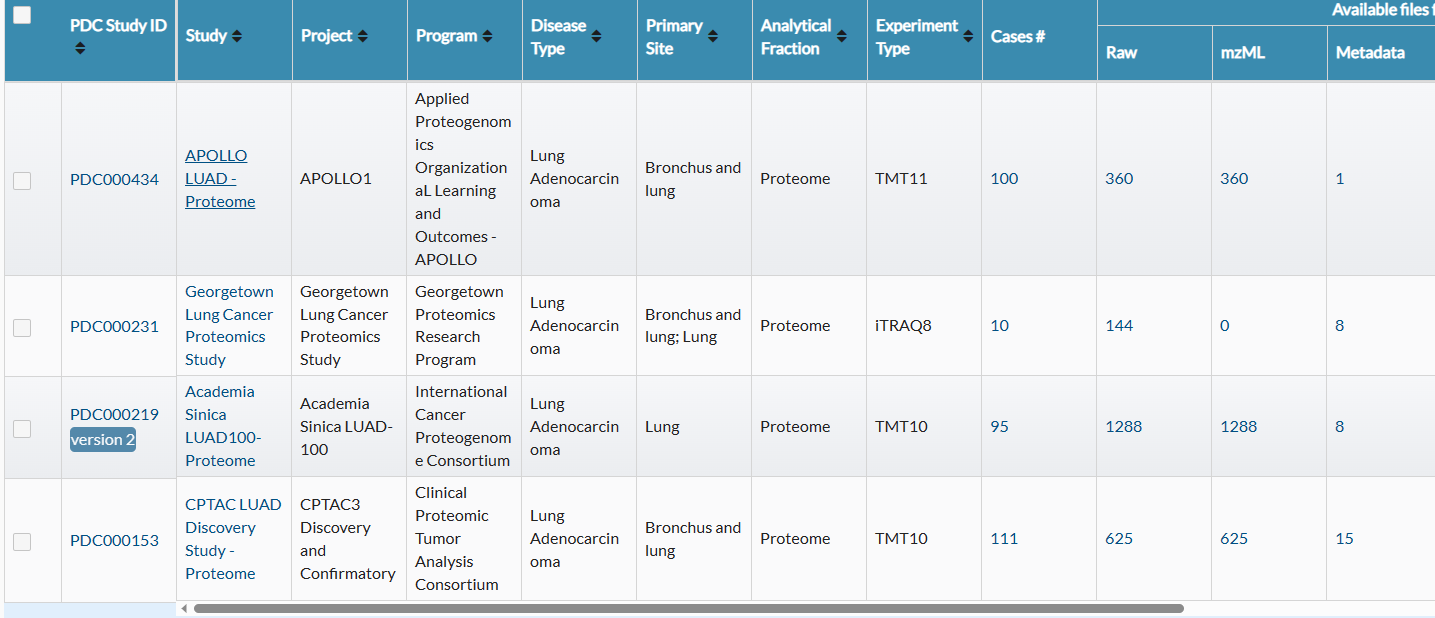
以CPTAC LUAD为例,点进去可以看到每个研究项目的详细信息:

如果需要下载原始数据信息点击Files下面的数字625,下载处理好整合好的数据点击6:
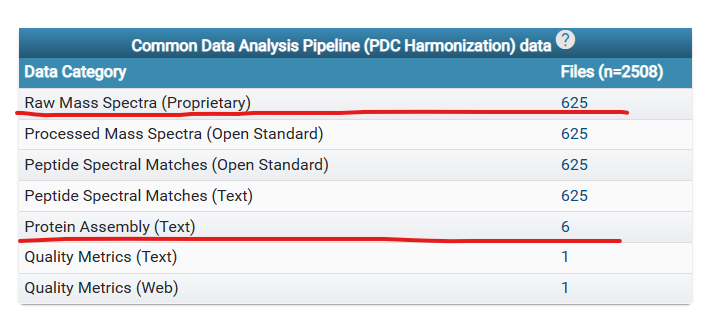
点击Protein assembly之后,如下图可以下载整合好的数据:
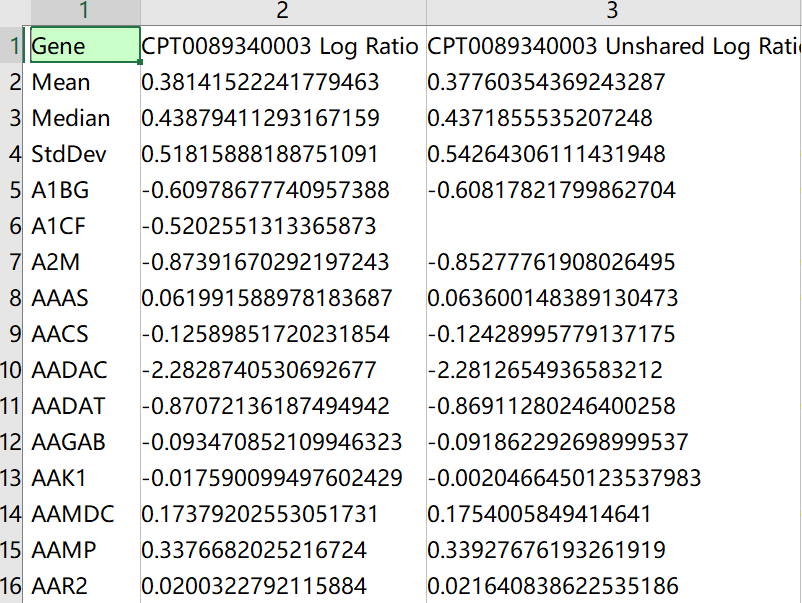
打开就可以看到咱们无比熟悉亲切的矩阵,需要注意的是数据中包含log Ratio 和Unshared log ratio,咱们只需要Unshared这个数据,需要用R语言提取一下。 然后就可以开心的进行分析了。
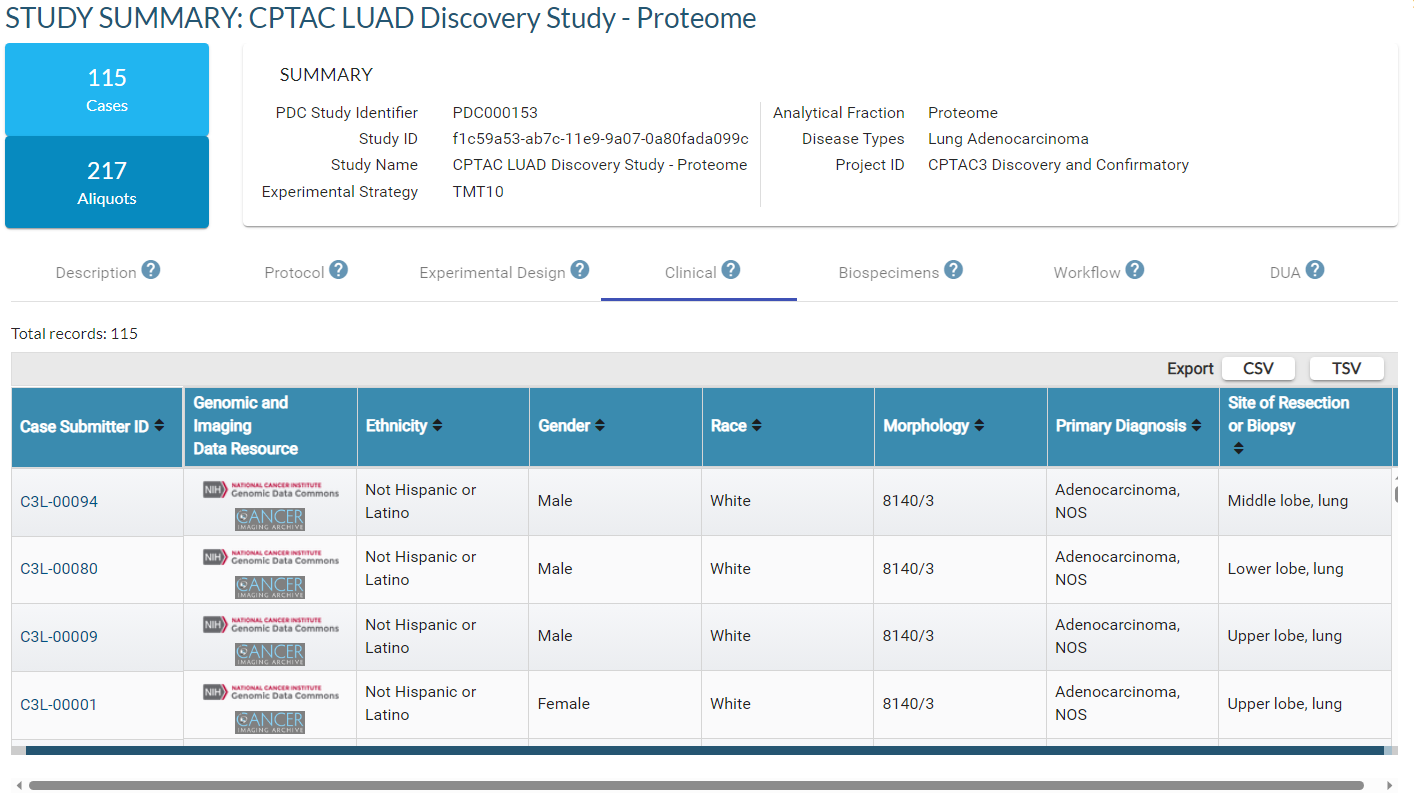
对于Clinical data,如下图可下载:
2. 分析工具
CPTAC数据库还提供了一些分析工具,方便用户对蛋白质组学数据进行可视化。

(1)Explore Quantitation Data
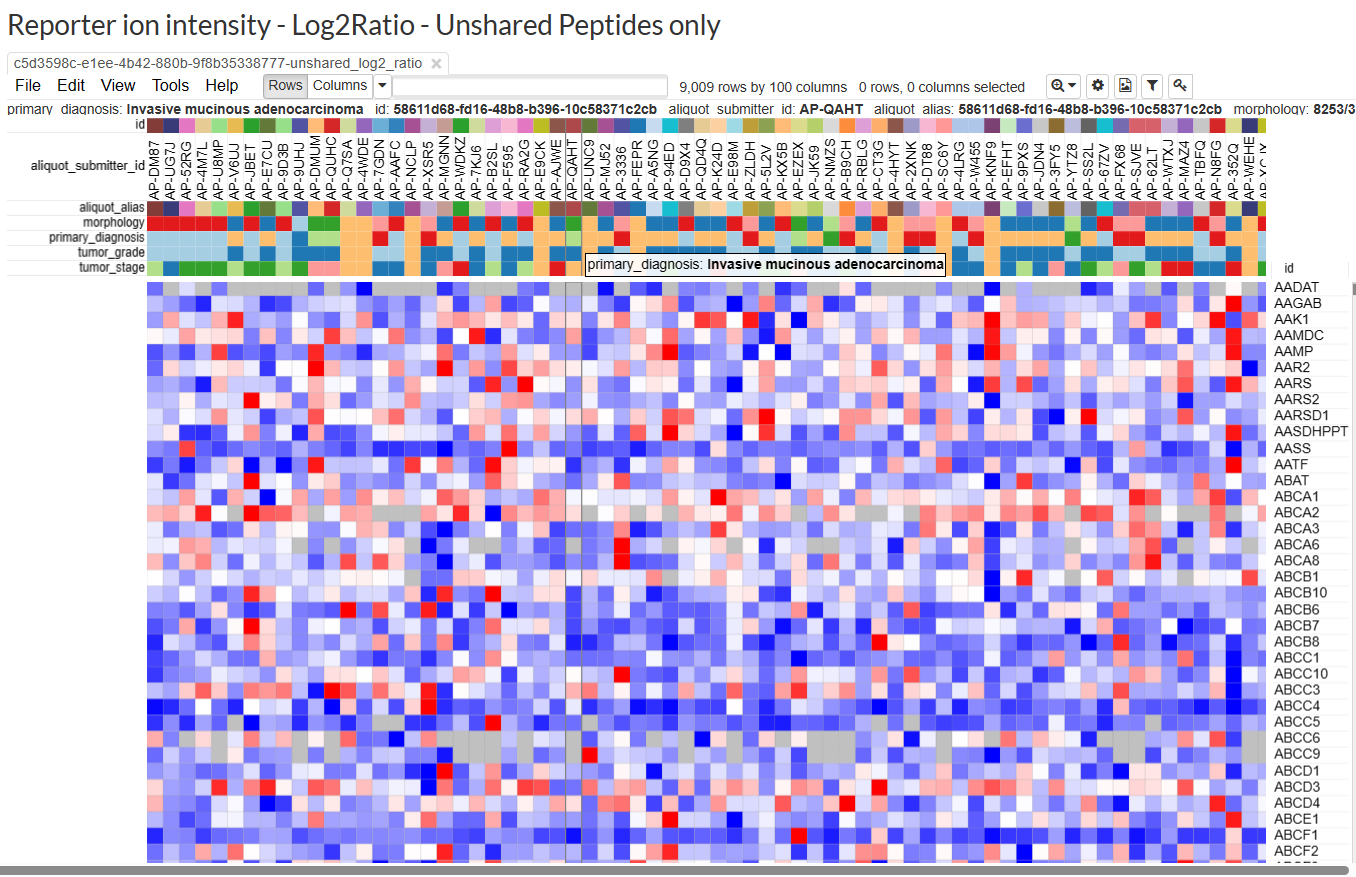
PDC通过标准分析流程产生结果后可用于鉴别蛋白质和翻译后修饰(PTMs)丰度的pattern,并通过热图展示。Explore Quantitation Data 可对每个study的蛋白定量结果进行分析和展示。其中行为基因(蛋白),列为样本,并可嵌入临床信息。可以调整很多参数,大家自行摸索。
(2)Peptide Genome mapping
主要是各种类型数据在基因组上的可视化展示。
(3)Pepquery
这是一个以肽段序列为中心的搜索,与Blast类似,Pepquery使用户能够在MS数据库中查询感兴趣的新的肽段或DNA序列。基因组改变导致产生新的蛋白序列,经过蛋白组学验证,这些新的序列可能会是潜在的疾病标志物或治疗靶标。Pepquery不需要定制化的构建数据库,区别于spectrum-centric的方法,使用peptide-centric,允许快速和方便的验证基因组改变导致的蛋白质组改变。
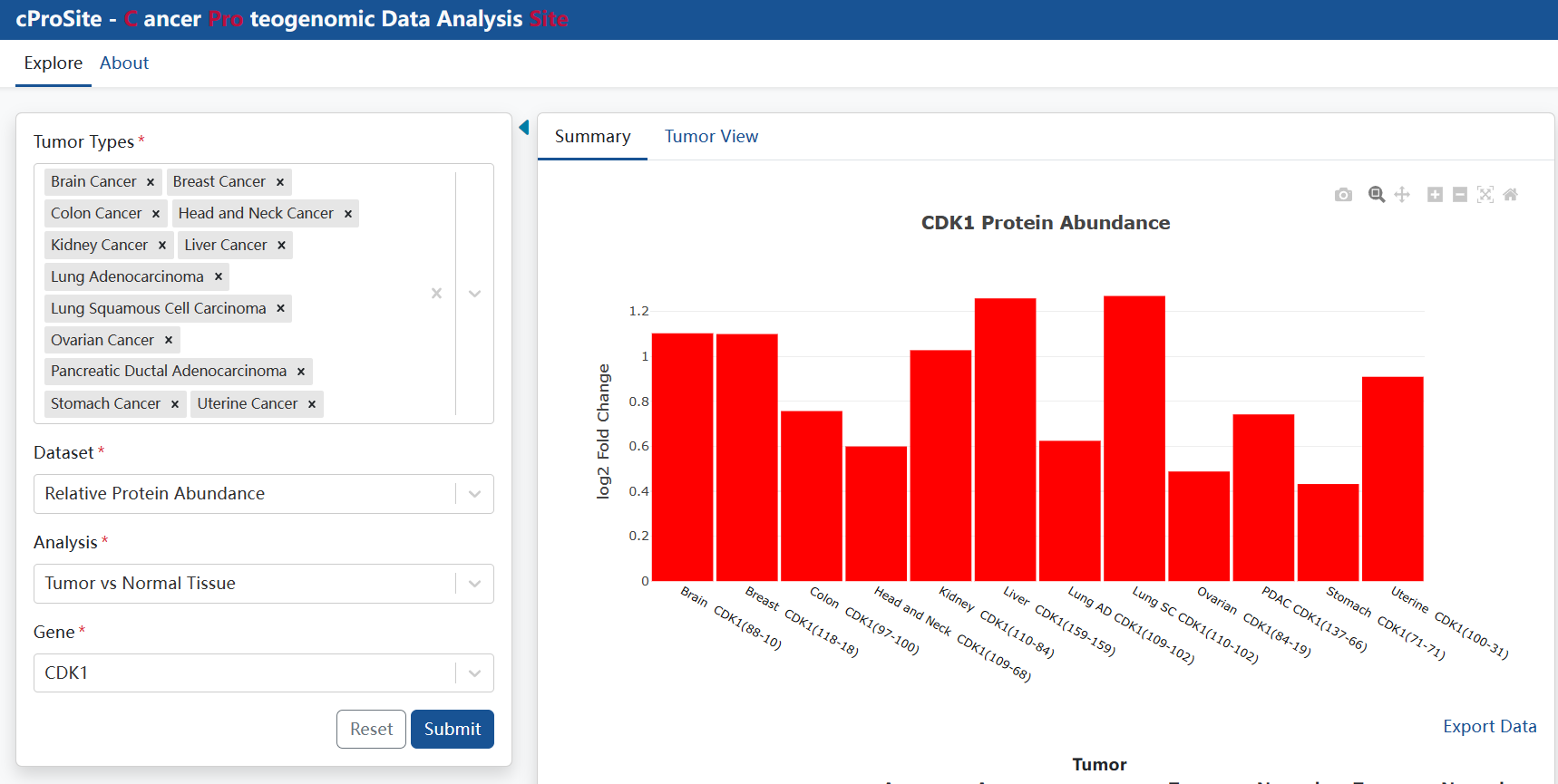
(4)cProSite
可指定肿瘤类型和数据集,针对单个基因,分析其在肿瘤和癌旁组织的丰度差异,磷酸化位点差异等。可用于验证目标基因是否在指定类型肿瘤中存在高表达,磷酸化等。也可以选择多种癌症类型进行泛癌分析:
寻求CPTAC数据库基因差异分析和生存分析的R代码
这个只要数据有了 和TCGA差不多,我已经更新了这个文章,加了我做的数据下载的小程序