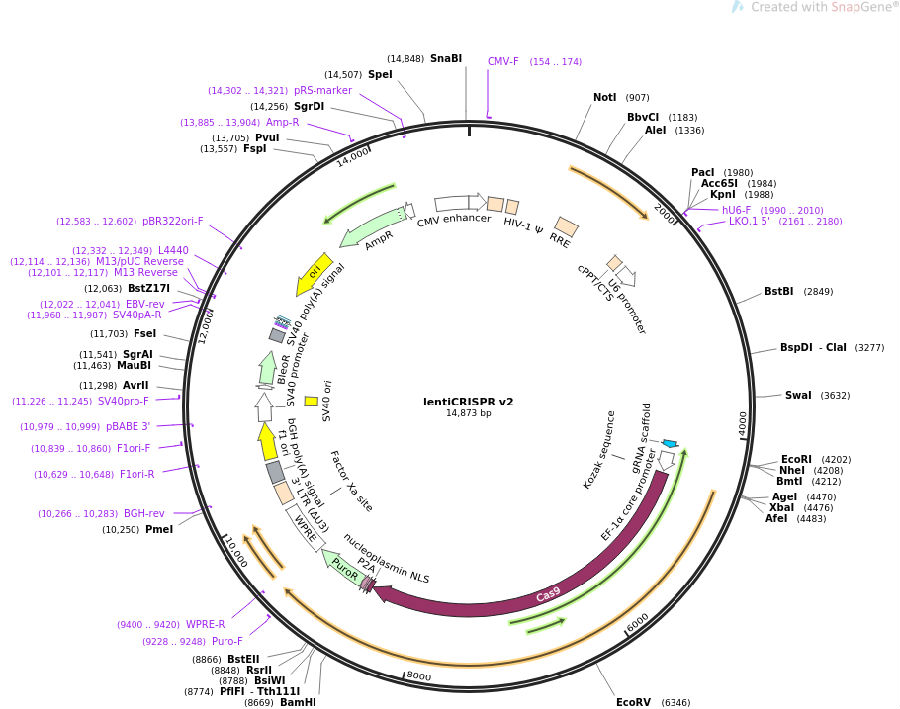
一 载体简要说明
1 克隆gDNA使用BsmBI酶切位点,载体可以切出1.9kb的filter,便于确定载体酶切成功,并且利于胶回收。
2 BsmBI酶切位点如图酶切后不需要用CIP脱磷,也不会自连。
3在cas9的C端有FLAG,可以WB或Immunostaining来检测。
4 EFS被优化的小而强,方便慢病毒的包装,极大的提高了慢病毒包装的效率。
5 优化的内部核糖体插入位点P2A,可以在只有EFS启动的情况下独立表达puromycin来筛选转染或感染成功的细胞。
二 gDNA设计说明
对于lentiCRISPR V2克隆,其合成的oligo末端与px330不同如下图所示:
具体举例如下:

一定注意oligo的方向,载体的hU6的下游正义链一定是基因组序列的有义链,否则编辑无效,且5’端必须第一个碱基必须是G如果不是G也要认为加一个G满足U6启动子的需求。
将引物稀释 100uM 后,再 10 倍稀释
退火
50ul 体系
引物 20ul(上下游各 10ul)、水 25ul、NEB buffer 2(10*)5ul 95℃,5min。然后关闭 PCR 仪,自然降温 40min。
LentiCRISPR V2 载体酶切
200ul体系:
BsmBI 2ul;10*buffer 20ul; 质粒 10ul(2ug);水 16ul 55℃,酶切 2 小时 胶回收
连接
2*solutionI 5ul
退火引物 4.5ul
线性化载体 0.5ul 16℃连接过夜,转化挑菌送测序。
进哥,我想问一下lentiCRISPR v2质粒,可以用来电转吗
您好,想请问LentiCRISPR V2包装慢病毒时,三质粒的转染比例是取的多少呢?
(2)A管加入60µg PEI,并充分涡旋混匀,B管加入过表达质粒和两个辅助质粒pSPAX2、pMD2.G,三质粒比例为4:3:1,共24µg;
在这里的体系:
稳定细胞株构建:第一讲 – 王进的个人网站
https://www.jingege.wang/2020/06/24/%e7%a8%b3%e5%ae%9a%e7%bb%86%e8%83%9e%e6%a0%aa%e6%9e%84%e5%bb%ba%ef%bc%9a%e7%ac%ac%e4%b8%80%e8%ae%b2/
这个酶切是55度吗,温度这么高
不好意思 这是搬过来的方法,可能有误。
请参考这个:CRISPR-Cas9 sgRNA设计和载体构建
王老师,您好,我想请问一下您有推荐用来设计oligo的软件吗?我在Thermo里面有些目的基因的oligo设计不出来
一文掌握gRNA的设计,常用方法汇总解析 – 王进的个人网站
https://www.jingege.wang/2020/12/18/5041/
这几个你都试试看哈 有很多这样的工具
“对于lentiCRISPR V2克隆,其合成的oligo末端与px330不同如下图所示:
具体举例如下:一定注意oligo的方向,载体的hU6的下游正义链一定是基因组序列的有义链,否则编辑无效,”
请问一定U6后为什么一定要是基因有意链呢?sp cas9切割的是基因组的双链,理论上来说不是有意义链也能切割引起突变吧
您好,您说的对,这边描述错了 谢谢提醒 这是网上转过来收藏的 没有仔细看 我现在修改一下
不是 刚刚又看了一下,没有问题 这里强调的是方向问题 只是它的描述容易产生误解。意思是需要U6启动子转录出sgRNA,注意双链oligo的正反,而这个sgRNA靶向的可以是正义或反义链
您好,请问酶切体系是200微升吗?我看您这里写的加起来是48微升。
CRISPR-Cas9 sgRNA设计和载体构建
不好意思,这个文章里有点乱
参考上面那个文章的体系,我用的也是这个
请问做不同种属的细胞敲除,比如质粒上原来是人源的U6启动子,现在想做鼠源细胞的敲除,需要把U6启动子换成鼠源的吗?
RNA聚合酶III识别结合U6启动子,应该是要种属特异性才能保证最大效率
好的,谢谢!
你好 这个LentiCRISPR V2质粒可以分享下吗 谢谢
可以的 加微信交流
我也需要LentiCRISPR V2质粒,可以分享吗,如何加你微信。谢谢
LentiCRISPR V2质粒可以分享下吗?怎么加你微信?
18021308280
大佬您好,请问这个质粒或菌株您还有吗?
您好 大佬不敢 这些质粒当然有的
最近想做这方面试验,求购该质粒以及慢病毒包装的其他几个质粒 谢谢!
你加我微信了是吧 微信联系
请问一下,这个载体的测序引物是EF-1a吗?我在网站里查到的是这个引物。但是我送公司测序都是无信号。谢谢!
你要读懂质粒那个圈的,插入片段的上游有U6 promotor,负责转录sgRNA;下游有EF1-a,负责转录cas9;所以两种选择,U6 forward primer或EF1-a reverse primer
好的,谢谢解答!
您好,我们组LentiV2质粒测序用的引物是LKO1-5,请问为什么选择这个引物呀?分子克隆小白,还请您帮忙解答一下,谢谢啦
您好,建议您下载序列用snapgene软件查看质粒图谱,或者直接在addgene网站查看图谱,我这边简单解释一下:
LKO1-5:GACTATCATATGCTTACCGT 这个引物序列是在U6启动子正义链上,按照5–>3’进行测序,而sgRNA在这个治理上是由U6启动子启动转录的,因此,从U6往下测序就可以确定你的sgRNA是否插入。
我常用的是EF1-a-R,这个引物结合的是EF1-a的反义链,ef-1a位于sgRNA下游,因此EF-1a-R引物往反方向测序也可以证实是否插入
你理解一下 根据图谱不难理解
理解了,谢谢王老师,开心