Snapgene是一款多功能的生物学操作软件,可以帮助用户方便的分析酶切位点、标签、启动子、终止子和复制子等质粒原件,生成详细的DNA序列文件。
这里主要为大家详细介绍:Snapgene在载体构建的应用教程。
链接:https://pan.baidu.com/s/194D8fXJ2w5NdUzafE18GMQ
提取码:s4gk
友情提醒:5.0以上版本似乎有防破解功能,破解后无法正常显示,本人安装4.1.9,没有问题
获取载体序列
以pEGFP-C1构建human p53 CDS过表达载体为例
(1)进入snapgene官网(http://www.snapgene.com/)
(2)点击主菜单栏中的“Resources”,选择“Plasmid Files”
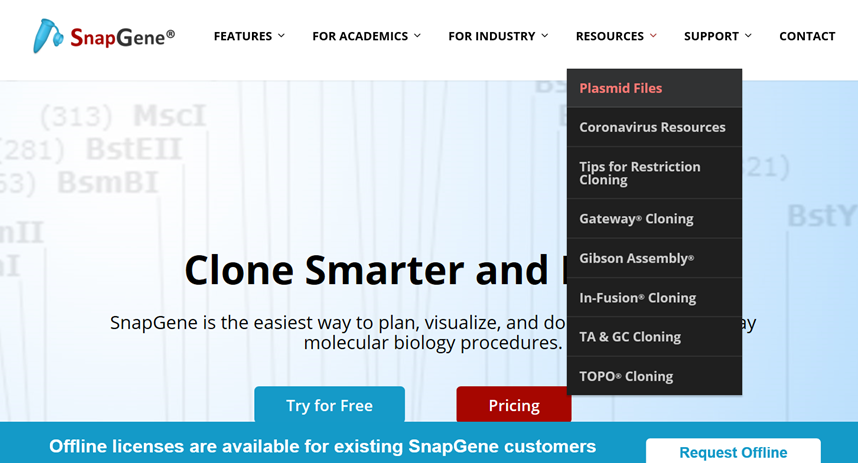
(3)在搜索栏输入载体名称“pEGFP-C1”,搜索
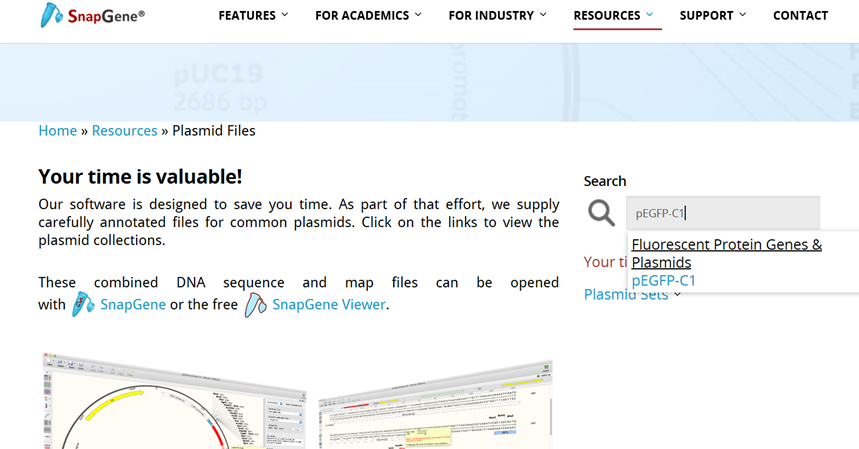
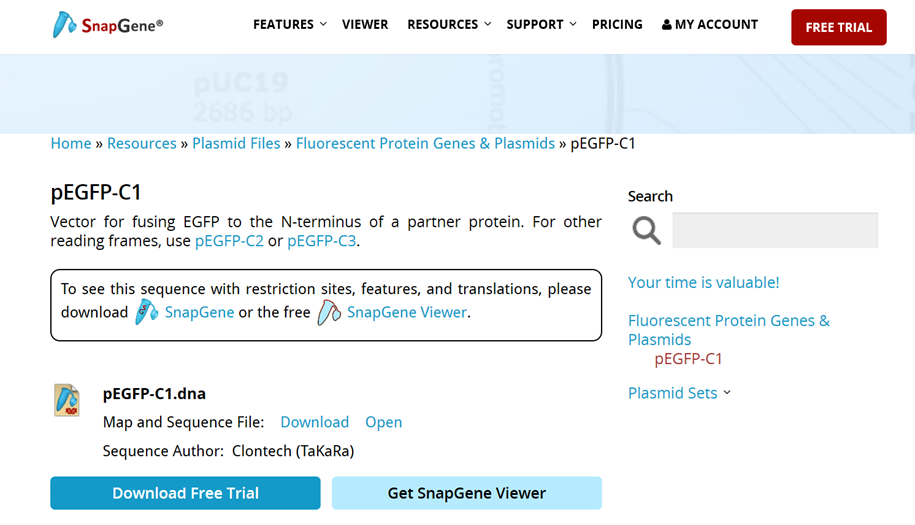
(4)如图所示,点击“Dowload”载体序列即可
酶切位点筛选
以pEGFP-C1构建human p53 CDS过表达载体为例
(1)先从NCBI获取P53的CDS序列(NCBI官网:https://www.ncbi.nlm.nih.gov/nuccore/):进入NCBI后,选择“Nucleotide”,输入“p53”
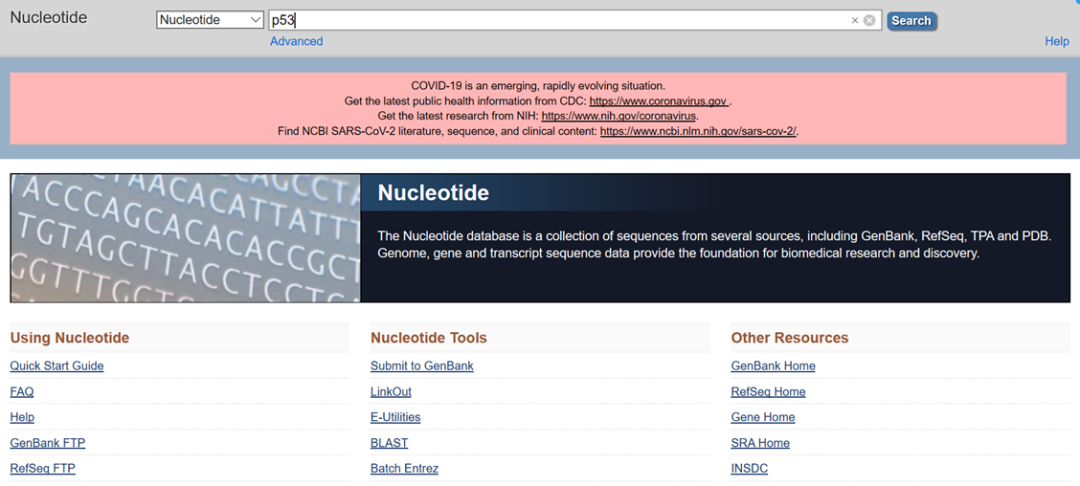
(2)在弹出的以下页面中,选择物种类别、核酸类别等进行筛选
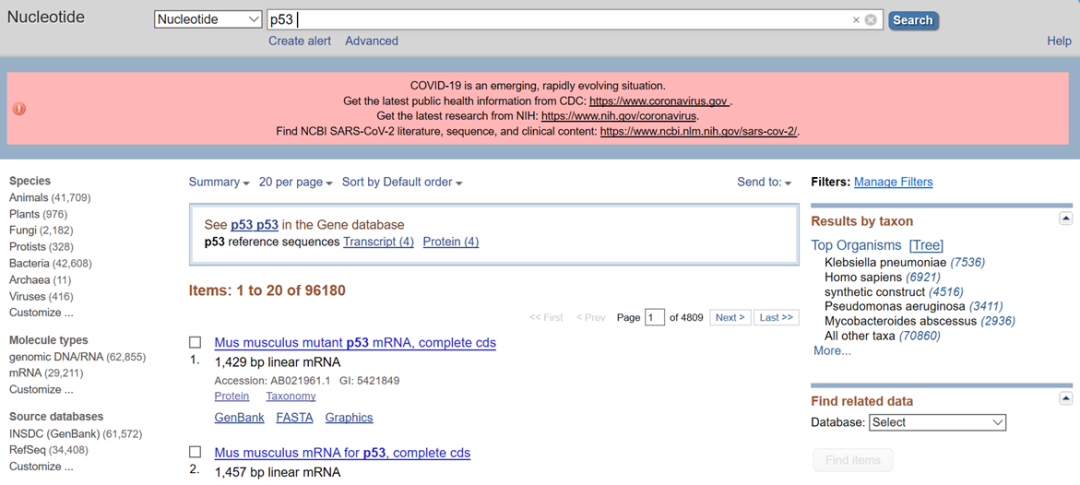
(3)点击进入置顶的这一条(也就是我们所需的人源p53 mRNA)
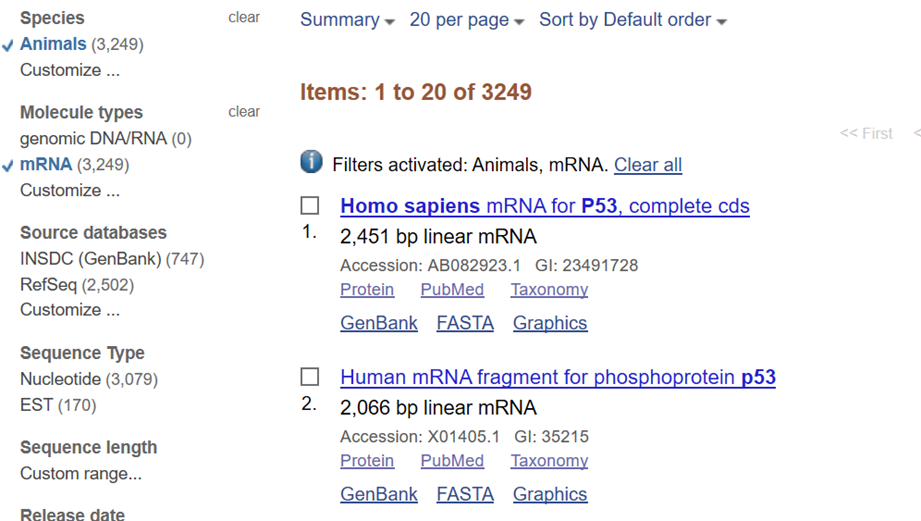
(4)点击“Send to”,选择“Coding Sequences”→“FASTA Nuleotide”,点击“Create File”,即完成目标基因(p53)CDS区序列的下载
(筛选酶切位点的方法:确定载体MCS区有哪些酶不能切割需要插入的目的基因,随后通过设计引物给目的基因两端引入这个不能切割目的基因的酶,从而完成酶切位点的筛选)
(5)打开Snapgene软件,点击“New DNA file”

(6)打开刚才我们下载的p53 CDS序列,复制粘贴到snapgene软件中,根据需要勾选相应的指标,点击“OK”

(7)我们就得到了以下界面(酶切图谱:展示了有哪些酶可以切割目标基因)
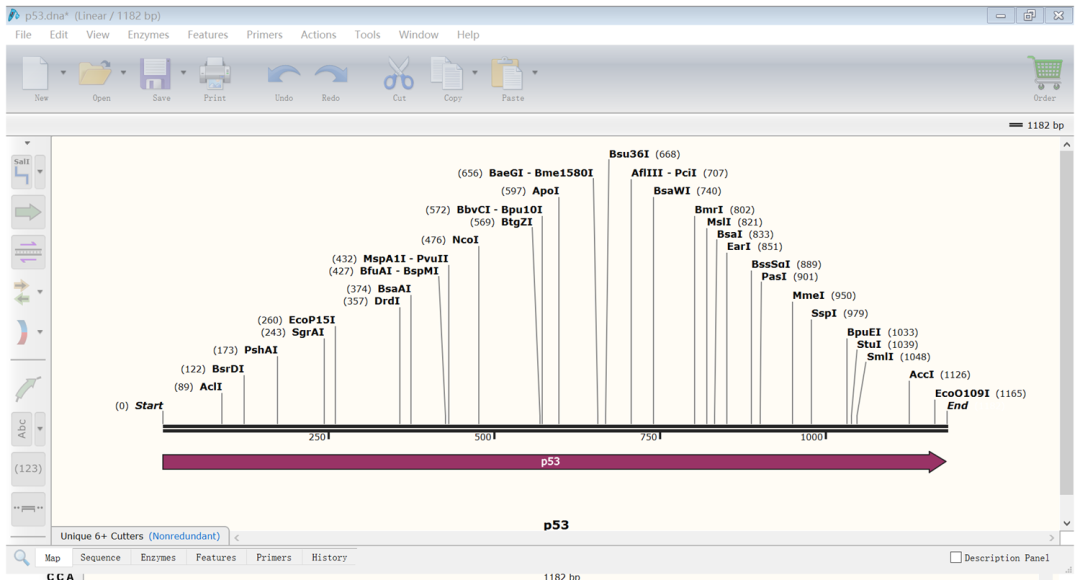
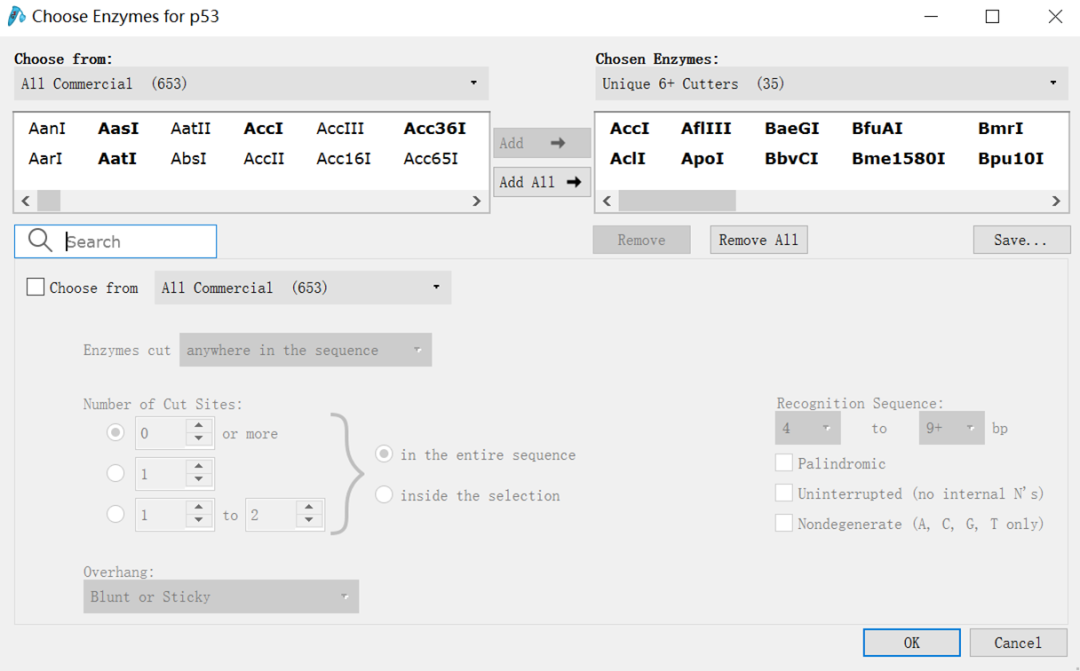
(8)将载体多克隆位点中的酶全部加入显示的图谱当中:点击“Enzymes”→“Choose Enzymes”,在弹出的新的页面中的搜索框中输入载体多克隆位点上酶的名字,点击“Add”添加到右边栏目里,点击“ok”
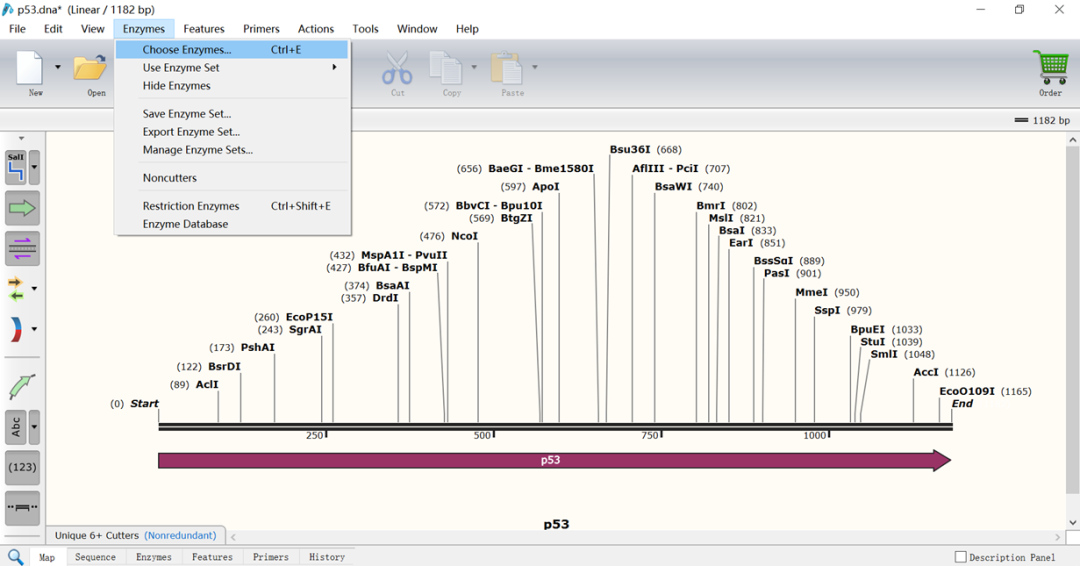
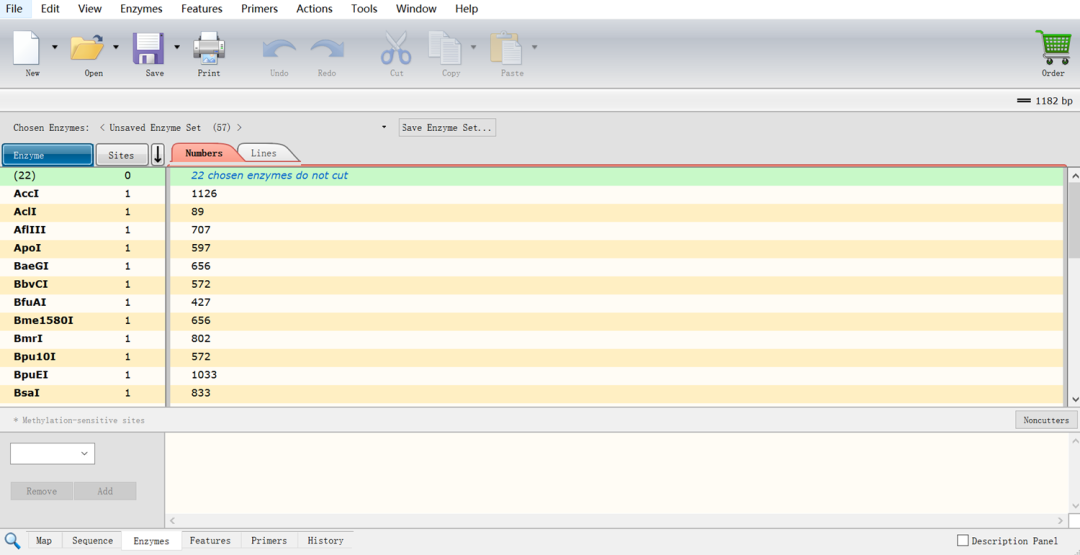
(9)点击底部菜单栏的“Enzymes”,再点击“22 chosen enzymes do not cut”,弹出的页面如下,蓝色标记的即为在载体多克隆位点出现但是在目标序列没有出现的且可以用的酶切位点

引物设计以及模拟克隆
以pEGFP-C1构建human p53 CDS过表达载体为例,通过酶切位点筛选,选择“EcoRI”和“BamHI”两种内切酶,其中需要注意的是:EcoRI在上游,BamHI在下游
(1)按照之前步骤,用snapgene打开p53 CDS序列
(2)选择底部“Sequence”页面
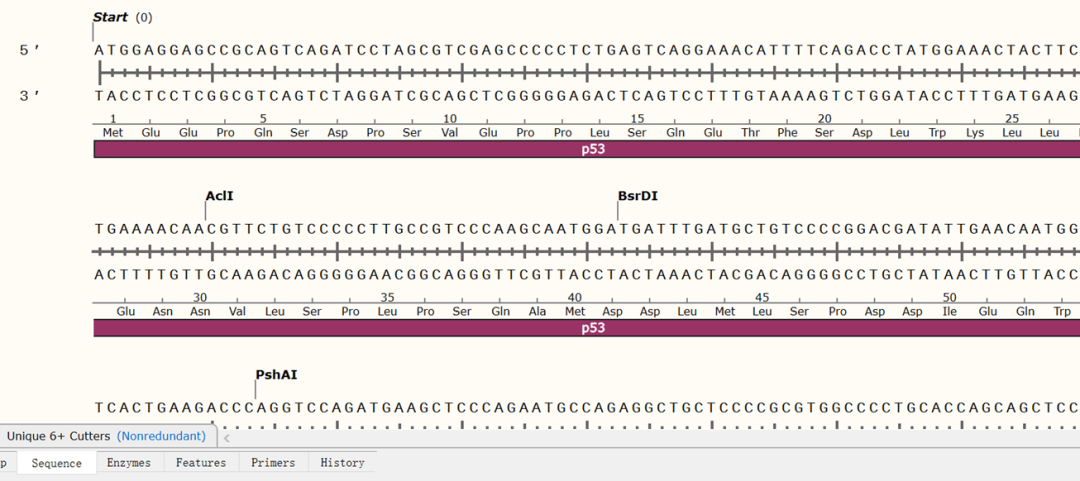
(3)接下来进行引物设计:首先先选上游前20个碱基

(4)点击“Primers”→“add primer”,在弹出的选项框中选择“TOP strand”
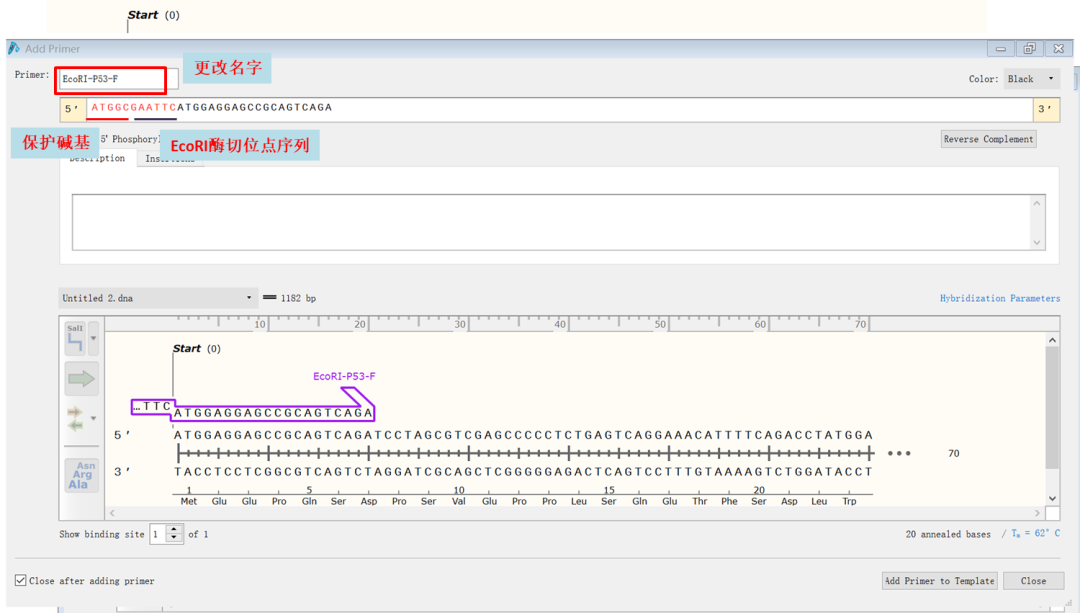
(5)按下图所示,更改上游primer的名字以及添加酶切位点序列(百度或用snapgene软件打开载体序列均可查找到内切酶对应的切割序列)和保护碱基的序列,然后点击“Add primer to template”
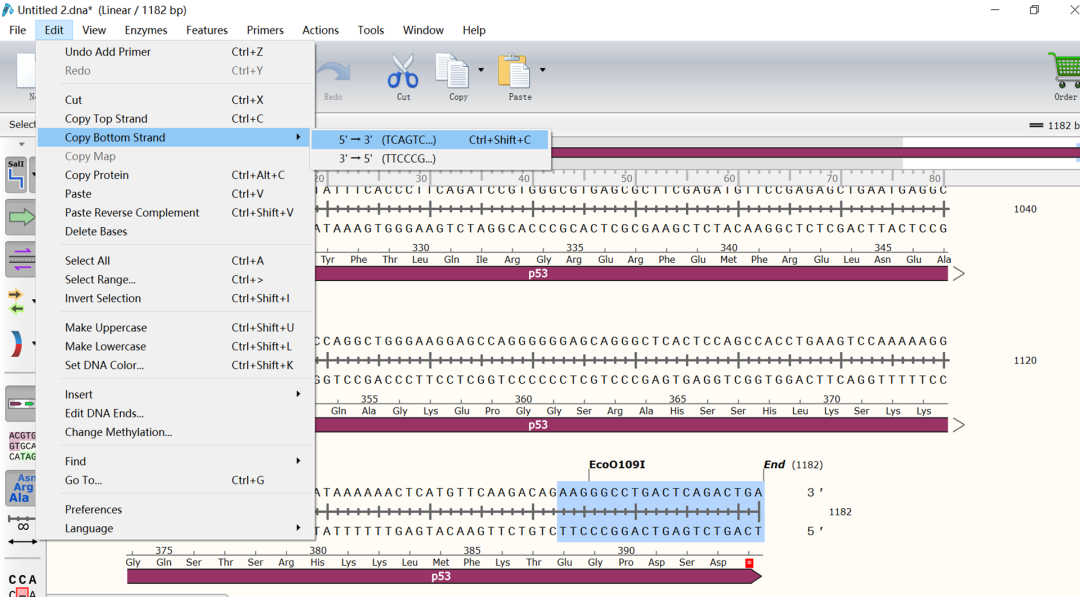
(6)然后选择下游20个碱基,点击“Edit”→“copy bottom strand”,选择“5’→3’”
(7)然后,点击“Primers”→“Add primer”,在弹出的选项框中点击右上角的×,直接关闭
(8)将序列粘贴到序列框中,同理更改名字以及添加酶切位点序列(下游酶切位点是BamHI)和保护碱基的序列,然后点击“add primer to template”
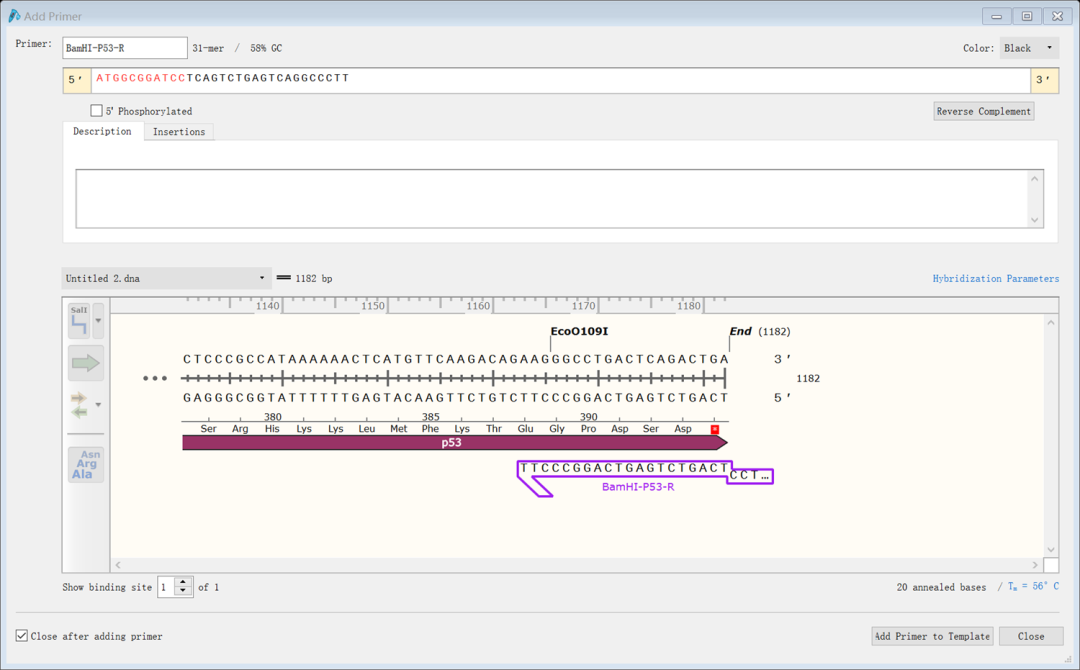
(9)点击“Map”,Ctrl键选择两个引物(如图),点击“Action”→“PCR”
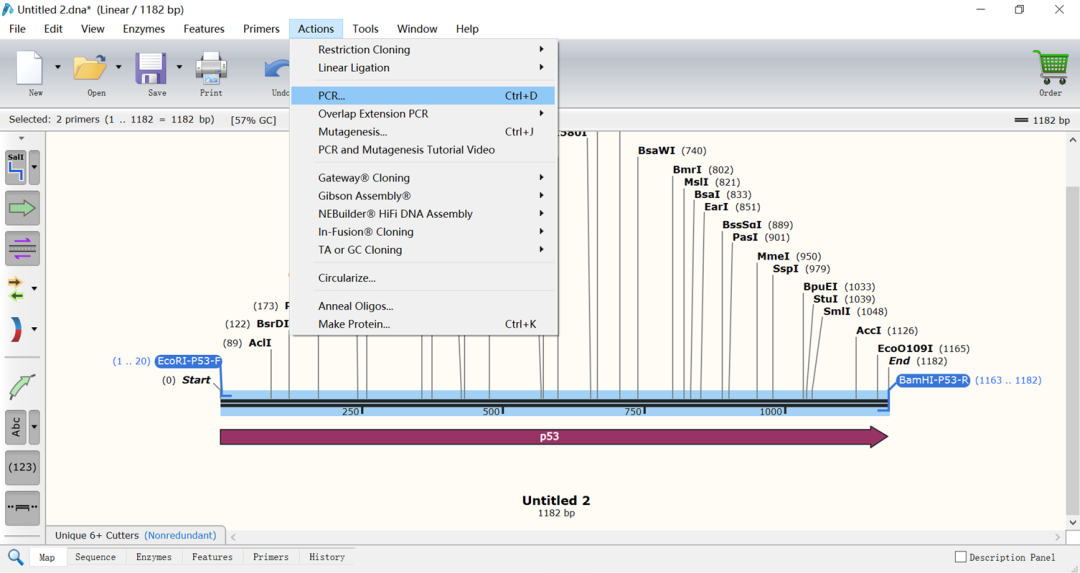
(10)点击“PCR”,即获得扩增产物(如下图所示)
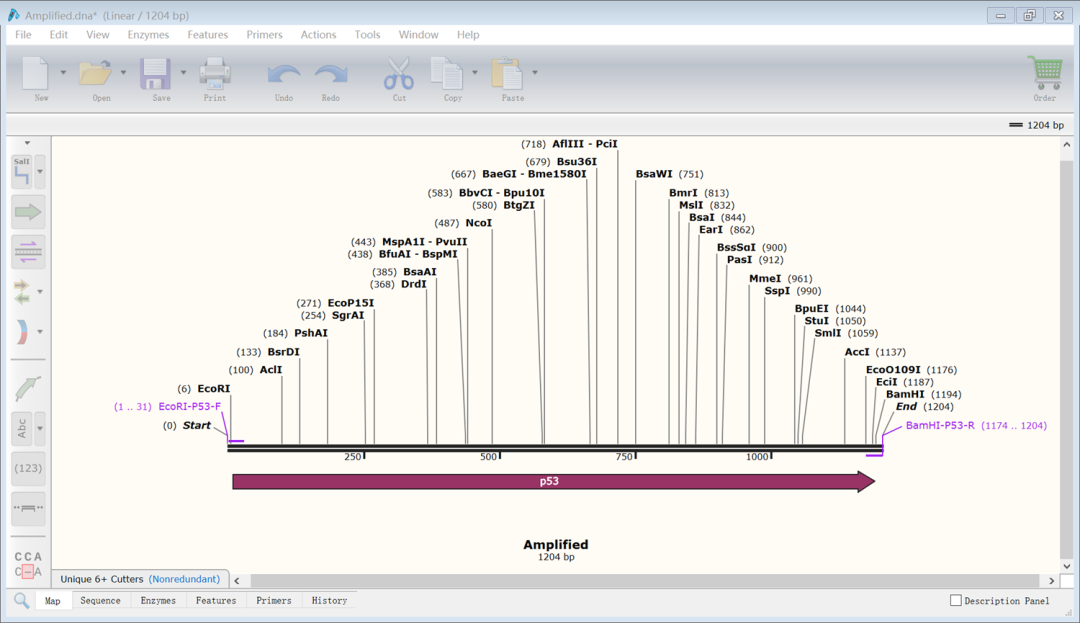
(11)打开表达载体序列,按Ctrl键选择两个酶切位点(蓝色标记的),点击“Action”→“Restriction cloning”→“Insert fragment”
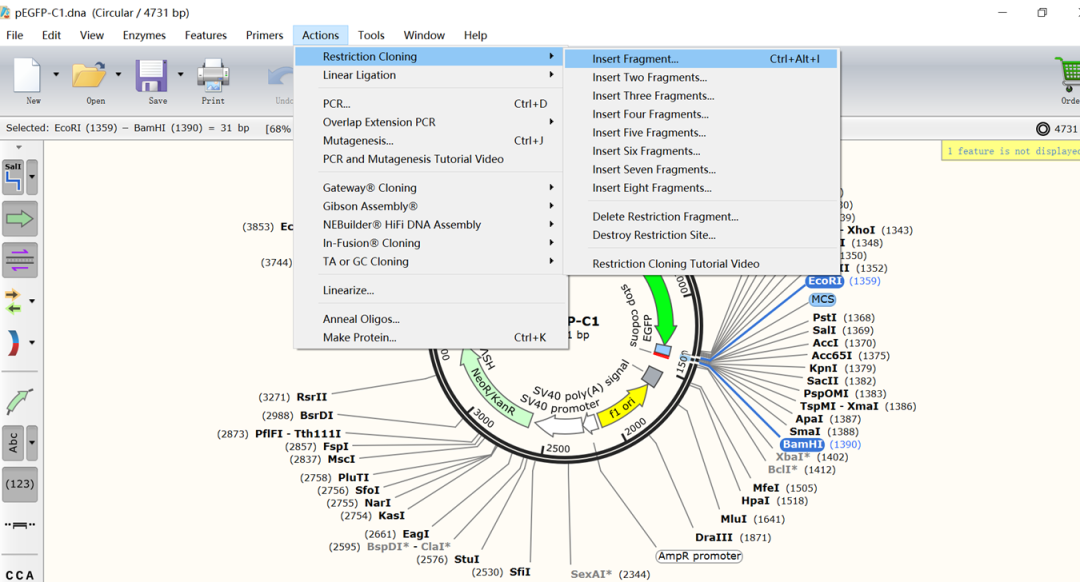
(12)点击“Insert”,选择刚刚扩增产物的文件“Amplified.dna”,然后分别输入相应内切酶的名字,点击插入的片段
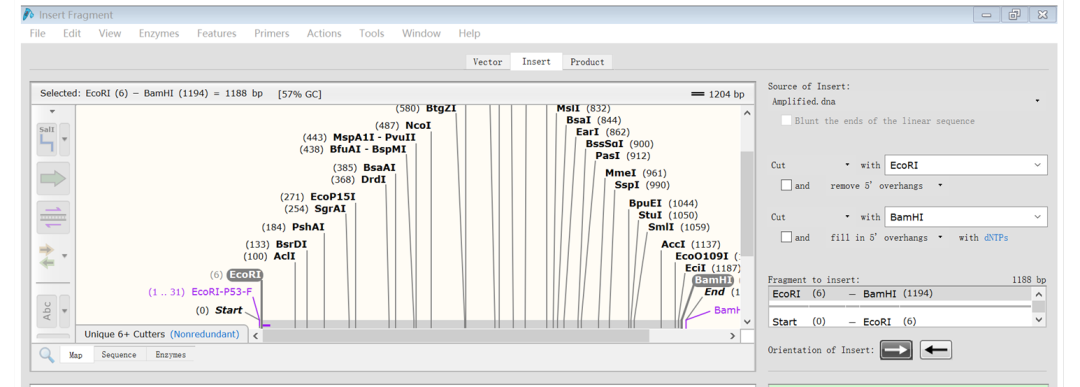
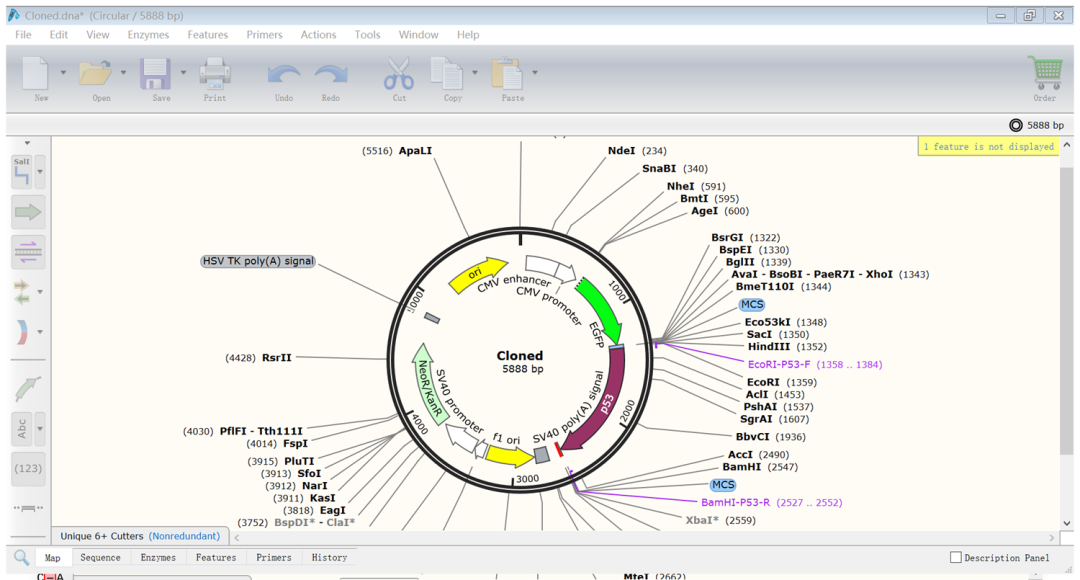
(13)最后在右下角点击“Clone”即可,结果如下
请问我一直不能打开文件 可以帮忙看看吗
博主你好,我看你用的保护碱基和常用的保护碱基不同,请问是自己设计的嘛,设计原则是怎样的?期待回复
显示载体查找不到是怎么回事?
您好,我下载好压缩包之后,4.1.9版可以解压,但是解压后的文件里没有看到有软件包,重新解压之后发现软件包只会出现一分钟,在点击安装的时候显示由于权限问题不能安装,然后自己消失不见;4.2.4版则直接不能解压,压缩包下载好后一分钟直接原地消失,这是为什么呢?
师兄,后面的图打不开了看不了流程,能解决一下么,感谢
刷新一下就好
安装之后点add feature就闪退是什么原因呢?
我没有这样的报错(4.1.9版本) ,不清楚诶 抱歉
我也是这样,您解决了吗?
学长你好,全文有MAC版本的安装包嘛
链接:https://pan.baidu.com/s/16TQ-wpcCxbnNAsH2guZl0g?pwd=sj7x
提取码:sj7x
你试试看
你好,mac版安装包有了,但是破解方法和破解软件没有。求指导。
需要提供SnapGene登记的信息,组名和登记密码填啥呢
同学,你有破解版的Mac版本了吗?可以分享一下吗?下面链接下载的还需要密码,应该是没有破解成功的。
我也是显示patched but failed to activate 安的是4.1.9,加好友18021308280显示被加频繁。
你好,抱歉 刚刚看见 下载安装everything软件,安装好之后打开,搜索snapgene,删除所有相关文件,确认不是你的实验文件
然后重新安装snapgene
链接:https://pan.baidu.com/s/15avxaFyGHa3dRUPBedjduw?pwd=5z83
提取码:5z83
师兄您好,网盘连接过期了可以重新分享一下吗?谢谢!
师兄,请问snapgene4.2.4 版本crack文件夹中ActivationCrack.exe怎么办?
什么意思 没看懂 没有这个文件?那应该是被360杀毒给删了 去360杀毒的隔离区找找
请问我的snapgene 为什么进去后,自己会突然隐藏序列,我该如何解决这个问题呢?
额 安装的是5.0+版本 会有这个问题,防破解的 建议安装4.1.9 我一直用的这个
那请问4.1.9的包装包还有吗?
网盘里面有的
请问这个问题后来是如何解决的呢?我也是到这一步就激活失败,十分感谢!
额 如果没有猜错 之前有装过其他版本 激活失败过 系统已经记录了 需要完全删除相关文件
可以直接自己下载everything软件 搜索graphpad 删除相关文件 重新安装激活
有问题也可以加我微信
这个怎么解决
用everything这个软件吧之前的所有安装记录删除
学长好,为什么添加下游引物的时候选择复制而不是直接添加底部引物呢?有什么区别吗
你好,其实你可以试一下就知道了 如果选择之后直接add,默认给出的是正义链上5–>3的序列,而非反向的反义链上5–>3的序列
你做PCR需要上下游引物一条在正义链 一条在反义链 往中间扩增目的基因
我是在添加了top Strand后,然后再Add primer,选择的bottom Strand。这时候选择的好像是一样的?我是按着找到的视频来的,不太了解
你这样操作和复制bottom strand 5–>3 是一样的,不管哪种方式 保证上下游引物各在正反义链 往中间扩增即可,都是没有问题的
原来如此,谢谢
请问为什么下载文件包之后没有crack文件呢?
请问是哪个版本,里面应该都有的 推测很有可能被你的杀毒软件清除了,你先检查一下
进哥,你好,我的不知道为什么出现了Patch but failed to activate
你加我微信了 是吗?微信指导吧
你好,我出现了和你一样的问题,就是patched but failed to activate,请问怎么解决的呀?谢谢~
ta给你回复了,但是回复在系统邮件中,因为你下载安装过其他版本,系统中有相关信息,所以软件禁止激活,需要完全删除
您好,安装后运行需要登录账号和密码
那就是没有激活成功 按照教程 解决不了的话可以加我微信指导你安装
你好。我想请问一下,安装的4.1.9版本的,为什么Crack文件夹里面少了 ActivationCrack 这个应用程序啊
你好 可能被杀毒软件删了 这种破解软件经常会被杀毒软件识别为病毒 并删除 可以关闭杀毒软件试试
好的,谢谢!已经解决啦。
师兄,snapgene似乎4.3.8版本就开始有汉化版的了,不知道您这边有没有安装包
我没有下载汉化版的 你搜搜看 找不到的话留言 我来找 其实这个软件还是英文版好用
你好,请问一下我上下游加完了primer后,按了pcr,只有上游出现了我需要的酶切位点,下游一直添加不了。我换了个基因的cds又可以了,请问是什么原因呢。我有两个2000-3000bp左右的cds都不行,换了个300左右的又可以了,cds的大小是否会影响呢
你好,我没有明白你的意思 可以加微信讨论
部分软件解压后显示有Trojan病毒,啥情况
没有关系,这种破解工具要修改相关系统文件,所以杀毒软件会提示病毒
你的网站背景好好玩?
哈哈,关注点应该是内容
王师兄,你好!如何用snap给INDEL ,或者SNP位点设计特异性引物呢
我之前没有接触过SNP或INDEL验证 不过根据你的需求 我整理了一下相关教程,主要策略就是扩增出来测序验证 或者 限制性片段长度多态性(restriction fragment length polymorphism,缩写RFLP)
有问题欢迎讨论
利用SnapGene设计SNP位点的RFLP检测和扩增测序方案
你好,请问我在别的地方看到上游引物设计这个酶切位点后面加上了kozak+ATG启动子,后面才是CDS区引物;下游引物设计酶切位点后加上了终止子+FLAG,后面是引物。这个和您的有什么区别呢?
你好,上游酶切位点后面面添加Kozak序列是为了提高蛋白编码基因真核细胞表达的效率,主要是提高翻译效率。换句话讲,如果做过表达质粒转染后发现mRNA水平有显著提高,而蛋白表达不变或上升不明显,则考虑添加Kozak sequence。你说后面加上ATG启动子,这个我不明白,ATG是起始密码子,是包含在CDS中的,启动子是在你的质粒上酶切位点的上游的。
然后下游引物加FLAG,FLAG是个标签蛋白,你根据自己的实验需求看是否需要,可以加5’,也可以3′
有问题可以再一起交流
安装破解完,打开序列,刚开始显示正常,切换窗口后序列消失,怎么破?
不好意思,好像是因为5.0版本之后都有防破解功能,才导致这个问题,我用的4.1.9版本,没有问题,使用一样。先用5.1.5里的清除工具完全清除之前的安装痕迹再重新安装。
有问题在讨论
请问为什么我点Patch And Register总是显示failed to activate呢,总是破解不了,请问有没有什么解决办法呢
请问有详细一点的报错信息吗?装的4.1.9吗?不应该会有问题
我也是显示patched but failed to activate 安的是4.2.4
要不加我微信 我远程给安装看看,只这样说不清楚
请问解决了吗 我的也一直显示failed to activate
方便的话加我微信 18021308280 远程看看