导语
近年来,长非编码RNA(lncRNA)得到了研究界的广泛关注。如今,人们已经鉴定了大量lncRNA,但大多数lncRNA的功能还未明确。为了解决这一问题,研究者们开始对lncRNA的互作蛋白进行研究,并为此开发出了越来越多的分析工具。 下面我们来介绍一下RIP技术。
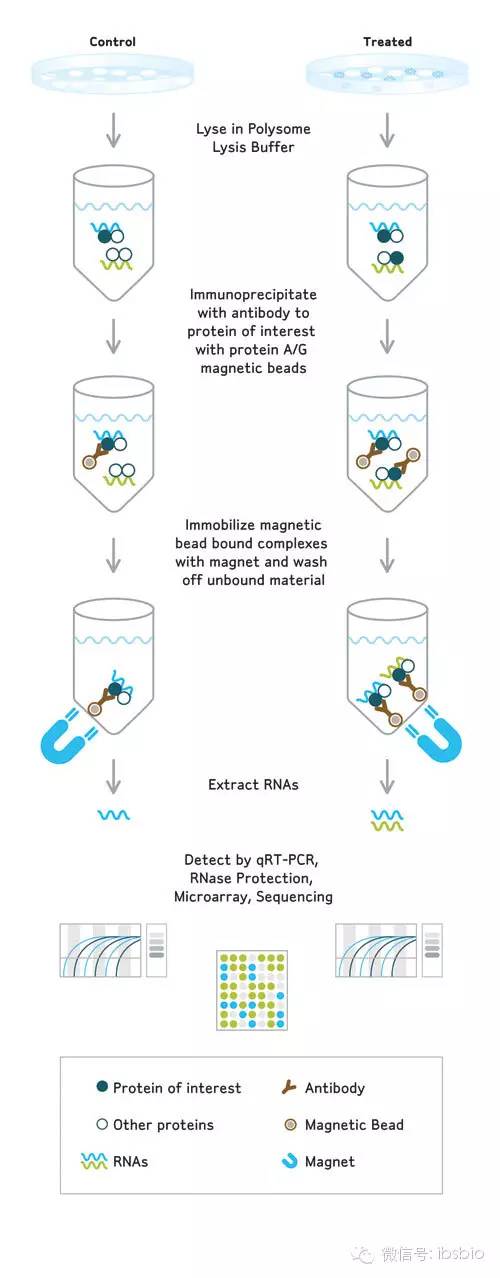
RNA结合蛋白免疫沉淀技术又称为RIP(RNA immunoprecipitation),可以说是RNA版的ChIP(染色质免疫沉淀),该技术能帮助人们分析与RNA结合蛋白相关的核酸。
在RIP试剂盒(如Sigma的Imprint® RNA Immunoprecipitation kit)的帮助下,研究者们能够利用针对RNA结合蛋白的抗体,从细胞提取物中捕获与蛋白相结合的RNA,再通过qPCR、芯片或二代测序技术对这些RNA进行鉴定。
不论是细胞质还是细胞核,都会发生RNA与蛋白的相互作用。据Active Motif公司产品经理Kyle Hondorp介绍,该公司的RNA ChIP-IT® kit专用于研究RNA与细胞核染色质的相互作用。该试剂盒通过甲醛固定来锁定RNA-染色质的互作,随后对染色质进行超声,并用DNAse I处理,最后利用磁珠进行沉淀。
“如你研究的是总mRNA,那么常规RIP试剂盒更合适一些,”Hondorp说,“常规RIP试剂盒可以处理所有细胞裂解物的总mRNA。”
生产Magna RIP™ RNA-binding protein immunoprecipitation kit的EMD Millipore公司,也在开发专门针对染色质的RIP试剂盒。预计这一新产品将于2014年第一季度发布,该产品有化学交联与native两种形式。据介绍,化学交联可以分析究间接的蛋白-RNA相互作用,研究更高分子量的蛋白复合物。而native方法展现的是,亲和力更高也更为直接的相互作用。
除RIP以外,CLIP(UV crosslinking and immunoprecipitation)也可以帮助人们捕捉与RNA结合蛋白互作的核酸。CLIP方案整合了交联与核酸酶消化,不仅允许对RNA-蛋白复合物进行进一步的纯化(如凝胶电泳片段分离),还能够揭示RNA-蛋白互作所发生的位点。最近人们又开发出了新型CLIP 技术——iCLIP(individual-nucleotide resolution CLIP),该技术能够以单个碱基的分辨率,来展示RNA-蛋白互作的详细信息。
据伦敦大学学院的Jernej Ule教授介绍,CLIP与RIP的关键性差异,在于沉淀复合物后的凝胶纯化步骤。CLIP技术可以给RNA-蛋白复合物的RNA末端带上放射性标记,并将这些复合物进行SDS-PAGE分离,之后转移到一张膜上。这样的过程可以提高纯化的特异性,减少非特异性的RNA。蛋白酶消化可以将RNA从膜上解离下来,用于制备cDNA文库,以备测序分析。
“CLIP要比RIP麻烦一些,不过我认为它很值得采用,”Ule说。“放射性信号的量及其特异性,能够帮助我们提高cDNA文库的质量,最终得到准确实用的数据。”
RIP实验流程
一. 细胞裂解液获取
A. 单层细胞或者贴壁细胞处理
1. 冷PBS清洗培养皿或培养瓶中的细胞两次
2. 加入冷PBS后用细胞刮将细胞刮下来,收集至enpendoff管
3. 1500rpm,4℃离心5min,弃上清,收集细胞
4. 用与细胞等体积的RIP裂解液重悬细胞,吹打均匀后于冰上静置5min
5. 每管分装200μl细胞裂解液,贮存于-80℃
B. 悬浮细胞处理:先收集细胞再计数,然后清洗裂解
C. 组织样品处理
1.冷PBS清洗新鲜切下的组织三次
2. 加入冷PBS后,用匀浆器或其他细胞分离设备使组织分散为单个细胞,计数
3. 1500rpm,4℃离心5min,弃上清,收集细胞
4. 用与细胞等体积的RIP裂解液重悬细胞,吹打均匀后置于冰上静置5min
5. 每管分装200μl细胞裂解液,贮存于-80℃
二. 磁珠的准备
A. 实验前准备
1、enpendoff管
2、磁力架
3、冰盒, RIP Wash Buffer置于冰上
4、抗体,置于冰上
5、涡旋震荡器6、枪、枪头放于超净台照射30min,枪喷DEPC水
B. 磁珠准备过程
1. 重悬磁珠
2. 标记实验所需的enpendoff管,样品包括目的样品,阴性对照与阳性对照
3. 吸取50μl 重悬后的磁珠悬液于每个enpendoff管
4. 每管加入500μl RIP Wash Buffer,涡旋震荡
5. 将enpendoff管置于磁力架上,并左右转动15°使磁珠吸附成一条直线,去上清,重复一次
6. 用100μl的RIP Wash Buffer重悬磁珠,加入约5ug相应抗体于每个样品中
7. 室温孵育30min
8. 将enpendoff管置于磁力架上,弃上清
9. 加入500μl RIP Wash Buffer,涡旋震荡后弃上清,重复一次
10. 加入500μl RIP Wash Buffer,涡旋震荡后置于冰上
三. RNA结合蛋白免疫沉淀
A. 准备工作
1、冰盒
2、360°旋转仪
3、RIP Wash Buffer 、0.5M EDTA 、RNase Inhibitor 置于冰上
B. RNA结合蛋白免疫沉淀实验过程
1. 准备RIP Immunoprecipitation Buffer
2. 将前上步的enpendoff管放磁力架上,去上清,每管加入900ul RIP Immunoprecipitation Buffer
3. 迅速解冻第一步制备的细胞裂解液,14,000rpm,4℃离心10min。吸取100μl上清液于上一步的磁珠-抗体复合物中,使得总体积为1ml
4. 4℃孵育3h至过夜
5. 短暂离心,将enpendoff管放在磁力架上,弃上清
6. 加入500μl RIP Wash Buffer,涡旋震荡后将enpendoff管放在磁力架上,弃上清,重复清洗6次
四、RNA纯化
A. 实验前准备
1. 枪、枪头、enpendoff管紫外照射30min,喷DEPC水以除RNA酶
2. Salt SolutionⅠ、Salt SolutionⅡ、RIP Wash Buffer、Proteinase K、10%SDS、 Precipitate Enhancer 置于冰上
3.RNase-free的乙醇、氯仿、异戊醇
4.DEPC水置于冰上
5. 离心机预冷
6. 冰盒
B. RNA纯化过程
1.准备Proteinase K Buffer。每个样品需150ul
2.用150ul Proteinase K Buffer重悬上述磁珠-抗体复合物
3. 55℃孵育30min
4. 孵育完之后,将enpendoff管置于磁力架上,将上清液吸入一新的enpendoff管中
5. 于每管上清液中加入250μlRIP Wash Buffer
6. 于每管加入400μl苯酚:氯仿:异戊醇,涡旋震荡15s,室温下14,000rpm离心10min
7. 小心的吸取350μl上层水相,吸入另一新的enpendoff管
8. 于每管加入400μl氯仿,涡旋震荡15s,室温下14,000rpm离心10min
9. 小心的吸取300μl上层水相,吸入另一新的enpendoff管
10. 每管加入50μl Salt SolutionⅠ,15μl Salt SolutionⅡ,5μl Precipitate Enhancer ,850μl 无水乙醇(无RNase),混合,-80℃保持1h至过夜
11. 14,000rpm,4℃离心30min,小心去上清
12. 用80%乙醇冲洗一次,14,000rpm,4℃离心15min,小心去上清,空气中晾干.
13. 10-20μl DEPC水溶解,-80℃保存,送测序
RIP实验常见问题
1:RIP试验需要注意什么?
(1)防止RNA与蛋白非特异性结合。
(2)避免RNA蛋白质结合被破坏。
(3)避免外源RNAse污染。
(4)抑制内源RNase的活性。
(5)选择适合做RIP的抗体。
(6)避免RNA结合蛋白的降解。
2:为什么抗体无法沉淀RIP裂解液中的目标蛋白?
(1)先进行IP或者WB,测试抗体可以与目标RBP结合。
(2)另选一个能与抗原其他表位结合的抗体。
(3)尽可能选用密理博RIPAb+抗体。
(4)稀释抗体成浓度梯度,进行IP测试。
(5)4℃过夜孵育抗体与RIP裂解液。
(6)确定抗体类型与Protein A/Protein G匹配。
3:提取RNA时,蛋白酶k消化不完全是什么原因?
当进行蛋白酶K消化时,确保水浴温度应设置在55℃左右,长期在65℃以上孵育会导致蛋白酶K失活。
4:提取RNA后,A260/A280比值偏离1.8~2.2区域是什么原因?
(1)需要缩短酚氯仿提取RNA的时间间隔。
(2)测试提纯后RNA中是否存在RNA酶,可能RNA发生了降解。
5:做RIP的时候和beads孵育的lysate的浓度如何确定?有些动物的文献提到用细胞板数细胞个数,想知道如果用蛋白浓度的话,大概在什么数量范围的蛋白总量对应50 ul BEADS?
50 ul beads对应的是一个reaction,而我们推荐一个reaction是100ul裂解上清(2×107 cells加入100ul RIP lysis buffer得到),所以只需要在裂解前计数一下,并按括号中的比例加入裂解buffer,后续100 ul 裂解上清就是一个reaction的蛋白用量。不建议通过裂解后测蛋白浓度的方法进行配比。
6:RIP可以分提核浆的RNA-蛋白质复合物吗?
理论上,可以采用能分别抽提核浆蛋白的蛋白抽提kit先分离样本,再进行RIP实验。但需要特别注意的是所用kit中的reagent不能有RNase和DNase,以及要能与下游的抗体beads孵育兼容。
7:因为RIP做完得到的是RNA序列,要做后续检测,比如测序、判断目标序列位置等,是不是都必须要做RT-PCR,得到逆转录的DNA后,后面就跟ChIP相同了?
常见的用real time qRT-PCR。如果对照的目的是保证两个样品间加入的细胞量一致或者进行定量,理论上说,使用input作为判别,计算不同样品上样量的差异。如果对照的目的是为了看IgG以及目的抗体富集的非特异性mRNA的量的话,那么可以找一个确定不会与目的抗体结合的RNA片段设计primer进行qPCR,用来看IgG以及目的抗体拉下来的背景情况。RIP可以看成是普遍使用的染色质免疫沉淀ChIP技术的类似应用,但由于研究对象是RNA-蛋白复合物而不是DNA-蛋白复合物,RIP实验的优化条件与ChIP实验不太相同(如复合物不需要固定,RIP反应体系中的试剂和抗体绝对不能含有RNA酶,抗体需经RIP实验验证等等)
rip-seq后续结果怎么处理啊,我从geo上扒到了一个数据集,但是不会分析,俊哥能给指条路吗
想请问一下文中提到的这些buffer的配方。谢谢老师
你好请问我看RIP实验步骤,有的是先将磁珠跟目的抗体或igg结合,再加入到细胞裂解液中形成磁珠-抗原-抗体复合物,再用蛋白酶K洗脱提纯RNA, 但是有的protocol是先将抗体加入到细胞裂解液中,再加入磁珠孵育。我看您写的就是前面的顺序,这个有讲究么?另外RIP的组别应该怎么设置?如果只是想验证目的蛋白与某个RNA结合,是否只需要一个正常处理的细胞样本(10cm皿细胞量)就可以做?分为input
、IP、IGG组?期待回复,非常感谢!
你好,顺序问题其实都可以,两步的话是经典步骤,一步法就是省时间,从准确性和经济性上我觉得还是两部分开更合适
您好,想请问一下国内哪个公司做RIP-seq做的比较好啊
您好 这个倒不清楚,也不能乱说 我只找过一家做,感觉结果不咋地,如果有条件,自己做RIP 然后送测序
我已经自己沉下来了RNA,想送去测序,问了几家公司,比较纠结,能不能冒昧问一下您是在哪家公司做的,可以规避一下
你好,请问IP和input样本最终是等质量逆转录然后投入等质量的cDNA跑RT-PCR吗?qPCR时需要跑内参基因吗?是否可以详细解释一下qPCR部分的数据处理过程?
m6A-IP(MeRIP)-qPCR计算相对表达量
你好,可以参考merip进行数据分析
RNA需要等质量,如果不够可以根据比例换算也可以
请问RIP-qPCR引物就是普通的qPCR引物吗?还有IgG是要加和目的抗体同样的量是吧?但是qPCR检测IgG也拉出很大量的某基因正常吗?
RIP引物建议产物长度短一点 80-120bp吧,太长可能会存在假阴性;
IgG加同样量;
IgG按道理不应该会有大量某基因,检查一下操作步骤吧
您好,请问,RIP实验,RT-PCR, IgG目的跑不出来,如何计算%input呢
您好,Input% = 10% x 2(C[T]10%Input Sample–C[T]IP Sample),IP sample包括目的蛋白抗体和IgG抗体,分别计算Input%
如果igG确实跑不出,说明你的背景很干净呗,讲道理多多少少会有残留,如果没有就按照Input%=0 或者就不放