有同学留言简并引物的设计方法,抱歉,我没有做过这个,所以网上找了一下,这一个方法看起来比较详细,希望可以有帮助。关键是整个设计思路,所用软件工具不一定要一样。
原文转自:raindy的网络硬盘
设计一对合适的引物是 PCR 扩增目的基因成功的关键,但许多人往往过度依赖 Primer Premier(以下简称 PP)或 Oligo 等引物设计软件,有时候引物没设计成,却身陷软件之中无法自拔。其实,不论特异性引物或简并引物,只要掌握了几个关键点,手动也可以设计出一对好引物。如果不是大批量设计引物或设计复杂的引物序列,下面的四个常用工具即可轻松胜任引物设计任务。下文以马铃薯 Y 病毒 CP 基因简并引物设计为示例,分享一些个人经验,希望对初学者能起个抛砖引玉作用。受专业领域及水平所限,文中有不当之处,敬请各位同仁、童鞋批评指正。
【相关工具】
- MEGA5-多重序列比对、选取基因区域、序列编辑
- DNAMAN8-检测两引物的互补性
- Oligo Calc-评估引物的属性
- Web Logo 3-直观显示简并碱基
————————————————-【华丽分割线】————————————————-
【基本原则】
设计一对好的引物,归结起来就是 5′端引物、3′端引物之间以及两者与模板的关系处理
得恰到好处:
(1)两引物的序列要与模板的序列紧密互补;
(2)两引物不能在模板的非目的位点发生错配;
(3)两引物之间尽量减少二聚体或发夹结构生成。
【延伸原则】
(1)引物长度:常用为 18-27bp,最大不可超过 38bp,否则容易导致延伸温度过高,
不适合 DNA 聚合酶反应;
(2)GC 含量:一般介于 40%-60%之间,且两个引物之间的 GC 含量相差不能过于悬
殊;
(3)碱基分布:随机分布最佳,但避免连续的 GC,GC 富集区容易导致错误引发反应。
【特别注意】
5′端引物的作用主要限定 PCR 产物的长度,对扩增特异性影响不大;引物的延伸是从 3′
端开始的,所以 3′端引物是影响特异性扩增的最关键因素,因此,在实际设计过程中,设计
3′端引物时,需要综合考虑以下几个内容:
(1)不要终止于密码子的第 3 位
(2)末位碱基避免使用碱基 A
(3)避免出现 3 个以上连续的 G 或 C,如 GCG 或 CCC 或 GGG
(4)ΔG 的绝对值不可超过 9
(5)与非特异扩增的序列同源性不能超过 70%或有连续 8 个互补碱基同源
(6)不能进行任何修饰
—————————————【华丽分割线】————————————————-
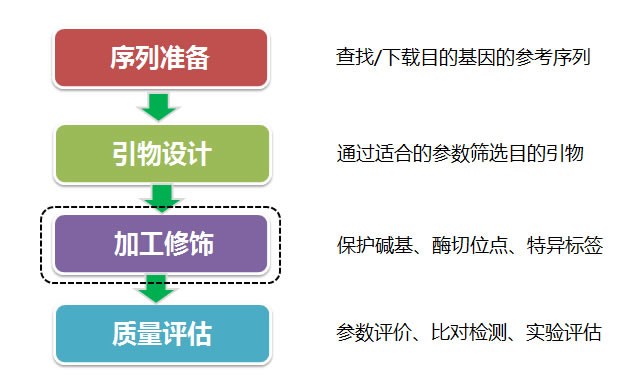
【设计流程】
【示例背景】
马铃薯 Y 病毒(Potato virus Y, PVY)是侵染马铃薯、烟草、辣椒等茄科作物并造成严
组株系产生,单一的特异性引物无法适应 PVY 不同株系的检测需求,需要设计一对简并引物以能够满足生产上的检测需求。
重危害的病毒之一,广泛分布全球各马铃薯种植区。RT-PCR 技术具有高度的特异性和灵敏性等特点,已经成为 PVY 检测最常见的方法。但由于 PVY 株系分化严重,不断有新的重
【详细图解】
1.序列准备:
(1)GenBank 下载 PVY 全基因组序列
(2)由于基因序列比较大,且数量多,推荐用 MAFFT 多重序列比对
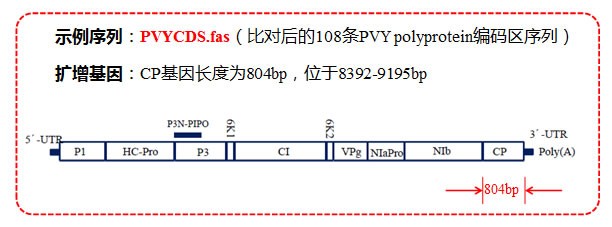
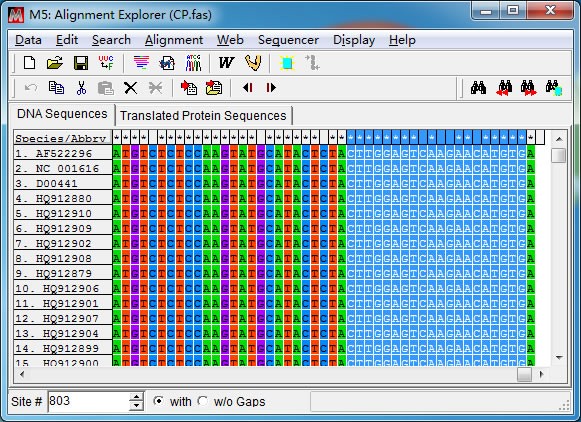
(3)扩增片段区域选择,CP 基因长度及位置如下图所示:
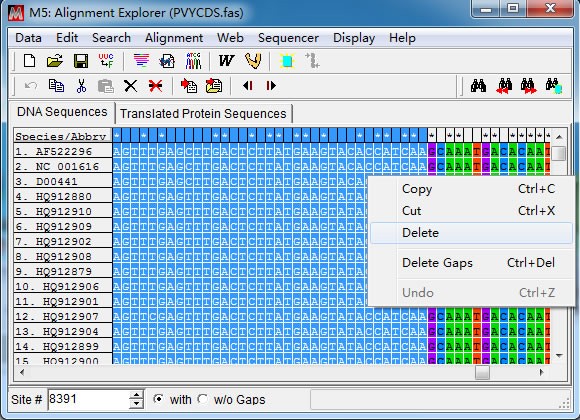
先将光标定位在第一条序列任意位置,然后在左下角”Site”处直接输入 CP 基因上游分
界点位置(8391)后回车。接着点击“Speicaes/Abbrv”和 8391 那一列的交界点时按下键盘上
“Shift”不放,移动光标到“1”那一列,此时点击鼠标右键“Delete”删除冗余序列。
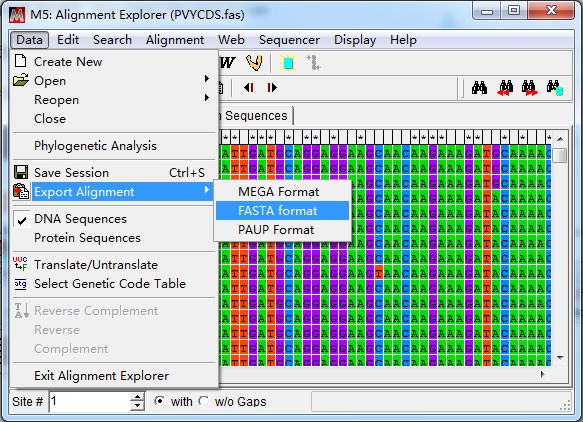
裁切后得到CP 基因编码区序列,“Export Alignment”导出 CP 基因序列(推荐 fasta 格式)。
2. 引物设计
推荐先设计 3′端引物,至于原因,上面的【特别注意】中已提到,这里不再赘述。
(1)选择引物序列
综合考虑引物设计原则及特别注意,在 CP 基因 3’端位置选取一段合适的序列
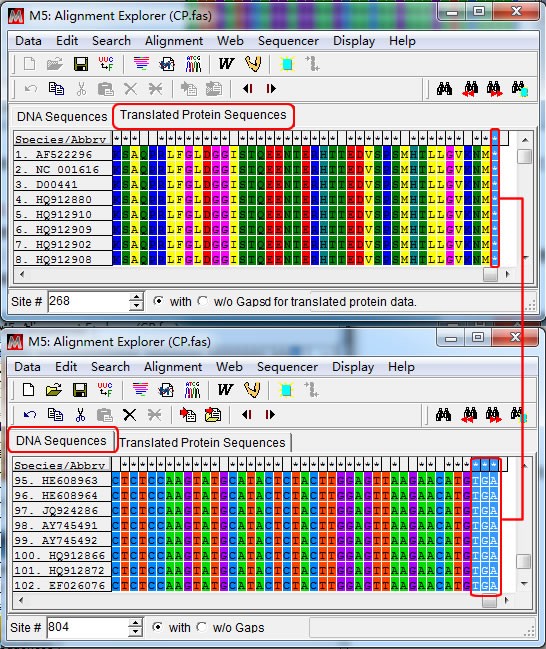
(784-803bp),804bp 位置为 A 且是第三个密码子位置,所以弃去末位的 A 碱基。
PS:是否位于密码子的第 3 位,可以通过“Tranlated Protein Sequences”-“DNA sequences”
切换查看,如下图,CP 基因的末端 3 个碱基是终止密码子所在的位置,A 是第三位密码子。
(2)生成 Seq Logo
选定序列后,参考上述步骤,删除冗余序列,保留 CP 基因 3’端序列,同样导出为 Fasta 文件,将序列粘贴入 Web Logo3(http://weblogo.threeplusone.com/create.cgi)的文本框内或直接上传序列文件,设置相关参数后点击“Create”生成 Seq Logo 格式的文件 CP-3.fas。
参数设置: “Output format”(输出格式)推荐用“PDF”,“Color scheme”(颜色方案)
推荐用“Classic (NA)”,设置完毕,点击右下角“Create Logo“即可得下图的 Seq Logo 格式文件。
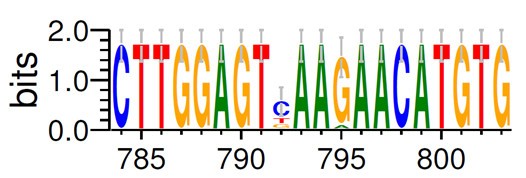
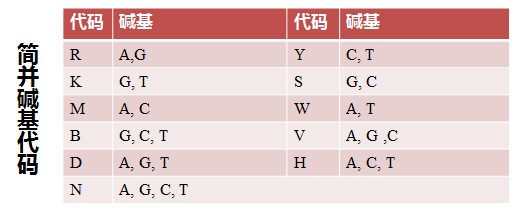
通过 Web Logo 生成的多重比对图片可以很直观得到 3’端序列为CTTGGAGT(C/T/G)AA(G/A)AACATGTG,查询简并碱基代码表(下图),可知 C/T/G=B,
G/A=R,简并度=3 ×2 =6,整理后 3′端序列为 CTTGGAGTBAARAACATGTG,故而需要合成的 3′端引物序列: CACATGTTYTTVACTCCAAG (与原序列是反向互补关系)。
3. 质量评估
(1)参数评估
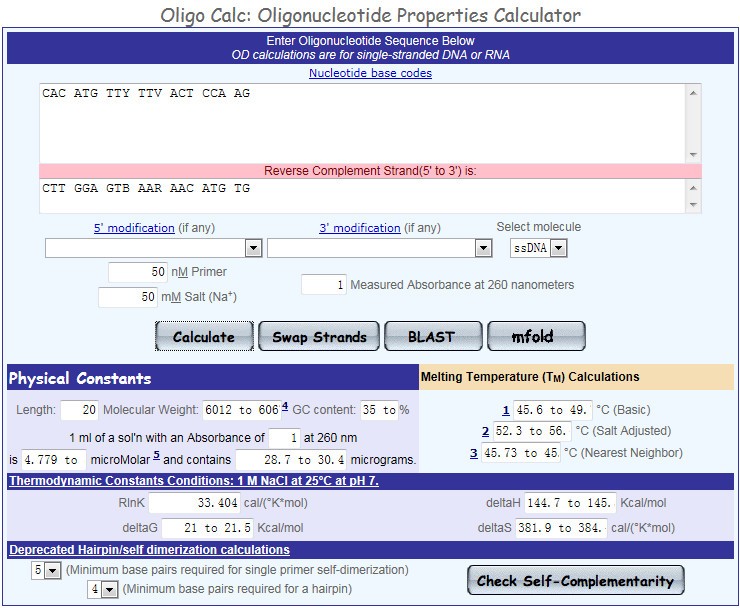
将初步得到的引物序列,粘贴入 Oligo Calc 的文本框内,按下“Calculate”按钮,得到引物的相关参数,如:长度、GC 含量、Tm 值等信息。
(2)自身互补性(Check Self-Complementarity)
点击“Check Self-Complementarity”,检测引物自身互补性,主要查看“Potential hairpin formation”、“3′ Complementarity”、“All potential self-annealing sites are marked in red (allowing
1 mis-match)”下方显示内容,如果三项均为“none“,则说明引物自身不会形成发夹结构且引物自身不会互补,引物没问题!
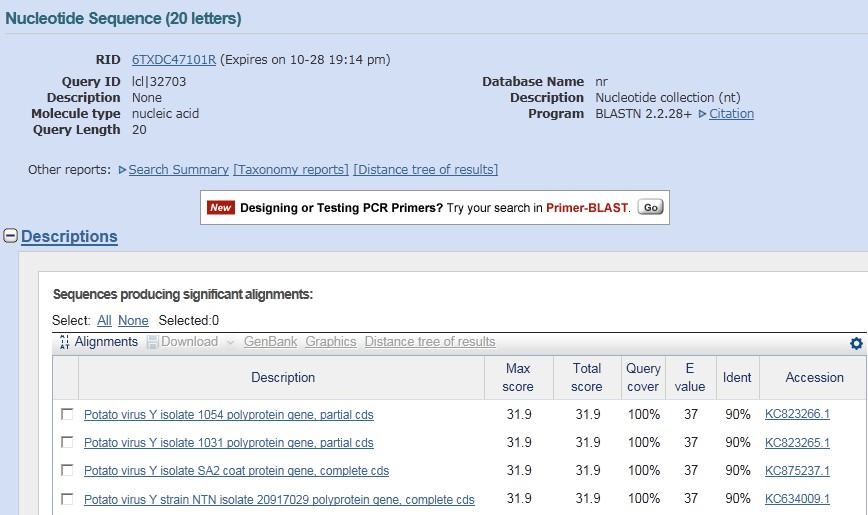
(3)BLAST 比对回检
点击“Blast”可以对引物进行 BLAST 比对检测,结果显示,都是 PVY CP 基因相关的序列,表明所设计的引物特异性良好。
(4)两引物互补性检查
同样方式,得到 5’端序列:GSAAAYGAYACAATYGATGC,根据引物设计原则,需要检测两引物之间是否存在序列互补。
打开 DNAMAN ,依次在“Primer”-“Load primer”,粘贴任意一端引物序列,如:5’端序
列-GSAAAYGAYACAATYGATGC,确定。
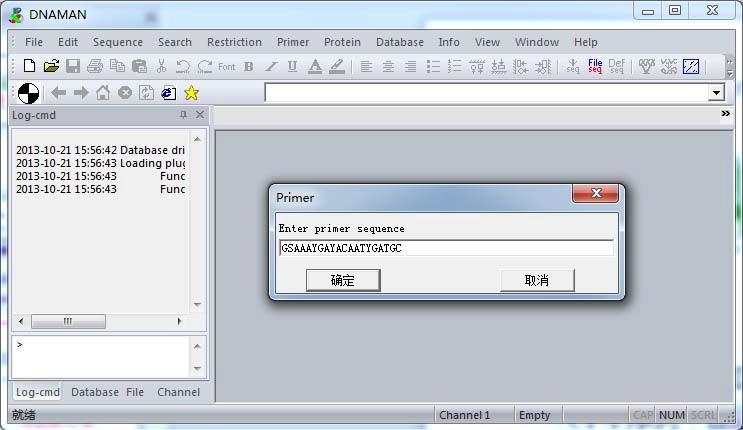
接着,在“Primer”-“Two primers Complementarity”-“Second Primer From Input”,粘贴入另一端序列,这里为 3’端引物序列:CACATGTTYTTVACTCCAAG,如下图:
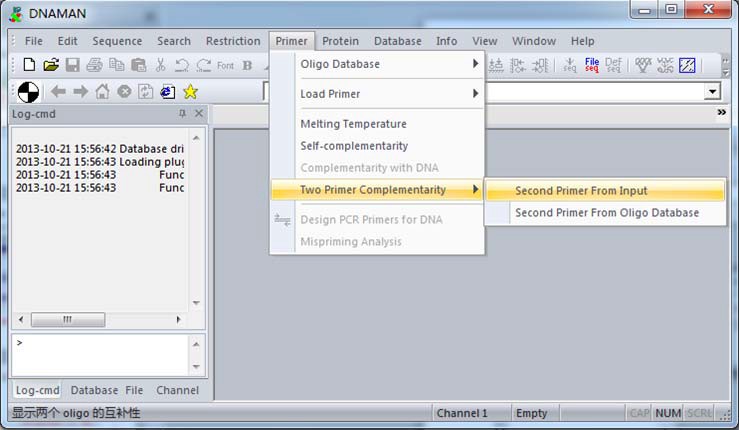
结果显示,两引物间仅有 3 个碱基互补,且自由能仅为 1.8 Kcal/mol。

(5)实验验证
PCR 扩增采用 50μL 反应体系为:10 ×TransTaq HiFi Buffer II 5 μL,2.5nM dNTPs 4 μL,
5′端引物(10 μmol/L)2 μL,3′端引物(10 μmol/L)2 μL,ddH2O 34.75 μL,TransTaq HiFi Polymerase(5 U/μL)0.25 μL,cDNA 2 μL。
PCR 扩增程序条件为:94℃预变性 5min,94℃变性 30s,55℃复性 30s,72℃延伸 1min, 共 30 个循环,最后一轮循环后 72℃延伸 10min。反应结束后,1%琼脂糖凝胶电泳检测 PCR 扩增产物,如下图所示:
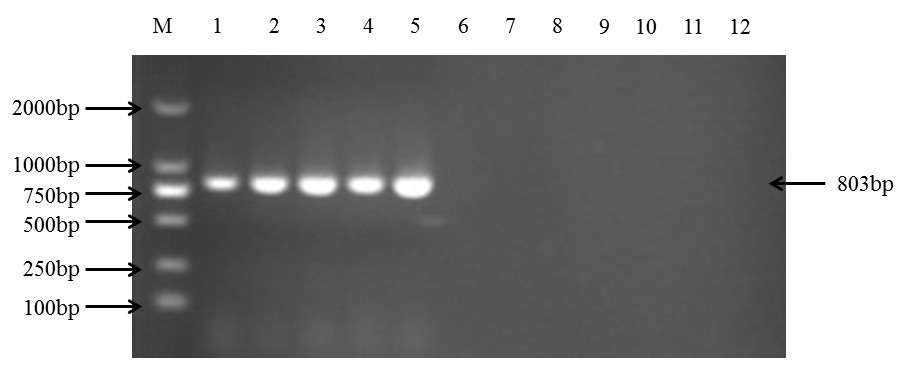
M.DNA 分子量标准(100bp);泳道 1-5:PVY 的不同株系(C、O、N、NTN、N-Wi);泳道 6-7: 阴性对照(健康马铃薯)和空白对照;泳道 8-12:PVX、PVM、PVA、PVS、PLRV
- 吴祖建, 高芳銮, 沈建国. 生物信息学分析实践. 科学出版社, 2010: 30-60.
- 高芳銮, 沈建国, 史凤阳, 方治国, 谢联辉, 詹家绥. 我国马铃薯 Y 病毒的检测及 CP 基因的分子变异. 中国农业科学, 2013, 46: 3125-3133.
- 史凤阳, 高芳銮, 常飞, 詹家绥. 马铃薯 Y 病毒简并引物的开发与检测应用. 2013 年中国马铃薯大会会议论文, 2013, pp: 289
求问没有序列该怎么设计引物呢,尝试了好几次都扩不出来
引物设计还是小白,但是进哥哥讲的已经很详细了。要多向您学习学习。
十分感谢王进老师您的知识分享,学习到了,也成功应用到了我的实验中。
并且在其他实验中浏览到您的网站的一些教程,受益良多!
不客气,能帮助到你就好