1. 简介
ArrayExpress是高通量功能基因组数据的一个公共数据库。ArrayExpress由两部分组成 ——ArrayExpress Repository,它是一个MIAME(Minimum Information About a Microarray Experiment)支持的微阵列数据的公共档案库,及ArrayExpress Data Warehouse,它是选自档案库的基因表达谱及一致性重注释的数据库。归档的实验能够通过实验属性,例如关键词,物种,阵列平台,作者,期刊或访问号进行查询。基因表达谱能够通过基因名字和属性,例如基因本体论术语进行查询,并且基因表达谱能够被可视化。ArrayExpress是一个快速增长的数据库,目前它包含的数据来自超过5万次的杂交和超过1500000次的独特表达谱。
ArrayExpress与Geo数据库类似,里面都存储了大量的芯片表达数据,对于数据库挖掘的学员们来说,ArrayExpress是一个不可或缺的数据库。比如如果你在在GEO数据库中搜索不到想要的结果时,可以在ArrayExpress数据库中搜索,它与GEO数据库互补,帮助大家完成数据的搜索和下载,方便后续的分析。今天我们先讲一下怎么在网页上下载想要的数据,后续会讲怎么用R实现数据的下载及数据的处理和分析。先放一个ArrayExpres数据库的主页链接(https://www.ebi.ac.uk/arrayexpress/)
2.数据下载
主页如下:
进到主页之后,我们可以先看一下这个数据库的信息,比如它是功能基因组数据,目前有72213个实验的数据,2374791个芯片数据,54.54TB的存档数据,这都是在描述ArrayExpres数据库的数据量的大小。了解完之后,我们可以在Search栏进行数据的搜索,举个栗子:我想要搜一个关于肺癌癌旁的研究,那我就输入关键字,然后点击搜索。
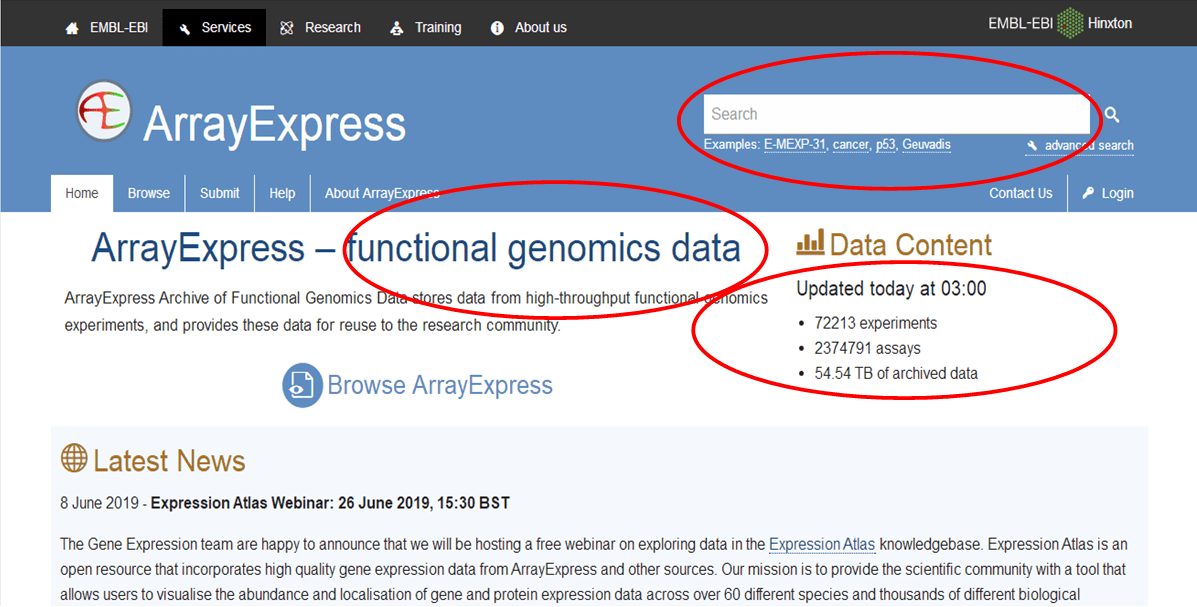
搜索完之后,我们看一下结果,有82个实验的研究出来了,其中有一些可能不是你想要的,怎么办呢,咱们可以加一些过滤条件,限定一下,以减少实验研究的数量。”Filter search results”这个就是添加过滤条件的地方,我现在加一些条件,比如我把研究的物种选成”物种是人(Homo sapiens)”,实验类型我选RNA Assay表达芯片,下面选Array assay,最后一个默认为全部芯片数据就好,然后点击filter即可过滤。
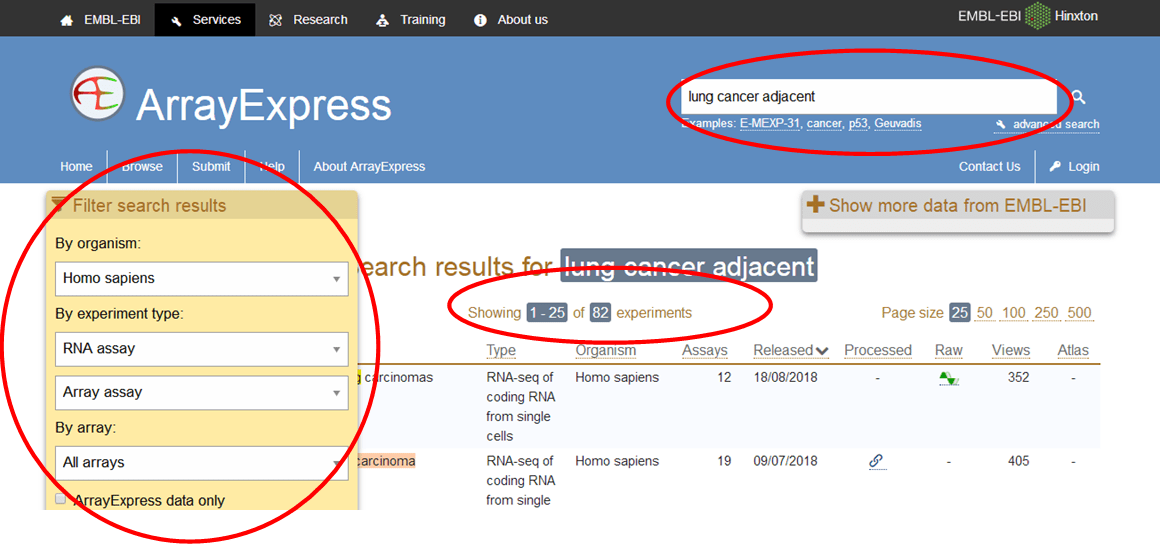
设置完过滤条件之后,点击搜索就会出现结果,看一下上一步过滤出来的结果,现在实验研究之用56个了,减少了。
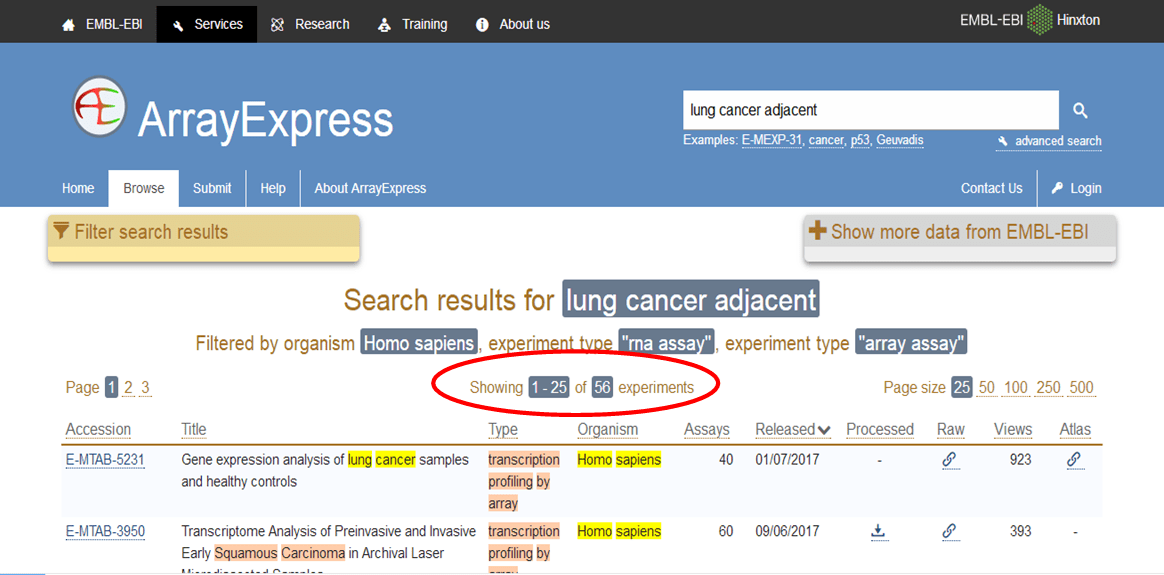
我把出来的这个结果对应的每个字段解释一下:(以E-MTAB-3950为例)
1. Accession:数据库ID,芯片的数据库编号;
2. Title:题目也就是文章的研究方向;
3. Type:实验类型,转录组分析;
4. Organism:物种,研究的物种是人;
5. Assays:芯片样本数,有60个样品;
6. Released:上传日期,2017年6月9号;
7. Processed:处理过的数据(包括:矩阵数据和单样品数据)。如果这里有下载的箭头,可以下载到它们。
8. Raw:原始数据链接,打开后可以下载数据。
第七步和第八步是可以下载数据的啊。下面接着讲的页面也是可以下载数据的。
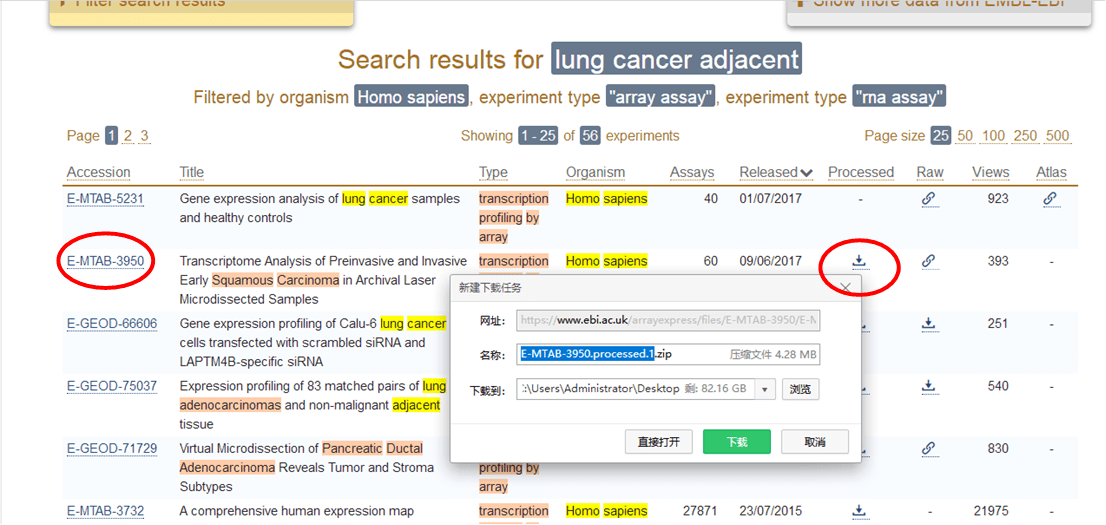
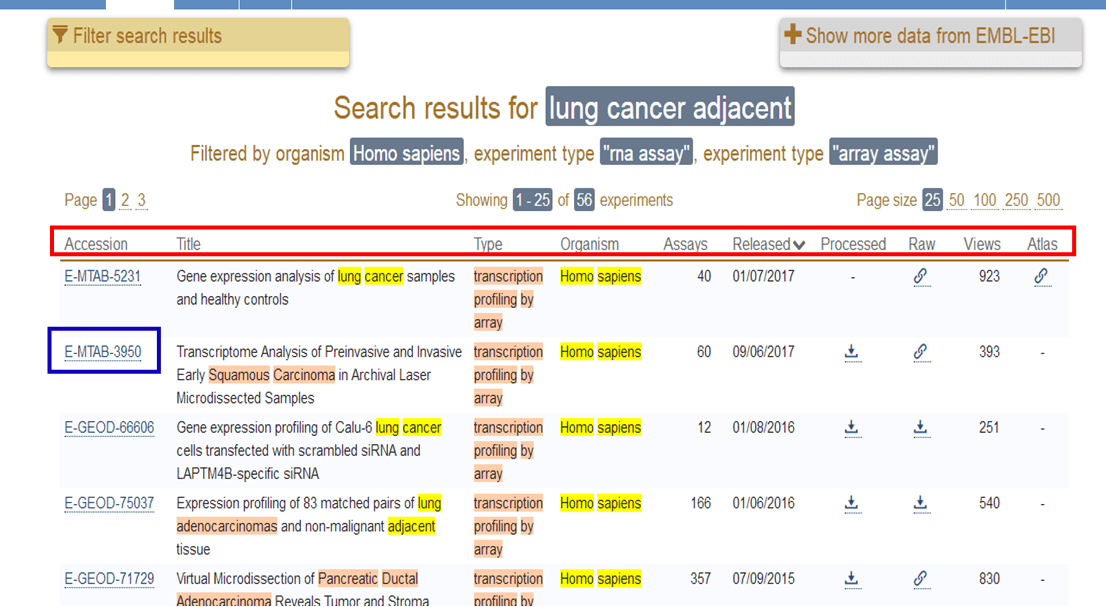
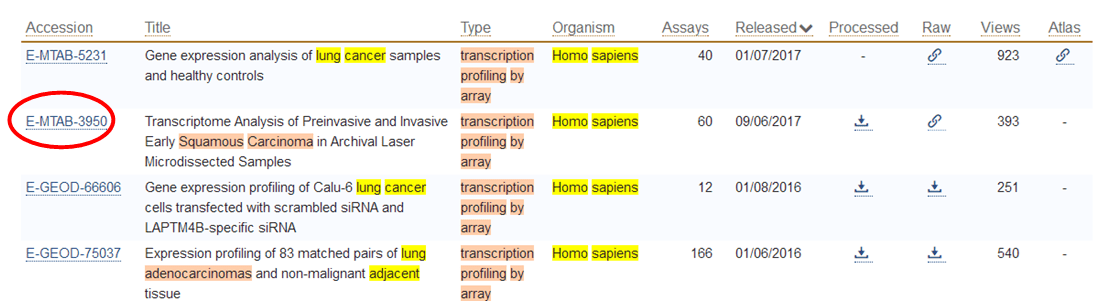
你还可以在下面的页面中点击E-MTAB-3950号,进入详细的页面进行查看。
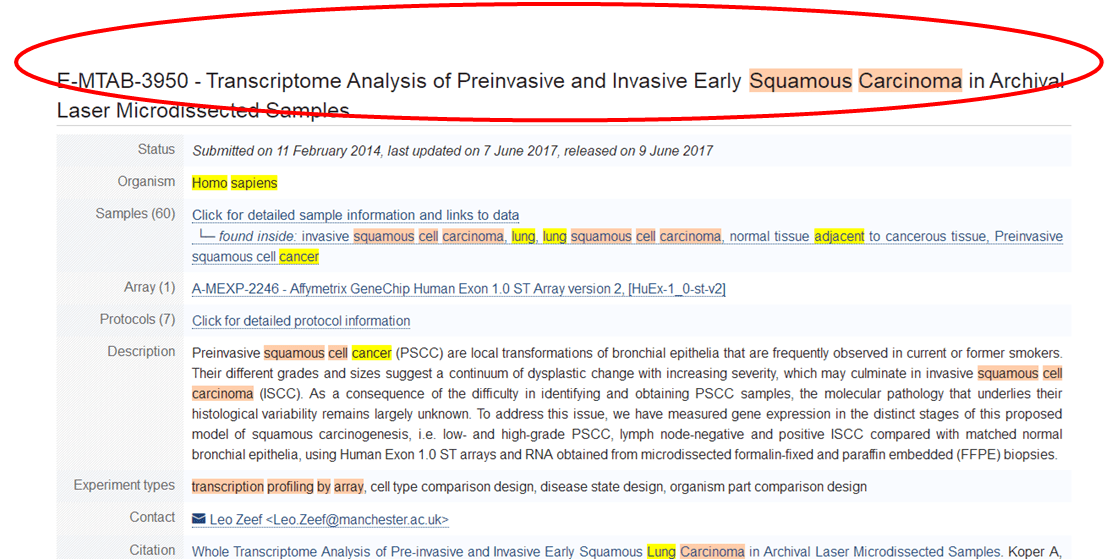
进去之后信息包括:
题目:E-MTAB-3950 – Transcriptome Analysis of Preinvasive and Invasive Early Squamous Carcinoma in Archival Laser Microdissected Samples
1. Status(状态):包括提交日期、更新日期和发布日期
2. Organism(物种):人物种
3. Samples(样本数):60个样品
4. Array(数组):
5. Protocols (协议):样品采集协议、阵列扫描和特征提取协议、归一化数据转换协议、核酸标记协议等。
6. Description(描述):浸润性鳞状细胞癌(PSCC)是支气管上皮的局部转化,在当前或以前的吸烟者中经常观察到。
7. Experiment types(实验类型):通过阵列转录分析,细胞类型比较设计,疾病状态设计,生物体部分比较设计
8. Contact(联系方式):作者邮箱信息
9. Citation(引用):这篇文章的引用方式
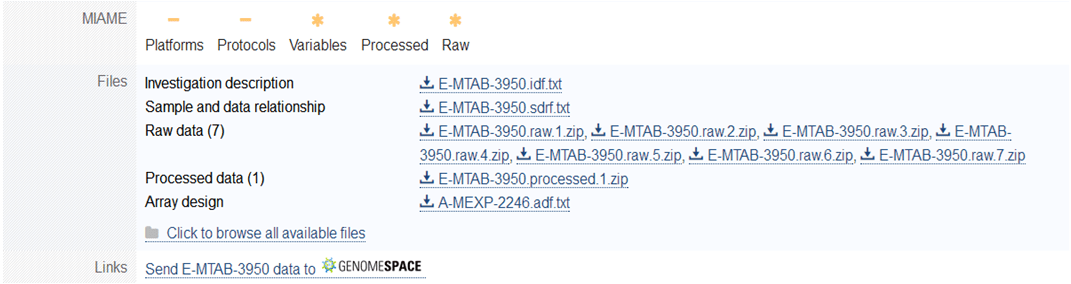
10. Files(文件):包括调查描述、样本和数据关系、原始数据、处理过的数据和阵列设计,在这里可以下载到你想要的数据。
请问下载的原始数据的第一列表示什么呀