我们一般做测序的时候,如果没有明确要求进行mRNA富集,一般给出的测序数据是包含编码和非编码RNA的。对于不同课题组经常会有不同的研究倾向,需要从数据中将所有非编码RNA提取或剔除出去。这个其实很简单,可以自己下载GFT文件,用Excel VLOOKUP函数进行比对,然后复制出来。咱们今天讲一下如何用R语言实现。
开始之前,大家先了解一下什么是Ensembl ID、Entrez ID和Gene symbol?
Gene ID 也称Entrez ID/EntrezGene ID ,是 NCBI 使用的能够对众多数据库进行联合搜索的搜索引擎, 其对不同的 Gene 进行了编号, 每个 gene 的编号就是 entrez gene id,就是一串数字,比如:TP53 的Gene ID是:7157。因为entrez ID相对稳定, 所以也被其他数据库, 如 KEGG 等采用。不同物种的同一个基因的Gene ID是不同的。NCBI的RefSeq数据库ID,一般是两个大写首字母,加下划线,后面为数字。两个首字母 ”NC”、”NM”、”NP_”分别代表DNA、mRNA、Protein。
Gene Symbol ,是HGNC数据库为基因提供的官方名称,HGNC是人类基因命名委员会(HUGO Gene Nomenclature Committee);人类基因组命名委员会。有专门的数据库:https://www.genenames.org/。主要是按基因的功能起的名字,字母一般为英文全称的缩写,由大写字母和数字组成,如TP53基因的Official Symbol就是由HGNC所提供。需要注意的是,因为基因命名的变化,可能一个基因有多个别名,甚至一些别名与其他基因名字一样,因此使用基因Symbol一定要注意,不建议一开始分析就用symbol。
Ensembl ID,是在Ensembl数据库中对基因的命名,常见的物种前缀:“ENS“表示Homo sapiens (Human),”ENSMUS“表示Mus musculus (Mouse),”ENSDAR“表示Danio rerio (Zebrafish);而常见的序列类型用G、P、T、分别表示gene、protein和transcript。这个和Entrez ID一样比较稳定,甚至优于Entrez,在版本更新之后会在相应ID后面添加一个版本号.**。TCGA下载的数据一般就是用的Ensembl ID。
接下来开始使用BioMart包提取基因注释信息:
# Load the required packages
suppressPackageStartupMessages({
library(biomaRt)
library(tidyverse)
})
# Use biomaRt functions to create the conversion table
biomart_table <- getBM(
attributes = c(
"external_gene_name",
"ensembl_gene_id",
"entrezgene_id",
"gene_biotype"
),
mart = useMart("ensembl", dataset = "hsapiens_gene_ensembl")
)
#########对于不同物种,请修改dataset,可以使用如下代码查看:
##mart = useMart('ensembl')
##listDatasets(mart)
##比如小鼠:mmusculus_gene_ensembl,斑马鱼:drerio_gene_ensembl
# Replace empty values with NA
biomart_table[biomart_table == ""] <- NA
biomart_table <- biomart_table %>%
dplyr::select(
"Symbol" = external_gene_name,
"Ensembl" = ensembl_gene_id,
"Entrez" = entrezgene_id,
"Biotype" = gene_biotype
) %>%
arrange(Symbol, Ensembl, Entrez, Biotype)
# Save the output for use in the app
saveRDS(biomart_table, "app_data/biomart_table_homo.Rds")
至此,就可以得到注释文件,接下来的任务就是匹配,可以使用Excel,方便的话还是用R:
biomart_table <- readRDS("app_data/biomart_table_homo.Rds")
results <- read.delim("diffexpr-results.txt") #数据第一列为Ensembl ID
results2 <- merge(results,biomart_table[c(2,1,4)],by = "Ensembl")
results2 <- merge(biomart_table[c(2,1,4)],by = "Ensembl",results)
write.csv(results2,"results.csv")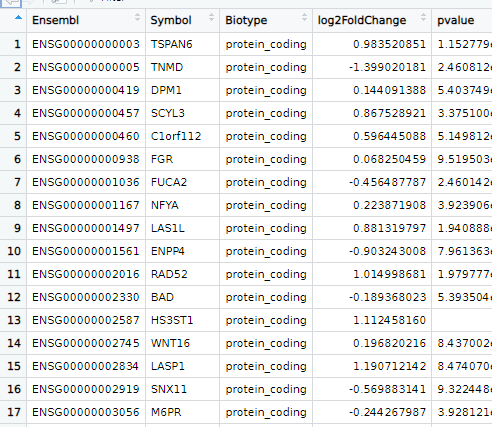
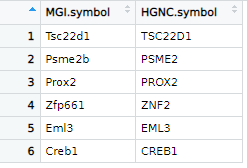
此外,还可以根据这个包进行人鼠同源基因名字转化,使用getLDS函数,它是biomaRt查询的主要功能,连接两个数据集,并从这些链接的biomaRt数据集检索信息。在Ensembl中,这转化为同源映射。
## using the asia mirror
human <- useEnsembl('ensembl',dataset = "hsapiens_gene_ensembl", mirror = "asia")
mouse <- useEnsembl('ensembl',dataset = "mmusculus_gene_ensembl", mirror = "asia")
#比如我有一系列小鼠基因:
mouse.genes <- c("Psme2b","Zfp661","Tsc22d1","Prox2","Eml3","Creb1")
#将其映射到人的基因上
MtoH <- getLDS(attributes = "mgi_symbol", # 要转换符号的属性,这里基因名(第3步是基因名)
filters = "mgi_symbol", #参数过滤
mart = mouse, #需要转换的基因名的种属来源,也就是第2步的mouse
values = mouse.genes, #要转换的基因集
attributesL = "hgnc_symbol", #要同源转换的目标属性,这里还是转为基因名,也可加其他
martL = human, #要同源转换的目标种属,也就是第2步的human
uniqueRows = TRUE)
###Error: biomaRt has encountered an unexpected server error.换成其它镜像也不行
###有人给出了权宜之计,用既往存档的数据,参考https://support.bioconductor.org/p/9144001/
##https://www.ensembl.org/info/website/archives/assembly.html
human <- useMart("ensembl", dataset = "hsapiens_gene_ensembl", host = "https://dec2021.archive.ensembl.org/")
mouse <- useMart("ensembl", dataset = "mmusculus_gene_ensembl", host = "https://dec2021.archive.ensembl.org/")
##换成2022的archive,又不行了
human <- useMart("ensembl", dataset = "hsapiens_gene_ensembl", host = "https://oct2022.archive.ensembl.org/")
mouse <- useMart("ensembl", dataset = "mmusculus_gene_ensembl", host = "https://oct2022.archive.ensembl.org/")
##烦躁,