半抑制浓度(或称半抑制率),即IC50,英文名称为half maximal inhibitory concentration。它能指示某一药物或者物质(抑制剂)在抑制某些生物程序(或者是包含在此程序中的某些物质,比如酶、细胞受体或是微生物)的半量。在凋亡方面,可以理解为一定浓度的某种药物诱导肿瘤细胞凋亡50%,该浓度称为50%抑制浓度,即凋亡细胞与全部细胞数之比等于50%时所对应的浓度,IC50值可以用来衡量药物诱导凋亡的能力,即诱导能力越强,该数值越低,当然也可以反向说明某种细胞对药物的耐受程度。
Graphpad操作演示:
1. 本案例以进哥最近实验数据为例,数据结构如下,两组,各三个平行:
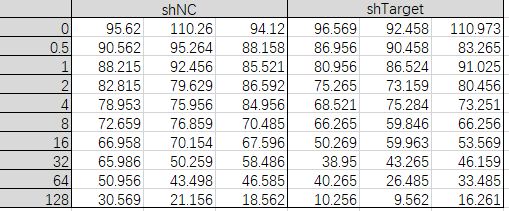
2. 打开Graphpad Prism软件,图表类型选择“XY”,在Data table选项中勾选“Enter or import data into a new table”;Options选项中勾选“Enter 6 replicate values in side-by-side subcolumns”,在本例中,药物浓度有6个复孔,因此输入数字“6”,如果我们已经计算了SD值,那么可以选择“Enter and plot error values already calculated elsewhere”选项。设置完成后,点击“Creat”创建图表。
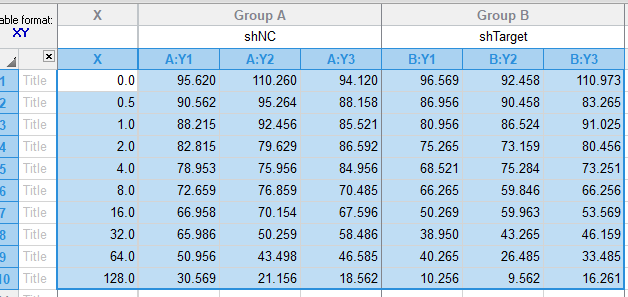
3. 复制粘贴数据
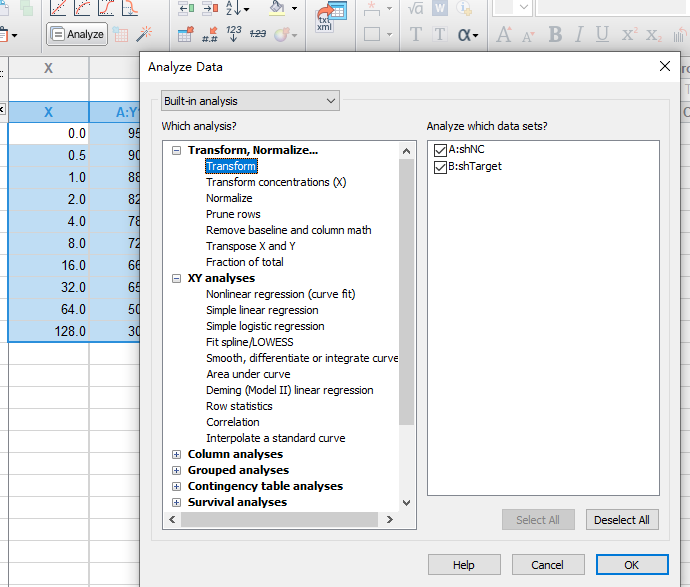
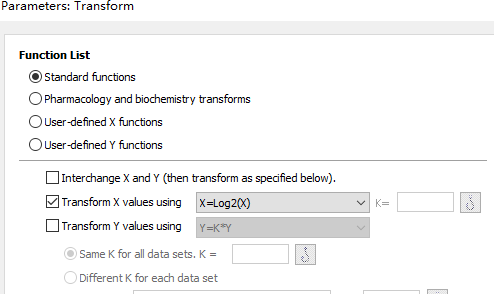
4. (可选,视情况而定)数据转换
大家可以看到,这是一个指数数浓度梯度,以2的倍数增加,对于这种类似的浓度梯度,如果不进行log对数转化,绘图时低浓度的点会挤在一起,结果不清晰也不美观。因此可以选择进行对数转化,此处log2转化:
,在上方工具栏点击“Analyze”,在新打开的Analyze Date对话框中,展开“Transform, Normalize”项目列表,选择“Transform”,点击“OK”打开Parameters: Transform对话框。
5. 在界面左侧的导航栏“Results”处,可以看到转换之后的数据,点击Graphs下面也可以看到默认的散点图。
6. 接下来我们进行曲线拟合,首先选中Data1,在工具栏中点击“Analyze”,在新打开的Analyze Date对话框中,展开“XY analyzes”项目列表,选择“Nonlinear regression (cure fit)”,点击“OK”。
在新打开的Parameters: Nonlinear Regression对话框中,选择“Model”选项卡,展开“Dose-Response-Inhibition”列表,选择“[inhibitor] vs. Normalized response–Variable slope”,点击“OK”即可执行计算。
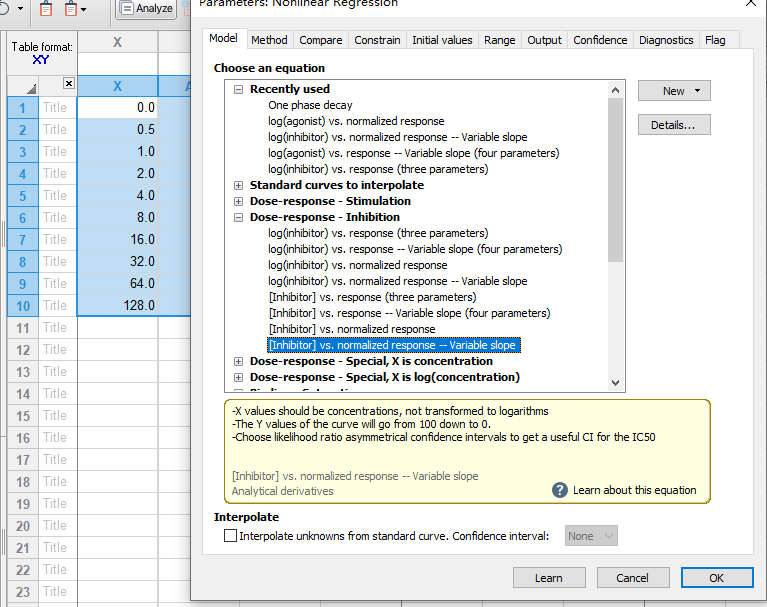
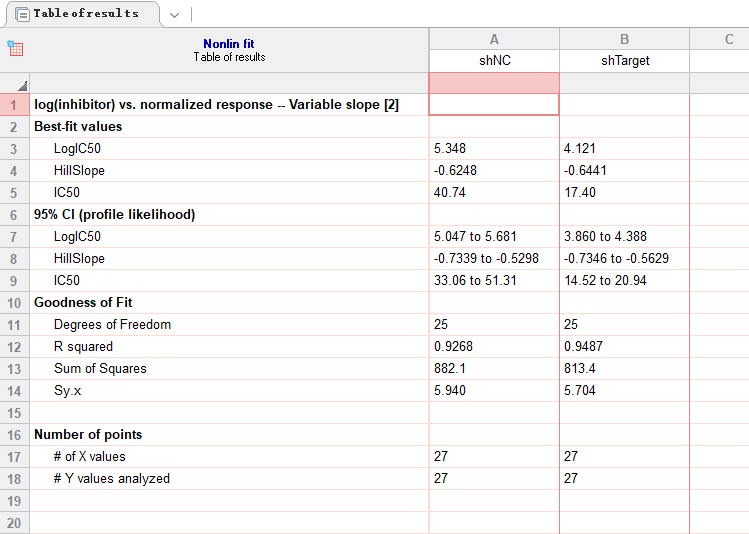
7. 计算完成后,在左侧导航列表中“Results”下的“Nonlin fit of Data 1”可看到计算结果。
shNC组IC50 = 40.74,shTarget组 IC50 = 17.40。
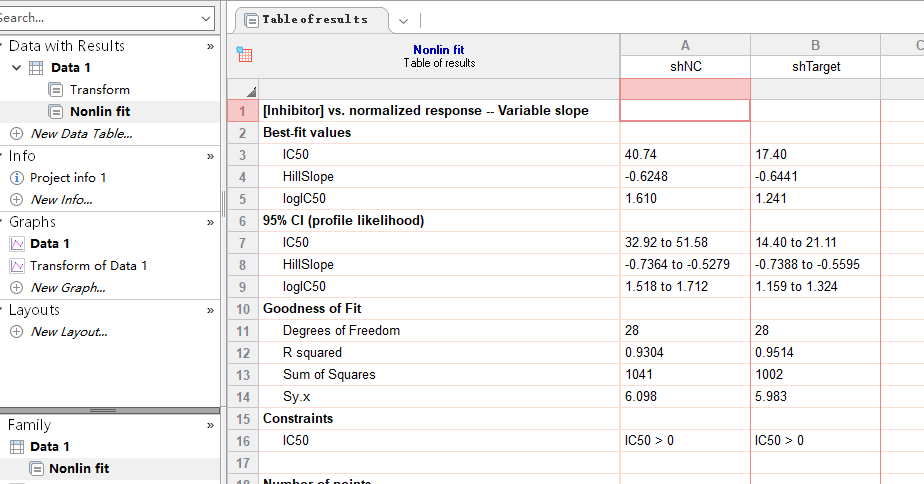
PS:如果使用对数转化之后的数据,选中transform数据,此时就选择“log(inhibitor) vs. normalized response — Variable slope”。需要注意的是,如果浓度是经过log10转化,那这里不需要改变公式,如果向这边教程经过log2转化,则需要修改公式:双击此时就选择“log(inhibitor) vs. normalized response — Variable slope”,弹出界面底下点击Clone this equation,弹出一个user-defined equation,将10改成2。
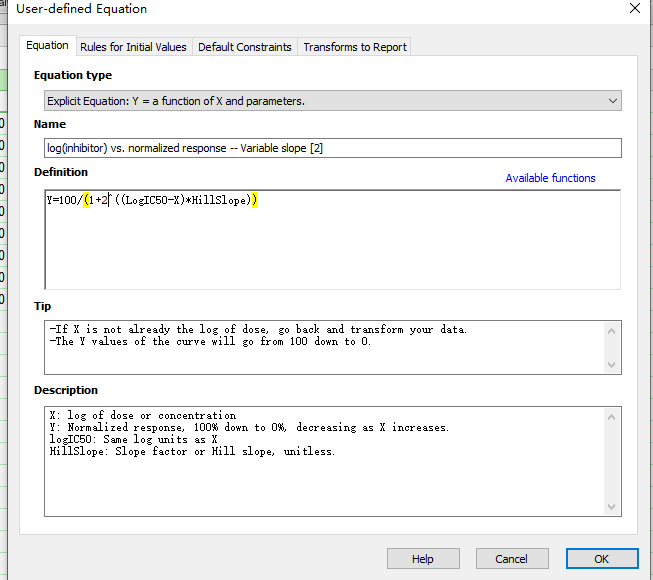
对应的,Transforms to report里面10变成2,点击OK:
可以看到,与前面未经过转化的结果一样。
8. 这时候再点击Graphs下面的Data1或Transform of data1,就可以得到拟合曲线:
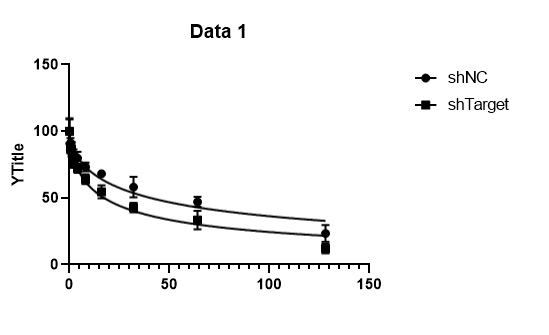
Graphpad图片美化就不多说了,大家用过的应该知道如何调整。
R语言操作演示:
可能用R来计算IC50和绘制剂量-效应曲线,大家会觉得大可不必。但是对于大量计算以及追求美观的朋友,用R处理不失为一个好方法。这边给出最基本的代码:
df <- read.csv(“IC50.csv”)
df.m1 <- drm(Response ~ Dose,Group, data = df,
fct = LL.5(fixed = c(NA,0,100,NA,1),names = c(“b”, “c”, “d”, “EC50”, “f”)))
par(lwd=2, cex=1, font.lab=2)
plot(df.m1, broken=F, bp=.1, bty=’o’,col = brewer.pal(9,”Set1″)[1:length(unique(df$group))],
lty=c(1,2),pch=c(15,17),legend = 0,
xlab=”Dose”, ylab=”Cell viability”)
##提取IC50数值
EC50.1 <- round(summary(df.m1)$coefficients[3, 1], digits = 3)
EC50.2 <- round(summary(df.m1)$coefficients[4, 1], digits = 3)
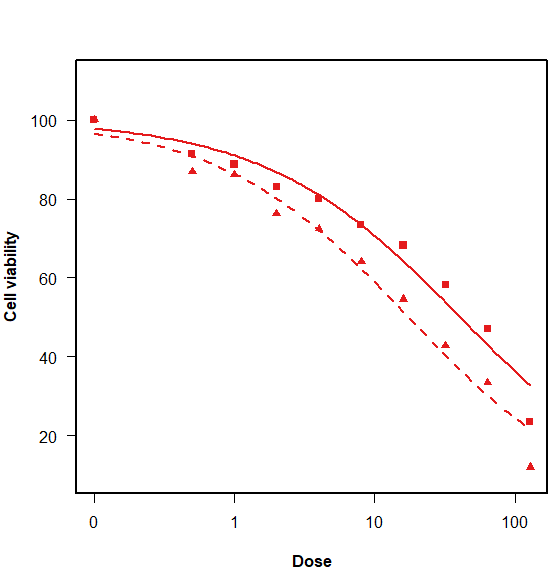
具体请看B站视频,
相关代码请至:
B站代码获取 – 王进的个人网站
请问在最后plot的时候报错“argument “name” is missing, with no default” 是什么原因呢?
小程序里面吗?得参考demo data的列名,需要一致,数据结构也应该一样