自从去年发了个TCGA数据下载合并的教程之后,不少同学加我微信或者网站留言遇到各种问题,最主要针对那些刚接触R语言的同学,我都会远程给他们解决,因为往往都是一些基础的问题。为此,考虑了一下还是做了这样一个工具,不需要太多R语言基础就可以运行,需要学习代码也可以看到,就设计了这样一个本地运行的Shiny app。相关使用教程演示已在B站发布,但是忘记教新手们怎么打开app了,这边上教程了(根据用户体验,已经更新了部分代码,与B站有一点点地方不一样):
- 以下网址获取代码:
2. 解压
用RStudio打开ui.R文件,一般情况下软件会自动检查相关R包有没有安装,如果没有会有提示,点击安装即可;
如果没有提示,可以运行一下ui.R前面几个library代码,确保所有需要的包都安装好。
3. 点击右上角Run app:
4. 复制你的文件夹路径,粘贴到下图文本框中,随后点击Refresh:
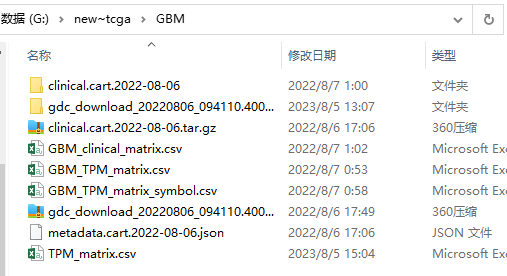
5. Merge Data界面会自动识别目录下的json文件以及解压后的数据文件夹,选择正确的文件夹,然后选择需要导出的数据类型(Count,TPM,FPKM):
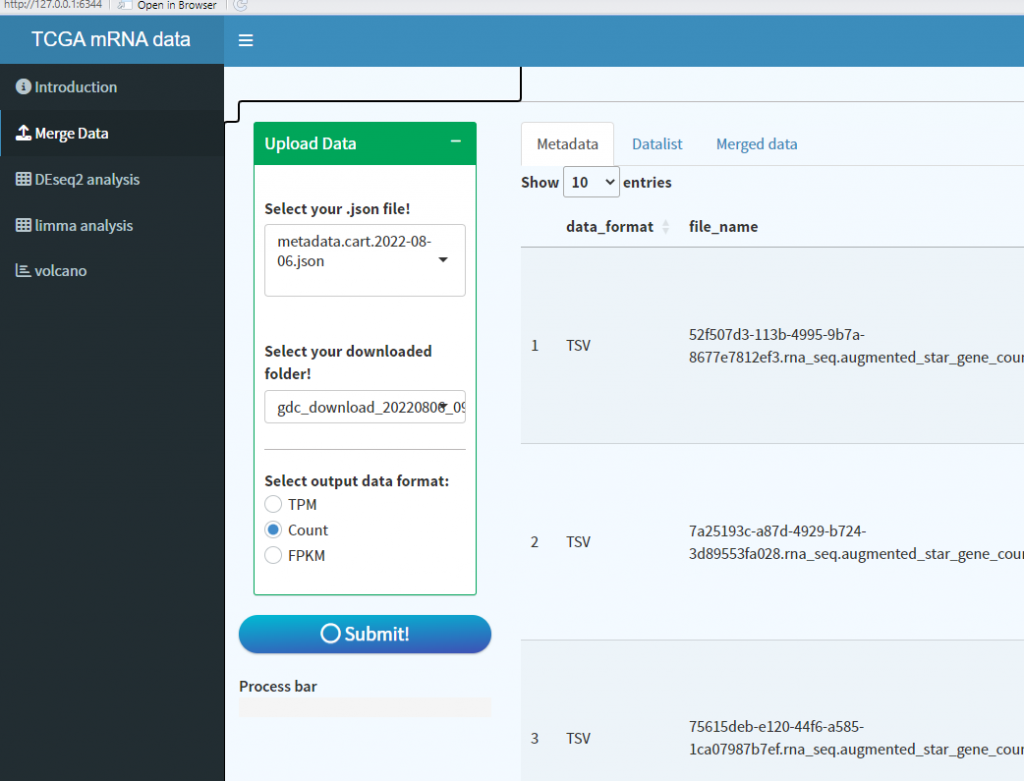
此时,Metadata会显示json文件中的样本及文件信息,Datalist会显示数据文件夹中的所有样本的tsv数据文件,两个表格数据应该是一样,不一样会报错。
6. 点击submit,自动跳转到Merged data,同时进度条开始滚动,耐心等待进度条完成。
完成之后右下角会有提示,已经完成合并,正在自动保存至目录下。此时你会在你的数据目录下看到整合好的csv数据,小程序表格中会显示前几个样本及基因作为演示。
7. 差异分析
切换到差异分析界面,对于count数据,使用Deseq2,刚刚第一部合并得到的数据应该此时在下拉列表看不到,你需要回到introduction界面refresh一下,重新获取目录下的文件。这时候你会发现DEseq2界面就会出现这个文件,一键submit即可进行差异分析。注意,DEseq2运行很缓慢,请耐心等待。。。你可以在R语言Console中看到分析的进度提示。结束之后点击Download即可下载完整数据。
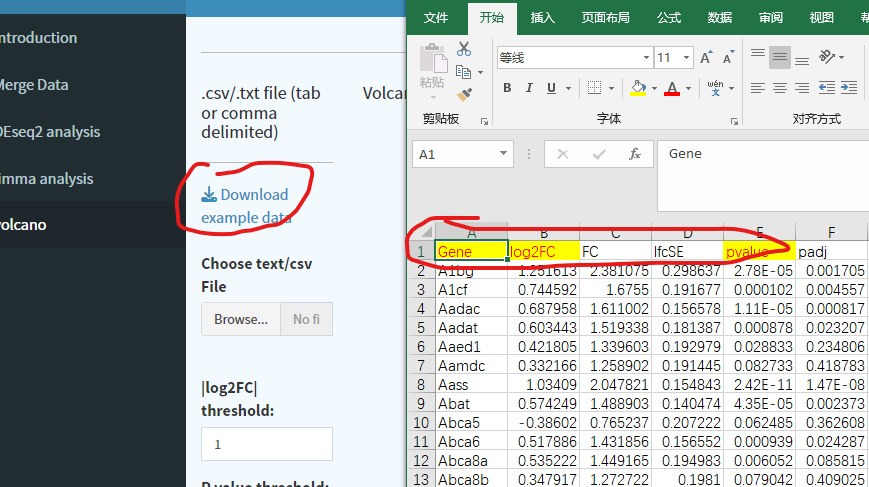
8.贴心的,为大家加上功能还算齐全的火山图绘制app
需要注意的是,这里是要上传差异数据,列名一定要按照example data设置,必须要改的列名是Gene、log2FC和pvalue,其他参数很简单
提交数据即可得到一个好看的火山图了
如果大家想要学习相关代码,包括合并数据、差异分析和火山图绘制,就是在小程序文件夹中server-那三个,特地分开,方便大家学习。
最后祝大家实验顺利!有问题留言!!!
小程序代码在这儿:
进哥,从你的微博里得到了好多知识,非常感谢你的付出。我刚接触生信分析,在研究中碰到一个问题,查了好多文献也没有得到确切答案,想请教你一下:我利用TCGA数据库中的肝癌样本和正常样本的count值做差异基因分析,在做数据清洗时我将count表达值<10和样本超过50%的缺失值的基因去掉,这样肝癌样本中的基因数从大约50000多变为了17000多,那么我后续要用log2(TPM+1)数据做生存分析,以及做免疫和药敏等等分析时,是不是都需要在这17000多基因池里面进行,而不是50000多基因池中进行,同时想问一下你在清洗数据的阈值是怎么定的,万分感谢!