转录因子(TF)是细胞中的蛋白质,它们可以特异性地结合到DNA上并调控基因的表达。转录因子靶基因预测是指通过生物信息学工具或实验方法,预测转录因子可能调控的基因群。这一过程对于理解基因表达的调控机制以及复杂生物学过程中的功能性网络至关重要。
目前,有多个数据库专注于转录因子及其靶基因的预测,例如TRANSFAC(转录因子的调控元件和靶基因数据库)、JASPAR(开放存取的转录因子结合位点模型数据库)、TRRUST(人类和小鼠转录因子及其靶基因的可信关系)、ENCODE(编码转录调控的项目数据库)等。这些数据库为科研人员提供了丰富的资源,以探索和验证转录因子与靶基因之间的相互作用,并揭示其在不同生理和病理状态下的功能。
之前进哥也整理了一些转录因子数据库:
“转录因子”的搜索结果 – 王进的个人网站
https://www.jingege.wang/?s=%E8%BD%AC%E5%BD%95%E5%9B%A0%E5%AD%90
也做了一个基于爬虫的shinyAPP用于预测调控目标基因的转录因子预测:
然后,有同学留言让加下转录因子靶基因预测功能,一直没时间搞,正好最近要用到,于是乎就做了一个,与之前的工具独立,数据库略有不同,但都是基于爬虫进行数据获取。入口如下:
APP操作非常简单,界面如下:
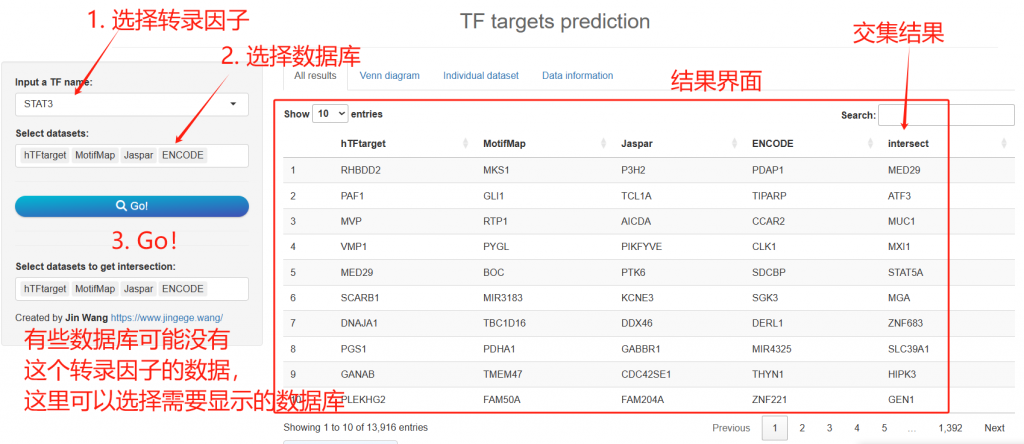
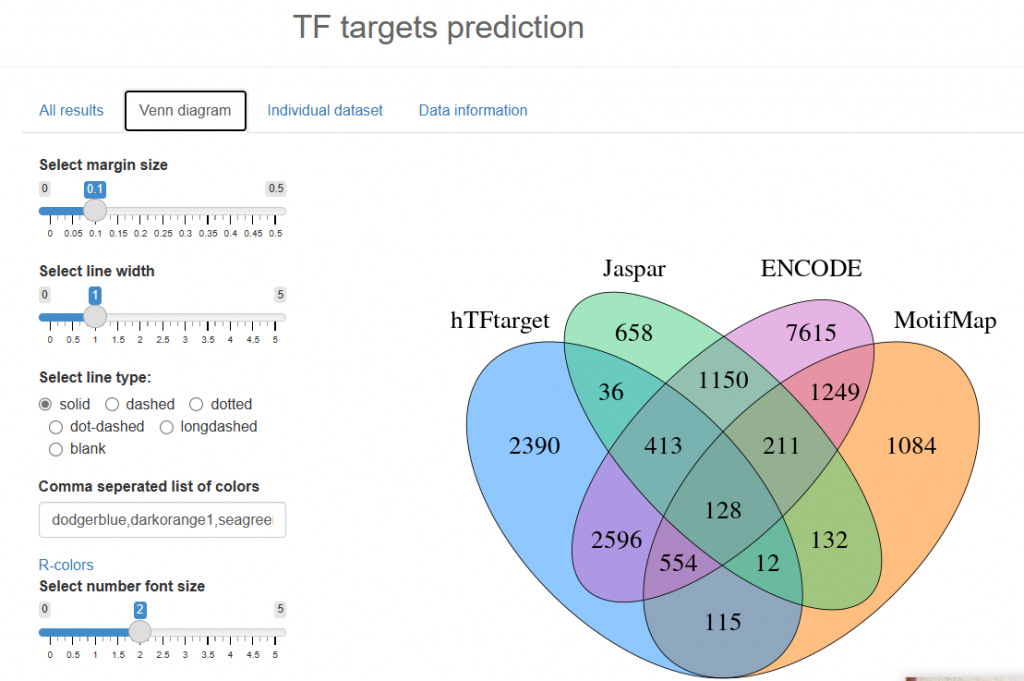
最后数据来源也给大家展示一下:
| Dataset | Source | |
|---|---|---|
| 1 | hTFtarget | http://bioinfo.life.hust.edu.cn/hTFtarget |
| 2 | KnockTF | https://bio.liclab.net/KnockTFv2/index.php |
| 3 | TTRUST | https://www.grnpedia.org/trrust |
| 4 | MotifMap | https://maayanlab.cloud/Harmonizome/dataset/MotifMap+Predicted+Transcription+Factor+Targets |
| 5 | Jaspar | https://maayanlab.cloud/Harmonizome/dataset/JASPAR+Predicted+Transcription+Factor+Targets |
| 6 | ENCODE | https://maayanlab.cloud/Harmonizome/dataset/ENCODE+Transcription+Factor+Targets |
重要说明:对于KnockTF,它是TF敲低或敲除细胞系的转录组数据集,可能有多个,这边爬取的是所有数据集的结果取并集,差异倍数取的是2倍,这是里面的筛选代码dplyr::filter(.,abs(log2fc)>1),如果需要看具体细胞类型的结果,可以从app下载所有数据下载后手动筛选出来。
最后,如果大家有什么建议请留言!我考虑有空的时候完善一下!
对啦,只有人的数据,其它种属不支持,毕竟相应数据库比较少,一个个分析也不难
老师你好,我用了您这个转录因子靶基因预测小程序,想预测EPAS1的靶基因但是一直显示无响应,请问可能是什么原因呢
这个为什么我一直打不开
老师您好,为啥搜索的时候一直显示disconnected,这个网站还能运行吗
hi,进哥,您有空的时候看下,这个网页现在老是提示
Disconnected from the server.
Reload
不晓得啥原因,是api被禁用了还是啥?
感谢 感谢
能不能出个反向应用的小程序,多数据库通过靶基因预测调控靶基因的转录因子