众所周知,一个转录因子能同时控制多个基因的表达,同时转录因子的功能作用也可能受多个调控因子的影响。转录因子对基因表达的调控至关重要,如何去科学的预测靶基因的转录因子,这是我们在做转录因子研究的时候需要考虑的问题。这里给大家罗列了几个常用的转录因子预测在线工具:
Datasets | Source | Evidence |
motifmap | http://motifmap.ics.uci.edu/ | motifs |
hTFtarget | http://bioinfo.life.hust.edu.cn/hTFtarget#!/ | ChIP-Seq data |
KnockTF | https://bio.liclab.net/KnockTF/index.php | Knockdown/knockout |
TTRUST | https://www.grnpedia.org/trrust/ | Pubmed |
Cistrome | http://cistrome.org/db/#/ | ChIP-Seq and DNase-Seq |
ENCODE | https://maayanlab.cloud/Harmonizome/dataset/ENCODE+Transcription+Factor+Targets | ChIP-Seq data |
Jaspar | https://maayanlab.cloud/Harmonizome/dataset/JASPAR+Predicted+Transcription+Factor+Targets | motifs |
- 关于转录因子调控基序(motif)就不多说了,学过分子生物学的同学应该知道,motifmap和Jaspar主要基于motif进行转录因子预测;
- 因为转录因子是和DNA结合的,因此通过ChIP实验可以拉下与转录因子结合的DNA片段,结合高通量测序可以得到所有的靶基因,hTFtarget、Cistrome和ENCODE在线工具主要基于该技术;
- 另外转录因子会调控基因表达,因此敲低转录因子之后相关靶基因表达会发生改变,KnockTF就是整理了众多转录因子敲低的GEO数据集;
- 与前一点相似,在没有调低转录因子的样本中,转录因子应该也与靶基因表达之间存在相关性。因此可以基于大样本的高通量数据进行表达相关性分析。
关于这些转录因子数据库的使用介绍,大家自行查找或看一下我的B站视频介绍,当然我网站上面也有一些数据库介绍。
https://www.jingege.wang/?s=%E8%BD%AC%E5%BD%95%E5%9B%A0%E5%AD%90
现在进入正题,也就是这个转录因子预测小工具的使用及数据来源,使大家使用的时候明明白白。
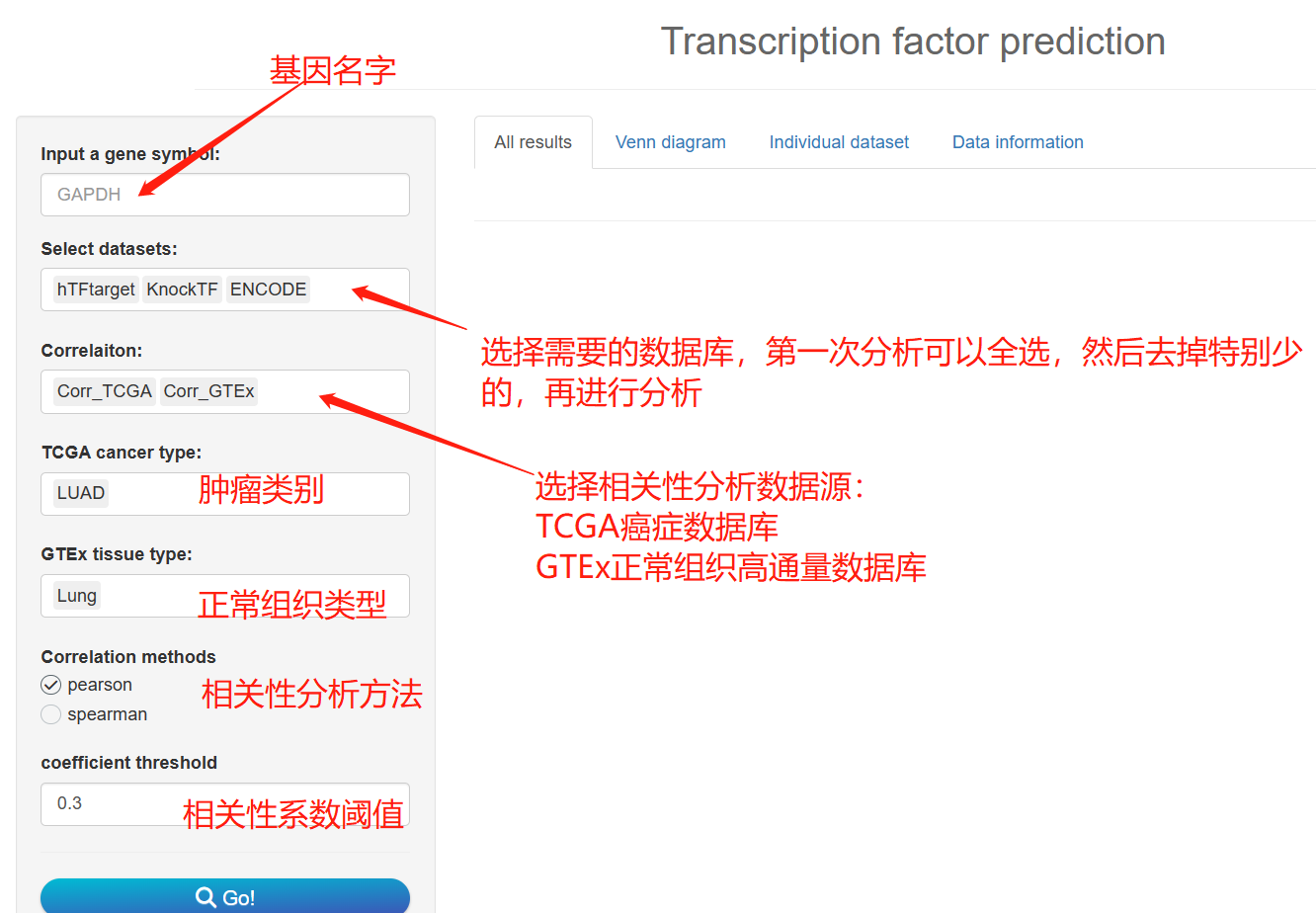
- 首先打开之后是这个样子,非常之简单,相关设置如下图:
2. 等待几十秒之后可以得到结果:
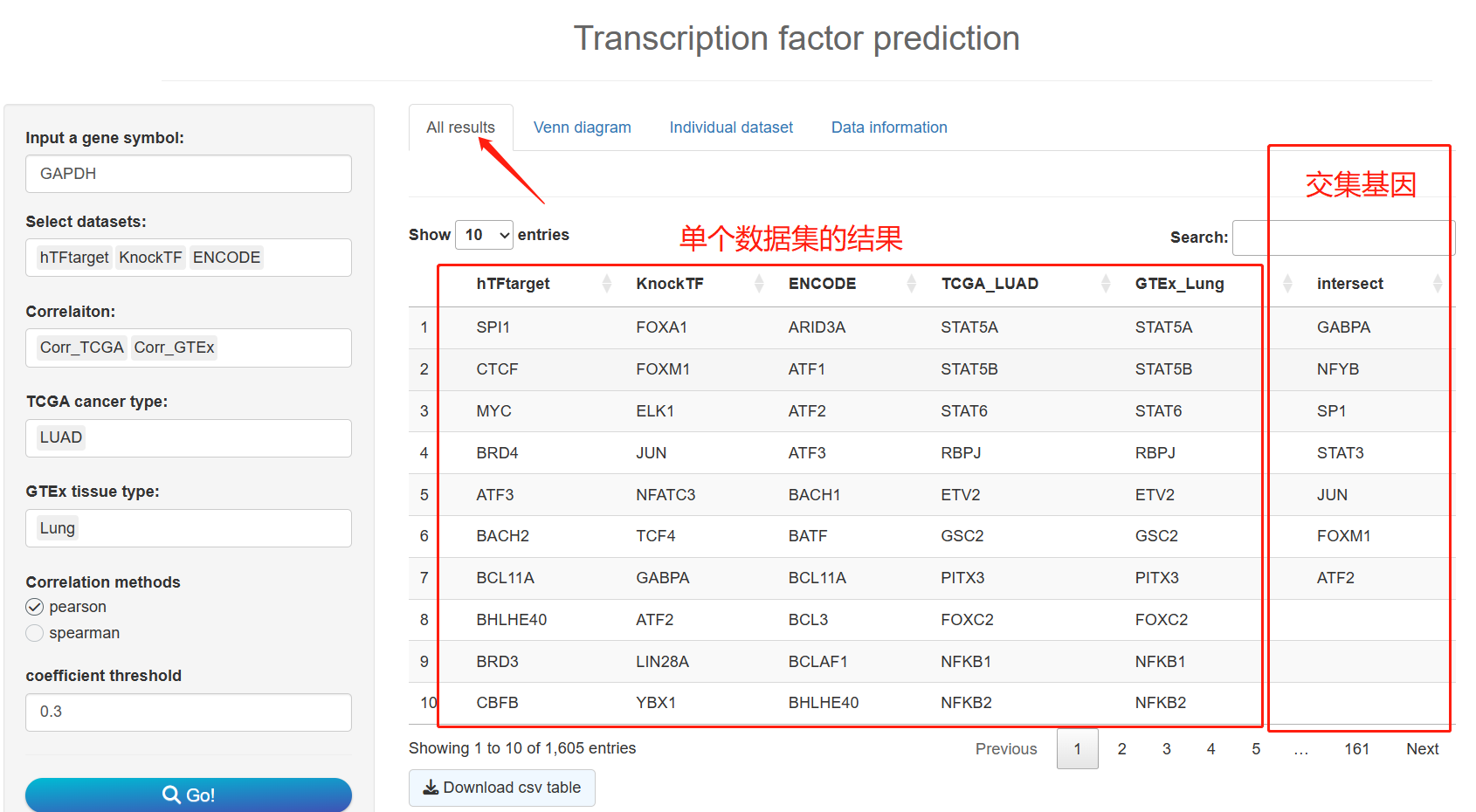
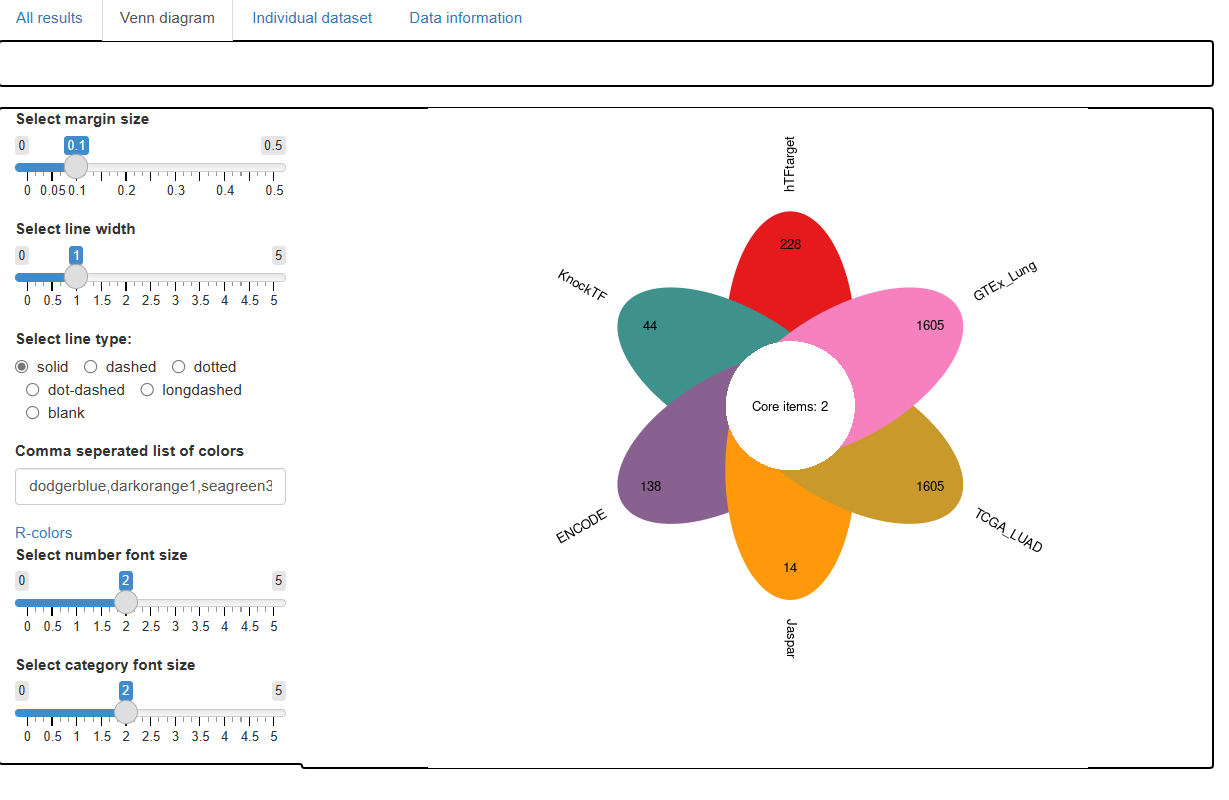
3. 贴心的给出可视化结果,小于等于5个数据集使用Venn图可视化,更多的话以花瓣图进行可视化,Venn图相关参数自行摸索,基于VennDiagram包:

这是6个数据集交集的flower plot,我没有给出图形参数设置,偷个懒,默认出图还是不错的:
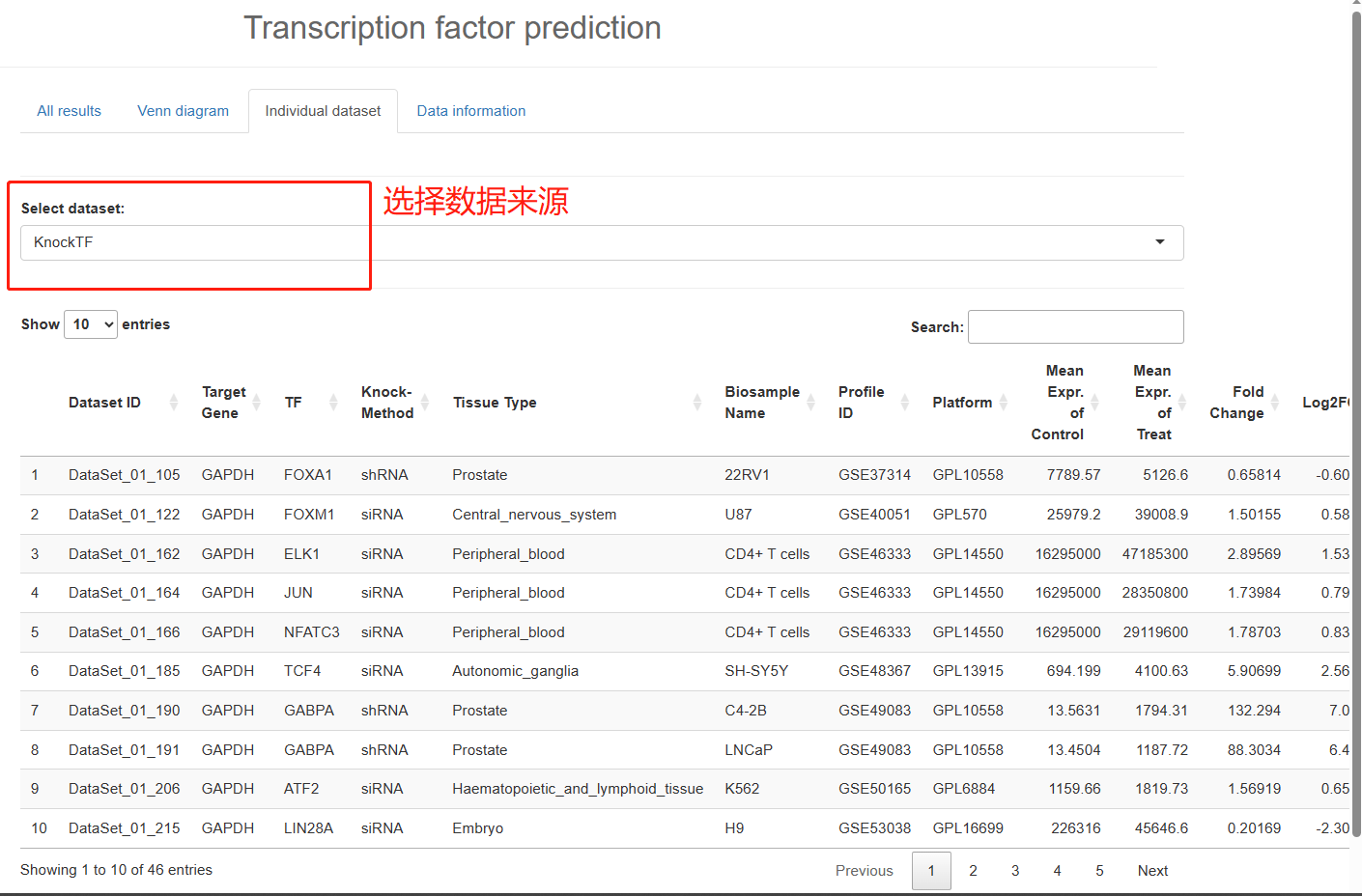
4. 其实针对单个在线工具,还会有很多其他参数,所以为了方便大家进一步筛选,app也提供了单个数据集的下载:
最后说一下这个app的原理:
- 对于hTFtarget、KnockTF、TTRUST和Cistrome,基于网页爬虫,所以结果是和网页搜索应该一致,对于Cistrome DB,爬虫速度相对较慢,尤其是存在多个转录本的时候。
- 对于ENCODE和Jaspar,是基于Harmonizonme数据库整理好的数据集,然后上传到我的云服务器。
- 对于相关性分析,基于Xena browser上面提供的TCGA/GTEx数据集,提取转录因子数据之后上传到我的云服务器,再基于相关性分析计算靶基因与转录因子表达的相关性。
That’s all!
Enjoy!!!
有建议请留言!
进哥,一直显示Disconnected from the server.
Reload
不能用了
请问下,为啥用不了,一点“Go”,一直是”Disconnected from the server“这个状态,点了“reload”还是一样。
老师,请问现在我输入一个基因点击GO后,新的页面左下角一直显示与服务器连接不上,这个是什么原因呢
进哥好,非常感谢您的分享,网站可以进去但搜索报错,有什么办法么?
哥哥 这现在始终登陆不进去啊。。。 是服务器的原因么 总显示失去连接
老师,请问靶基因转录因子预测工具 TCGA cancer type这里是只能填一个吗?比如:COAD READ一起填,提交后就显示 Disconnected from the server
老师好,靶基因转录因子预测小工具提交后显示:Disconnected from the server.出不来结果呢
这个为什么一直显示disconnect from server呀
GTEX 相关性会报错
为什么显示一直与服务器断联呢
网站是不是在维护呢,进入APP会输入目标基因进行选择后发现连接不上
进哥,网站最近打不开,能不能修复一下
进哥,KnockTF这个库预测不出来,是不是有bug呀
进哥,您好,我用您设计的小程序预测某个转录因子靶基因,没有得到结果,但是可以在相关文献上查到相应的靶基因。想问一下是否是种属的原因?这种情况该怎么办呢?
不知道默认基因启动子序列是多长,好像各个预测也不太一样,可靠性上打折扣了,能否完善序列输入模块了。
进哥,想请教这个分析的结果默认的是人的吧,有没有考虑将小鼠TF板块纳入呢
可以把代码放出来不,想放自己机器上跑,然后出图:)
进哥,给个citation呗
请教进哥,这个小工具预测的是输入基因的上游还是下游的转录因子呢
进哥,请问为什么我从Jasper直接预测的和在您的app里预测得到的不一样呢,是基因序列选取的版本不同嘛,还是怎么回事,麻烦进哥解答一下
感谢进哥,我想问一下如果我想预测一个酶的活性位点该如何操作?
为什么GTEx中与任一数据库预测的转录因子都没有交集呢
进哥,请问一下转录因子的靶基因也可以用这个预测吗?
目前不可以 改天更新一下 加一个模块
感谢。十分期待能够预测转录因子靶基因。
同求,感谢进哥
请问目前可以了吗
进哥你好,感谢您的分享,我想问一下取交集的TCGA数据和GTEx数据是指TCGA数据和GTEx数据中的所有基因么,还是指其中与靶基因正相关的基因呢,感谢!!
相关性系数绝对值大于设置阈值的转录因子,不是所有的
感谢进哥,前几天使用网站一直没问题,但是今天网站进不去了,是否网站出现什么问题呢?
可能大概并行用户太多 卡住了,公共服务器,难堪重负
感谢进哥的教程,但是没有找打小程序的位置?
页面底下视频上面有个按钮,蓝紫色的那个 很大的按钮