https://www.graphpad.com/guides/prism/latest/statistics/stat_anova-approach-vs_-mixed-model.htmThe problem: Repeated measures ANOVA cannot handle missing values
这是官网对于缺失值的说明,其实没有什么办法
Repeated measures ANOVA calculations require complete data. If a value is missing for one partiicpant or animal, you’d need to ignore all data for that participant or animal. The only way to overcome this (using ANOVA) would be to impute what the values of the missing values probably were and then analyze without any missing values, correcting the results (reducing df) to account for the imputing. This is not a preferred method, and is not offered by Prism.
进哥你好,我想问下在做基因敲除的时候,通过结合转移将目标质粒转入转入目标菌株,长出接合子后进行摇瓶培养(含相应的抗性),此时是得到单交换菌株,我不理解的是此时的单交换菌株中是否目标质粒已经敲进目标菌株的基因了(或者说此时菌株中的目标基因已经敲除了)。
您好,前两天比较忙 没时间看留言。
第一次同源重组得到的单交换菌株中含有目标质粒,但是目标基因还在里面 还需要进行第二次交换才能把目的基因敲掉,具体看下文原理图
同源重组敲除基因原理及方法
您好,偶然在网页刷到,想请问您知不知道关于针对革兰氏阳性菌设计Cas13a的gRNA设计方法,最近找了很多文献都是做病毒,看到有做细菌的提到的网站CRISPR-RT,我们课题组的小伙伴也没法打开,甚至给那些做出来的发表文章的师兄师姐发了邮件也是石沉大海。所以看看您有没有接触这方面的知识,可以传授一下吗?谢谢指导。
您好,如果没有现成工具 不放通过序列设计 一个是手动设计 一个是有根据序列设计sgRNA的工具
可以加我微信讨论
大神你好,想问下研究关于lncRNA和miRNA互作的DIANA TOOLs lncBase Predicted v.2 这个你用过吗? lncBase是否不开放了?为什么进不去了呢?(PS: 您的网站和人一样都太优秀了)
你好,V2的链接确实进不去了,现在有LncBase V3, 好像是没有predict模式了
嗯嗯,谢谢大神~
大佬,能发一下mirbase里gma-miR166v和w的MIR序列和成熟体1序列吗?PmiREN没有他俩的链接,我自己搜到的一直处于搜索中。
您好 我没有查到miR166w/v的序列诶 貌似miRBase也没有 你是哪儿看到的
感谢大神的分享,学习到很多!!!
大神不敢当,一起学习一起积累
老哥,网站做的真棒!!!!
谢谢๑•́₃•̀๑,有机会的话可以一起学习
学到很多!感谢您的付出!
不客气,有需要一起讨论
您好,大神,我是在别人转载的文章里面看到你的对CRISPR技术的理解,我觉得很有趣,也很容易懂,您是也有涉及到基因编辑相关的工作吗,是否可以有其他交流请教的方式?
您好,我的简历里面有其他联系方式,需要的话可以加我微信
进哥哥,这个网站的的主题可以分享吗?
你好 网站底下有 :Blog Diary by Theme Palace
师兄您好,请问您有RNA分析工具——RAIN的网站吗?请问RAIN的使用方法是怎么样的呀?
没有接触过 我学习一下发你
您好,我看了您分享的Prism重复测量方差分析教程,觉得很棒。有一个小问题想请教您,我的数据里不同分组的重复数不一样,按照您的教程走会显示有缺失值导致出不来结果,请问有什么解决方案吗?
https://www.graphpad.com/guides/prism/latest/statistics/stat_anova-approach-vs_-mixed-model.htmThe problem: Repeated measures ANOVA cannot handle missing values
这是官网对于缺失值的说明,其实没有什么办法
Repeated measures ANOVA calculations require complete data. If a value is missing for one partiicpant or animal, you’d need to ignore all data for that participant or animal. The only way to overcome this (using ANOVA) would be to impute what the values of the missing values probably were and then analyze without any missing values, correcting the results (reducing df) to account for the imputing. This is not a preferred method, and is not offered by Prism.
感谢您的回复!
被大佬浪漫到!
您好,近期我在学习shiny,可以分享一下您shiny网站的热图的实现代码吗?
当然可以 要不你加我微信?
怎么加微信呢
您好 我的简历 页面有手机和微信二维码 手机号18021308280
师兄,你好,我想问一下 肺癌细胞系做转染,有没有比较好的细胞系推荐呢?转染效率高一点的
你好,这个我就不知道了,转染试剂和不同转染试剂和用量都有关系
大佬,看了您关于endnote文献信息自动更新的文章,我想问 如果我用的是自己的网络,无法在线链接web of science这个样数据库,是不是就不能更新文章信息了?
嗯嗯,不能在线更新文献的信息,连不上可以手动下载更新
来感谢大神的软件嘿嘿嘿
不客气,嘿嘿
老师好,您的image J划痕插件非常好用,谢谢!最近在用image J处理划痕结果的时候遇到一个问题:显微镜拍照软件上我没有选对物镜倍数,导致我有一组图片的分辨率是4800*3600 pixels,其他是1600*1200 pixels,image J计算划痕面积好像就是根据像素来算的,导致我这一组数据错误。请问还有什么补救的方法吗?我可以粗暴的把这组数据除以9,得到的是正确结果吗?希望得到您的解惑,谢谢!
如果有标尺,可以将图上距离变成实际长度(nm/um)
没有的话很简单,将原图右击编辑,用自带画图软件调一下大小,–>左上角重新调整大小–>像素–> 调成一致的 当然ImageJ PS都可以实现
再然后 /9当然也可以。。。
背景这个几何线条的审美实在是太酷了,不知道是怎么建设的呢
哈哈 就是网页加了个背景插件Canvas-nest.js,算是一个解压小游戏
谢谢分享
我也觉着这个很漂亮,第一眼就被吸引了。给博主赞赞赞
哈哈?
您好,想请教您一下,流式的单参数直方图中,阳性细胞群出现两个峰,阴性的那个峰是否可能是死细胞引起的呢?
您好,不清楚您的具体实验方案,出现三峰根据我的经验的话,有多种可能,一个如你所说可能死细胞,解决方法加死活染料去死细胞,还有抗体量不足或者细胞过多,导致部分细胞染色不充分,会多出一个弱阳性峰;再有也可能有黏连细胞,出现比正常阳性还高的强阳性;当然不能排除可能存在的强阳性和弱阳性细胞;
具体什么原因要根据你的实验方案及检测指标分析,方便的话继续留言讨论
老师好,最近老板让我找乳腺癌脑转移动物模型的成瘤率、最佳注射时间这些细节,看看实验可行性怎么样,我应该去哪里找相关资料呢,?找了好多文献并没有讨论到具体的问题~~~~·
您好 如果文献中提的不具体,您可以去一些实验动物相关的公众号,会有相关视频,还有一个实验方法网站 比如bioprotocol和JOVE,里面有很多实验视频,你可以找找
太强了,进哥。南方医免疫学ph. D student
幸会 有机会多交流
师兄您好,想请问您一下是否有什么数据库可以检索某一个蛋白是受哪些信号通路调控或者说预测可能受哪些信号通路调控的?
你好,如果只有一个蛋白名称,那可以考虑用String数据库,他可以分析预测与之相互作用和调控的蛋白,进一步富集分析可以得到可能调控的蛋白。如果有某种疾病的表达数据集,比如TCGA上的某种癌症数据,可以采用单基因GSEA富集分析预测期功能和相关信号通路
感谢师兄回答,我去试试先
进哥网页太棒了,向你学习!请教一下进哥,您建过小鼠结肠癌原位瘤模型吗?我感觉直接手术开腹腔把细胞打到盲肠,成瘤率有点低。也有人是直接从屁屁的粘膜里用灌胃针注射,这个没做过我心里没数 🙁
首先谢谢认可,结肠癌我没有做过,第一点你说开腹将细胞打到盲肠,这个意思应该是打到盲肠黏膜下吧。这个应该就和裸鼠皮下成瘤差不多吧。
灌胃针注射这个我觉得不太好控制
如果未来增大成瘤率,建议采用组织移植,而不是细胞悬液。具体操作就是先在裸鼠皮下注射结肠癌细胞形成瘤体,在将对数生长期瘤体剪成两三毫米大小的组织块接种至粘膜下,这样能保证成瘤以及瘤体大小
我没做过结肠癌,纸上谈兵理论上的建议,仅做参考
谢谢进哥哥!
您好大佬,想学习个人建站,主要用啥技术嘞。想学习建站,求指教
您好,首先需要空间或服务器,空间就是只能放网站,服务器相当于一台主机,需要自己安装系统和各种软件,我用的阿里云的云服务器
然后安装必要的建站所需程序,阿里云可以一键安装
接下来购买域名,也可以从阿里云买–>域名解析。。。
再然后选择建站程序,我用的WordPress
最后内容,排版。。。
有需要加微信讨论
请大佬多多更新,太强啦。已经分享给好友啦。
内参筛选我看进哥分享了Norfinder,不知道有没有用过Genorm,我最近用这两个软件进行内参筛选得到的结果不一样,我只想用单内参进行实验,是不是意味着我只用Norfinder就好,感恩
您好,使用NormqPCR包来选择多内参
这篇文章里面两种算法都有,算出来一只最好,不一致的情况选一种就好了,不用纠结太多
?太齐全了,感恩大佬
TCGA和GETX的数据库应该怎么分析呢,看不懂那个代码,可以请教么
加我微信吧,我的简历里面有,明天具体讨论
进哥,非常棒的网址!请问这个网址是通过什么平台建设起来的呢?谢谢进哥
谢谢,服务器是阿里云的,系统是Debian,网站程序是WordPress
搭建不难,关键是坚持,加油
您好,我想问一下,计算TIDE score时是用TCGA的FPKM、Counts数据中的哪一个呢?
您好,建议用标准化之后的数据,比如TPM和FPKM,建议TPM,现在FPKM大多已被弃用。TPM表达量在进行TIDE的标准化,按照网站说明
兄弟,可以合作兼职吗,我这边中美生物医药博士,目前在企业上班,行业就业相关的,如课题设计,文章修改,投稿,就业简历等,业余时间做,目前做的还可以,主要是和你一样服务大众,我们可以互补优势,我个人水平是没问题的,你有平台等,顺便赚个小钱,上海压力太大了,赚钱买房找对象,哈哈哈。微信:bioweiyong
当然可以,我加您微信
进哥哥,请问TCGA数据库更新后是不是之前您做的差异分析的代码也用不了了呀
能加您的vx吗
当然可以
差异分析的代码是基于Xena整理的TCGA数据,如果你用新版下载数据,只需要修改前面的数据下载代码 差异分析是一样
你好,我在叠加荧光图片和明场图片时一直提示The source stacks must have the same number of images是怎么回事,该怎样解决。感谢答复
看你的提示,应该是 图片的问题,是不是荧光那个图片已经是merge过的。如果解决不了可以加微信发图片给我看看
意外发现这个网站,真是意外之喜,这是一个有温度有爱的加油站,进来就不想离开
谢谢好评,继续努力,有机会可以一起学习
好佩服你呀!已收藏
谢谢啦,一起努力,一起学习
进哥哥,求一个有各个细胞系miRNA-seq表达量的线上数据库,谢谢
https://sites.broadinstitute.org/ccle/datasets
进哥哥,请问有没有网上的数据库可以显示不同细胞系中各个miRNA的表达量的呀
据我所知 CCLE上面有MiRNA的数据的,你看一下,看看能否在线分析 或者自己下载数据进行分析
进哥,您好,看了您的讲解真的感觉很透彻。还想咨询一下是否有存储比对之后的sam或bam文件的数据库,即mapping这一步不需要自己做
您好,这个我不清楚,不过推测应该没有吧,没必要整个中间文件的数据库吧,如果您找到了也可以分享一下?
不错,有机会合作
谢谢,没有问题,有机会合作
偶然在搜colorspace时路过学长你的网站,做得很棒,学术生活紧紧有条,我要向您看齐!
谢谢,有机会一起学习
查询实验步骤的契机点进了您的个人网站,网站很漂亮很专业很佩服你!加油!
谢谢美誉,继续努力,一起进步
因为查询一个问题看到你的个人网站,一开始以为是哪个大牛的网站,结果竟然只是一名在读博士。实在是太牛了,赶紧收藏下来!!!
哈哈,谢谢?,有问题可以一起讨论
我今年已经毕业,目前在苏大做博后
您好,学习了您的《R语言ggplot2优雅的创建字母显著性标记图》,对我新手R学习帮助非常大,但现在遇到一个新的问题,尝试了很多次都失败了,想请教您一下。做字母显著性分析,当双因素(品种,处理)存在时,如何在同一个坐标轴内,不做组间的比较,只做不同处理组的内单因素方差分析?感谢博主师兄!
您好,您的意思是绘制分组柱状图?统计是不同品种内的根据不同处理的ANOVO分析,然后添加字母标记?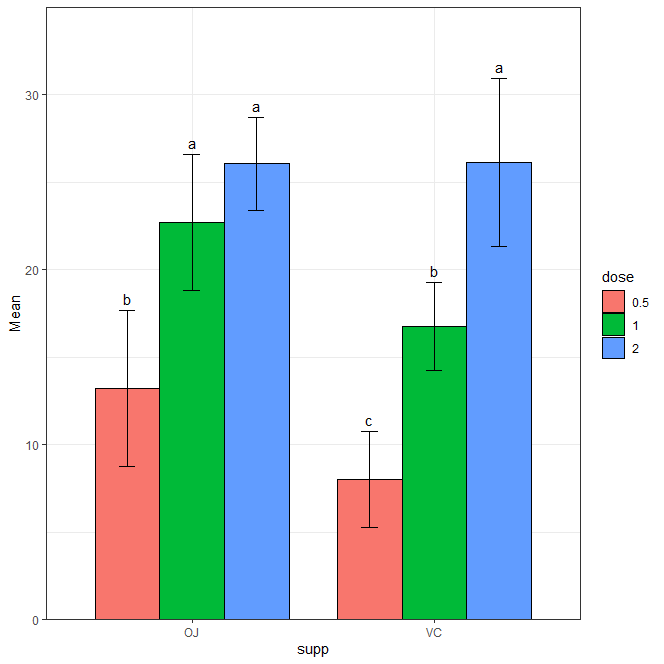
如果是这样,当然OK,ANOVO统计时将不同品种拆开分别统计,再合并
比如同样用toothgrowth数据画的如下图:
再有问题可以添加微信
十分感谢进哥不吝赐教,成功搞定了,抱拳抱拳!
我佩服的人不多,进哥把个人网站维护得这么好、无私分享知识,我深感佩服
谢谢啦,荣幸之至,有幸的话可以交个朋友
进哥,今天偶然接触到你的这个网站,幸甚。把学习和生活融合,把对知识的探求和对爱情的向往贯彻一生,我觉得这是一个科研人员的最高浪漫。
谢谢你了,我也很荣幸得到你的认可
你好,请问一下入门生信需要学的东西包括那些?目前在学R语言,但是没有合适的方法入门,有什么推荐的课程和书籍吗?谢谢。
你好,其实我没有什么特别好的方法,我认为没有必要专门进行系统学习,因为发展很快。最好的就是积累,要用的时候就去学习,文献看到一个图或一种分析,查一下怎么实现的,就这样一点点积累就好,我就是慢慢积累的。现在这些方法资料网上都能查到,需要培养的能力更主要是如何查资料
进哥,非常感谢您的回复,我想使用TIDE下载肝癌的TCIA资料,如果您肯把账号借我用一会,那真的是十分感谢,谢谢进哥
methprimer的网站出了问题,作者知道新的网址吗
您好,确实,应该是网站被黑了,过两天看看维护团队能不能解决,实在不行就手动设计
你好 methprimer可以用了,之前应该是被黑了
MethPrimer Home
http://www.urogene.org/methprimer2/
我试了这个方法,但是批量处理魔棒部分会报错,您分享的代码我运行出错。
您好,冒昧打扰您,我看到您关于细胞划痕实验的处理教程,想请教您这种方法可以做批量分析吗,最后计算的时候是直接取面积,不再除以高度吗?
你好,image J的批量可以通过宏命令脚本实现,可以参考这篇文章的方法Image J批量添加添加
关于计算结果就是面积,单位默认是pixel,可以根据标尺换算